 Укоренение в среднюю точку.
Укоренение в среднюю точку. Укоренение в среднюю точку.
Укоренение в среднюю точку.Для следующих бактерий:
Название |
Мнемоника |
Rhizobium etli |
RHIEC |
Burkholderia cenocepacia |
BURCA |
Ralstonia pickettii |
RALPJ |
Pseudomonas aeruginosa |
PSEAE |
Escherichia coli |
ECOLI |
Yersinia pseudotuberculosis |
YERPS |
Haemophilus influenzae |
HAEIN |
Pasteurella multocida |
PASMU |
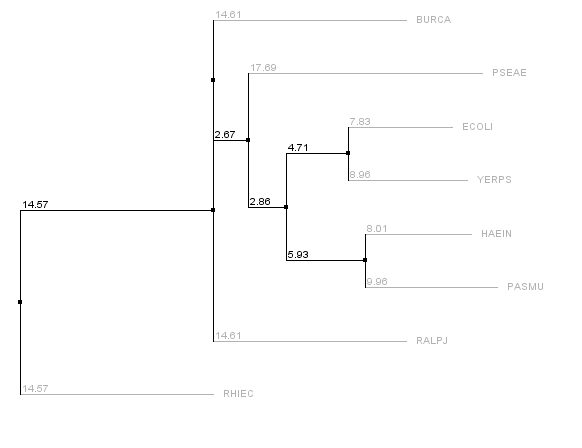
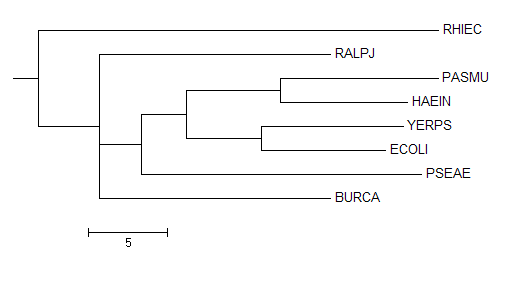
Были получены последовательности белков Пептидил-тРНК гидролаз, далее было построено выравнивание при помощи программы JalView. В этой же программе было реконструировано филогенетическое дерево по этим белкам методом "Neighbor Joining Using % Identity". Полученное дерево представлено ниже.
Данный методод не строит укоренённые деревья, что и видно из рисунка. В отличие от правильного дерева (в котором листья BURCA и RALPJ образуют отдельную кладу), построенного в предыдущем задании, здесь эти ветви имеют другую топологию, по этой же причине отличаются нетривиальные ветви полученного дерева.
При помощи программы retree пакета PHYLIP было выполнено укоренение в среднюю точку полученного дерева (средняя точка - расстояние между двумя листьями, расположенными наиболее далеко друг от друга).
Полученное укоренение совпадает с изначальным укоренением в правильном дереве.