Пакет Pftools
- Входной файл в формате msf
Программой noreturn получен файл al12.noreturn
- Расчет весов строк выравнивания программой pfw
Запуск программы: pfw al.noreturn > al_2.msf
В файле al.msf последовательности имели веса, равные 1, в новом файле al_2.msf (выдача программы ниже) последовательности имеют разные веса, меньше 1
Name: RS12_CLOTE Len: 15 Check: 9161 Weight: 0.2432 Name: RS12_STRPN Len: 15 Check: 9216 Weight: 0.0986 Name: RS12_BACSU Len: 15 Check: 9269 Weight: 0.1512 Name: RS12_LISMO Len: 15 Check: 9296 Weight: 0.1661 Name: RS12_LACDA Len: 15 Check: 9292 Weight: 0.1416 Name: RS12_PEDPA Len: 15 Check: 9137 Weight: 0.2094
- Создание профиля программой pfmake
Запись программы pfmake al_2.msf /usr/share/pftools23/blosum62.cmp > al.prf
- Подготовка файла с последовательностями в fasta-формате, в которых будет производится поиск
Запись программы seqret sw-org:bacteria bacteria.fasta
- Поиск по профилю
Запуск поиска pfsearch -C2.0 -f al.prf bacteria.fasta > al.pfsearch. Порог 2.0, т.к. при пороге 1.5 нашлись 15160 последовательности
Всего 1840 находок - Анализ списка найденных белков, сравнение его со списком всех белков подсемейства
- Число верных находок ("True positive hits", TP) = 153
Число ложных находок ("False positive hits", FP) = 1 687
Число ненайденных белков подсемейства (ложноотрицательных результатов, "False negatives", FN) = 1
Чувствительность TP/(TP+FN) = 0,99
Селективность TP/(TP+FP) = 0,084 - Селективность моего паттерна - 0,84, подобрала порог 4,36 с селективностью 0,80
ROC-кривая для порога 4,36 в файле Excel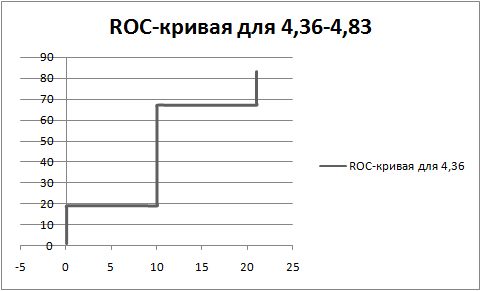
- Число верных находок ("True positive hits", TP) = 153
© Migur Anzhela 2010