
Нужно было вычислить информационное содержание (IC) последовательностей Козак в геноме Danio Rerio. Известно, что GC-состав Danio Rerio - 38.6%.
Для этого я выполнила следующие действия:
IC(b, j) = f(b, j) × w(b, j)При этом, если f(b, j) = 0, то IC(b, j) = 0
IC(j) = ∑IC(b, j)
Скачать таблицу с вычислениями можно по ссылке.

На странице практикума 6 я опубликовала всю выдачу MEME возможных сигналов коронавируса Human betacoronavirus 2c EMC/2012. Теперь среди предложенных мотивов я хочу отыскать такой, который бы подтвердил гипотезу о сигналах регуляции синтеза мРНК поздних генов.
Проверить я решила первую выдачу MEME, так она мне показалалась наиболее достоверной. Для этого я загрузила PMW первого мотива (создается MEME) в программу FIMO. Результат работы программы можно увидеть на Рисунке 2.
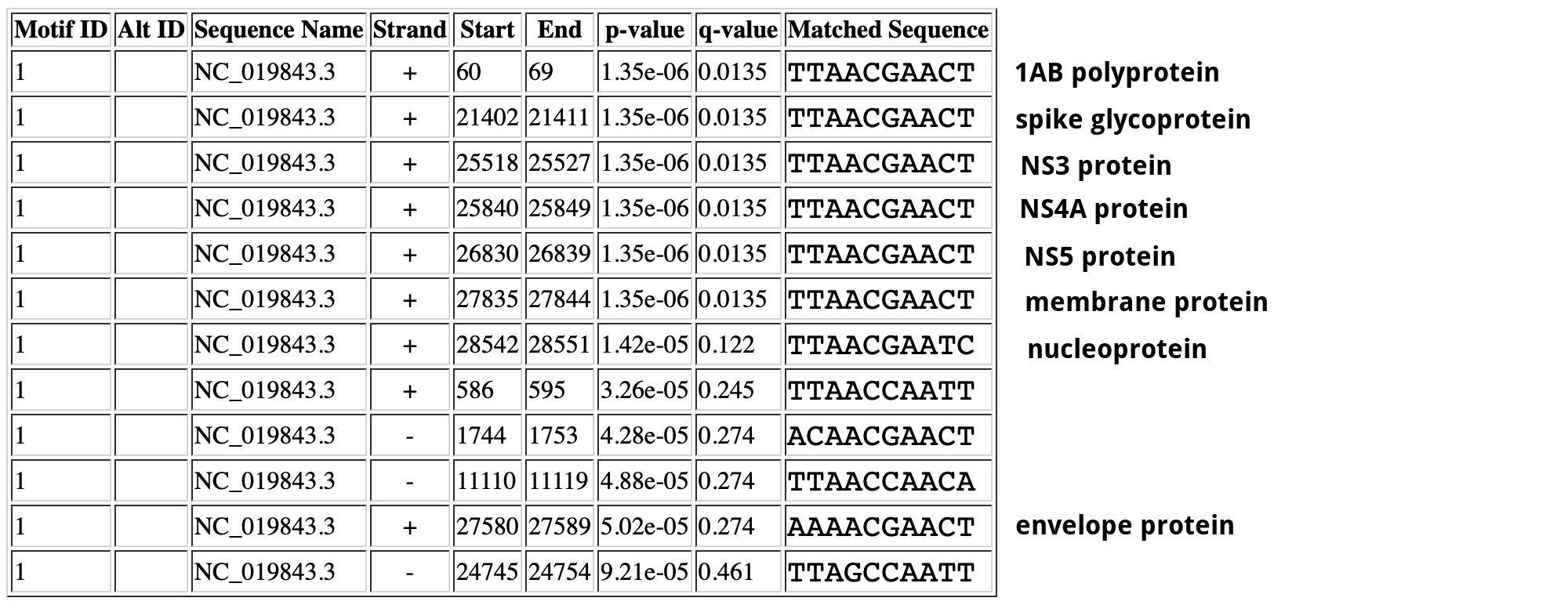
В выдаче программы FIMO было 12 находок, из которых 8 располагались перед генами. Другие 4 находки попали в гены, кодирующие белки: 3 оказались внутри гена ORF1ab полипротеина, 1 - внутри гена S.
Мне стало интересно, почему не перед каждым поздним геном нашелся сигнал. А именно сигнала не оказалось перед генами orf4b (кодирует NS4B protein) и orf8b (кодирует ORF8b protein). Я еще раз посмотрела на координаты генов в геноме (Таблица 1) и обнаружила, что ген orf8b находится внутри гена N. По ссылке можно увидеть их выравнивание. Преждевременной терминации трансляции белка гена N не происходит, потому что рамка считывания другая. Похожая ситуация с геном orf4b, он "наезжает" на ген orf4a. Возможно, у таких генов особый сигналинг.
| Ген | Координаты | Кодируемый белок |
| orf1ab | 279..21514 | 1AB polyprotein |
| S | 21456..25517 | spike glycoprotein |
| orf3 | 25532..25843 | NS3 protein |
| orf4a | 25852..26181 | NS4A protein |
| orf4b | 26093..26833 | NS4B protein |
| orf5 | 26840..27514 | NS5 protein |
| E | 27590..27838 | envelope protein |
| M | 27853..28512 | membrane protein |
| N | 28566..29807 | nucleoprotein |
| orf8b | 28762..29100 | ORF8b protein |
Информация, которую несёт мотив:
В википедии я взяла LOGO для последовательности Козак различных мРНК человека. Её можно увидеть на Рисунке 3. Затем для гена полипротеина и каждого позднего гена я взяла окружение старт-кодона (по 5 нуклеотидов до и после) и файл загрузила в MEME. На Рисунке 5 можно увидеть LOGO равнивания 'последовательностей Козак' вируса. Можно заметить, что на позиции -3 вируса действительно чаще встречается аденин, как и у последовательности Козак человек. Больше сходств я не нашла.
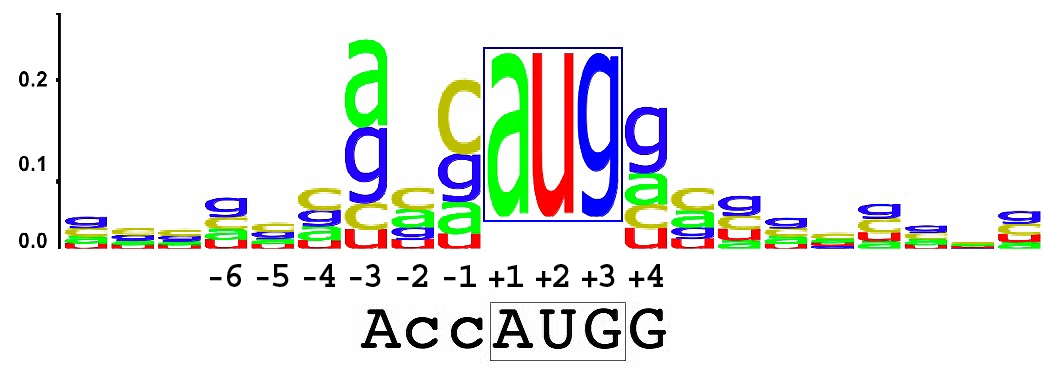
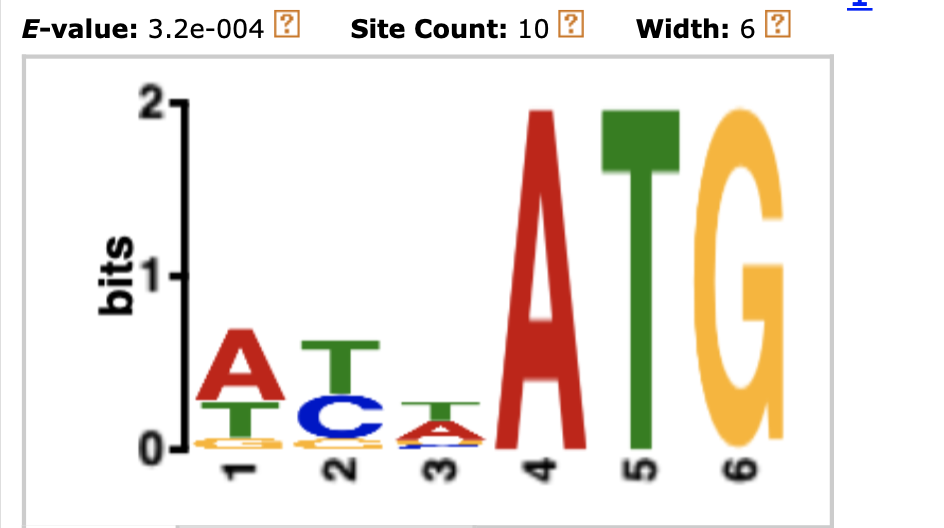
Для определения специфичности сигнала был выбран другой штамм Coronavirus Neoromicia/PML-PHE1/RSA/2011 вида Middle East respiratory syndrome-related coronavirus. Был выбран и более дальний родственник Severe acute respiratory syndrome coronavirus 2, который тоже относится к роду бетакоронавирусов. Выдача FIMO показана на Рисунке 5.
NC_044512.2 - SARS-2
KC869678.4 - Coronavirus Neoromicia/PML-PHE1/RSA/2011
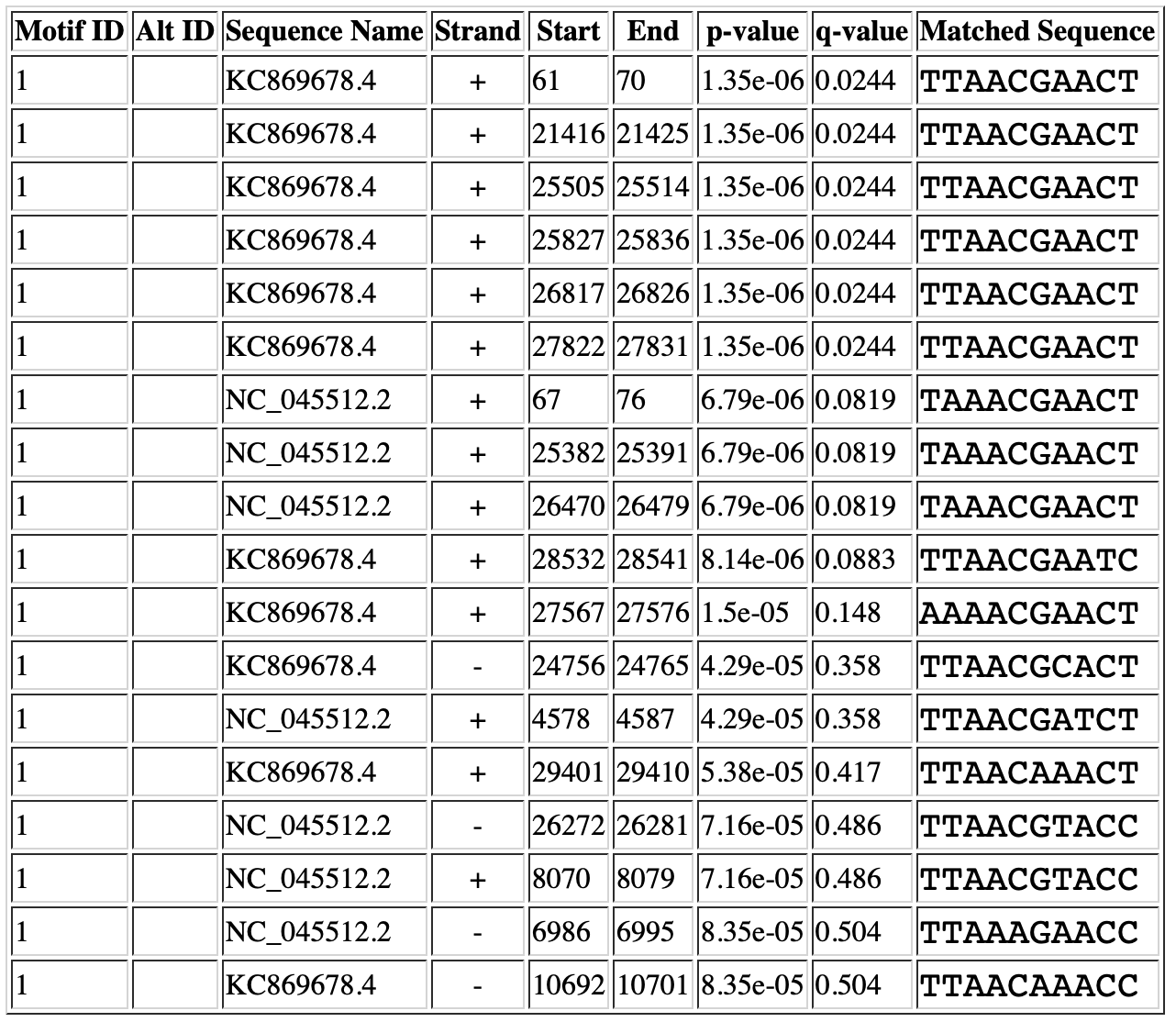
Находок довольно много, особенно в другом штамме. Причем, если убрать из рассмотрения находки на обратной цепи, наблюдается полное соответствие консенсусной последовательности. Также можно заметить, что нашлись сигналы в лидерной последовательно, что также является хорошим знаком.