

В данном практикуме нужно было осуществить поиск белков гомологичных CLPX_ECOLI (АТФ-связывающая субъединица ClpX протеазы Clp Escherichia coli) среди протеомов выбранных ранее бактерий.
Для начала был создан общий файл со всеми выбранными протеомами:
cat ARTS2.fasta CORDI.fasta MYCLE.fasta MYCTU.fasta NOCSJ.fasta RHOJR.fasta STRAW.fasta > proteomes.fasta
Далее blastp
makeblastdb -dbtype prot -in proteomes.fasta -out bac_db
blastp -query clpx_ecoli.fasta -num_threads 4 -db bac_db -evalue 0.001 -out blast_res.txt
Получено 19 находкок (файл с результатом ). Находки с наименьшим E-Value - АТФ-зависимые Clp протеазы всех бактерий (ATP-dependent Clp protease).
При помощи программы FastMe ("Gamma distributed rates across sites" — No, "Starting tree" — BIONJ, "No refinement", 100 бутстреп реплик) было реконструировано филогенетическое дерево найденных белков:
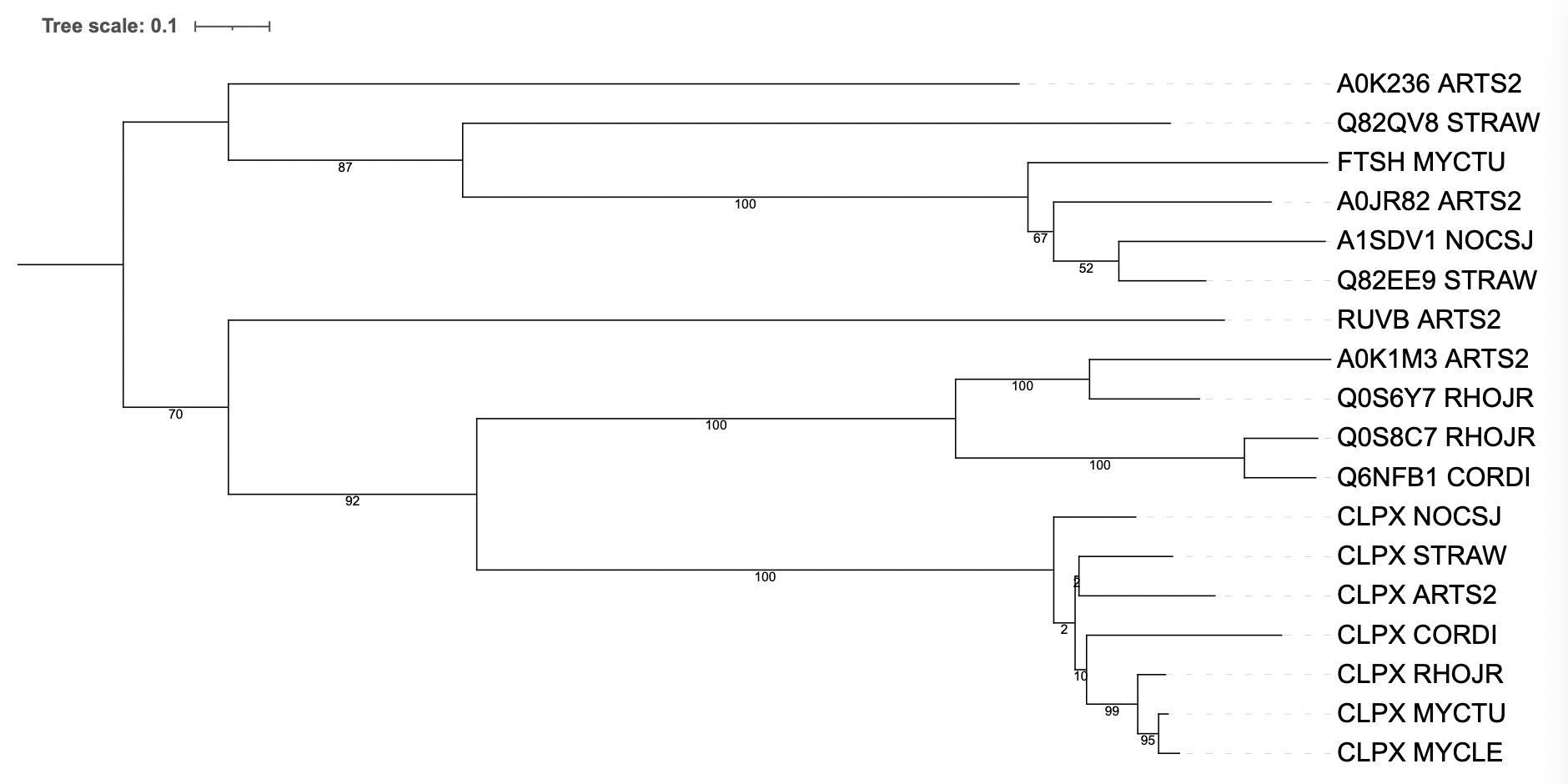
Рис. 1 Реконструированное филогенетическое дерево гомологов CLPX_ECOLI
Исходя из дерева можно выделить пары паралогов:
Также можно выделить и пары ортологов:
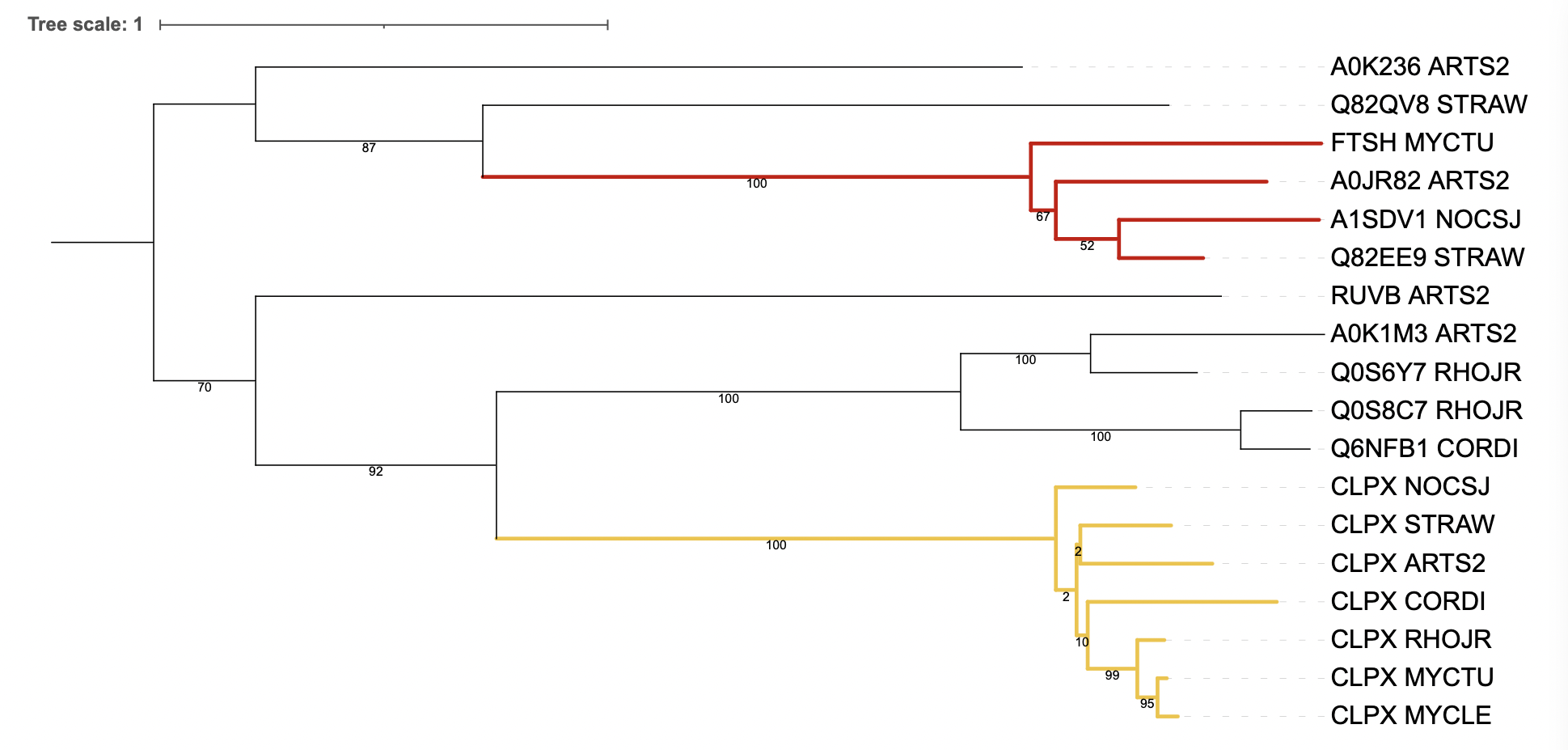
Рис. 2 Филогенетическое дерево гомологов CLPX_ECOLI с выделенными ортологическими группами
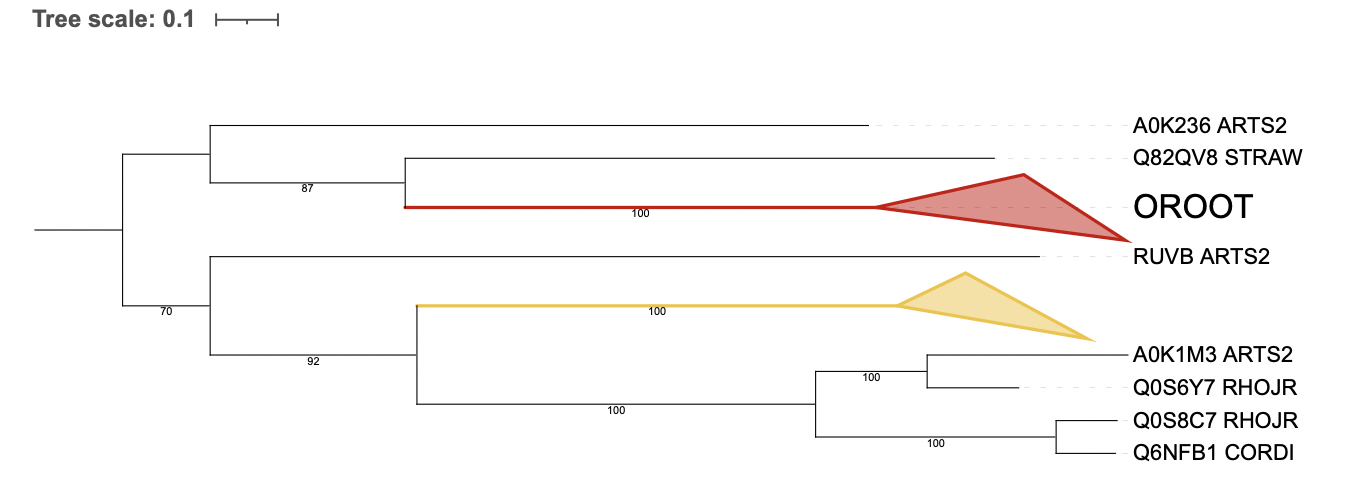
Рис. 3 Филогенетическое дерево с коллапсироваными ортологическими группами
Также для удобства сравнения дерево из первого практикума ("эталонное"):
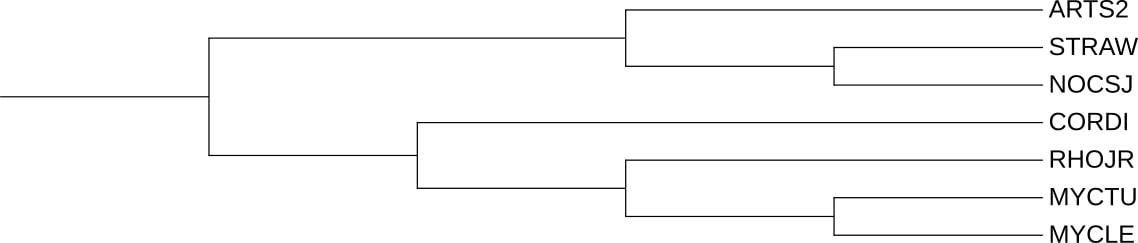
Рис. 4 Дерево из первого практикума
Жёлтым цветом обозначена группа АТФ-связывающих субъединиц протеаз Clp. Все эти белки встречаются по разу в каждой бактерии и имеют E-value "машииный" ноль. Ветвь несколько отличается от исходного дерева бактерий, но некритично: группа {MYCLE, MYCTU, RHOJR, CORDI} сохранила архитектуру, а взаиморасположение остальных трёх элементов "перепутано" (тут стоит сказать, что но бутстрэп поддержка этих ветвей очень низкая). Также можно заметить, что ветви сравнительно короткие, что может свидетелсьвтовать о быстрой эволюции и возможно неверном построении ветви.
Что касается красной группы, то она отлично сопоставляется с "эталоном". Группа {ARTS2, NOCSJ, STRAW} отделяется от далёкой MYCTU и при этом сохраняет внутреннюю иерархию.