Нуклеотидный BLAST
1. Таксономия и функции последовательности из №6
Являются частью 4 предыдущего задания.
2. Сравнение трёх алгоритмов поиска BLAST
Параметры поиска выбраны, исходя из задания (хотя требование "совпадают по всем остальным параметрам" нельзя выполнить в силу того, что возможный размер слова - Word size - у megablast и двух остальных алгоритмов не совпадает):
- Max target sequences = 1000
- Ajust for short queries = yes
- Expect threshold = 10
- Word size = 28 (megablast) / 11 (other)
- Max matches = 0
- Match/mismatch score = 2,-3
- Gap Costs = exist:5 extent:2
- Filter: low complexity regions
- Mask: for loopup table only
Поиск BLASTn по роду "Brada" (Taxonomy ID: 307617) выдаёт всего три результата. Расширяем область поиска до семейства Flabelligeridae (Taxonomy ID: 279648) и опять не попадаем в желанную 10-ку: 9 результатов в выдаче. Поднимаемся до порядка Flabelligerida - результат не меняется. Ограничение вышестоящим таксоном - инфраклассом Canalipalpata(Taxonomy ID: 105391) - уже радует нас 43-мя результатами в выдаче по алгоритму megablast:
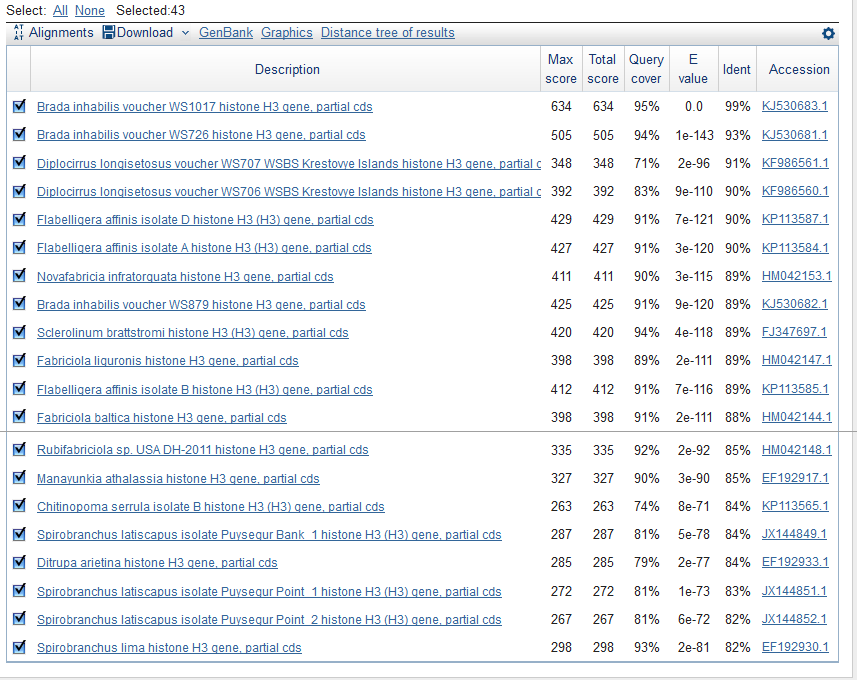
110-ю результатами выдачи по алгоритму discontiguous megablast:
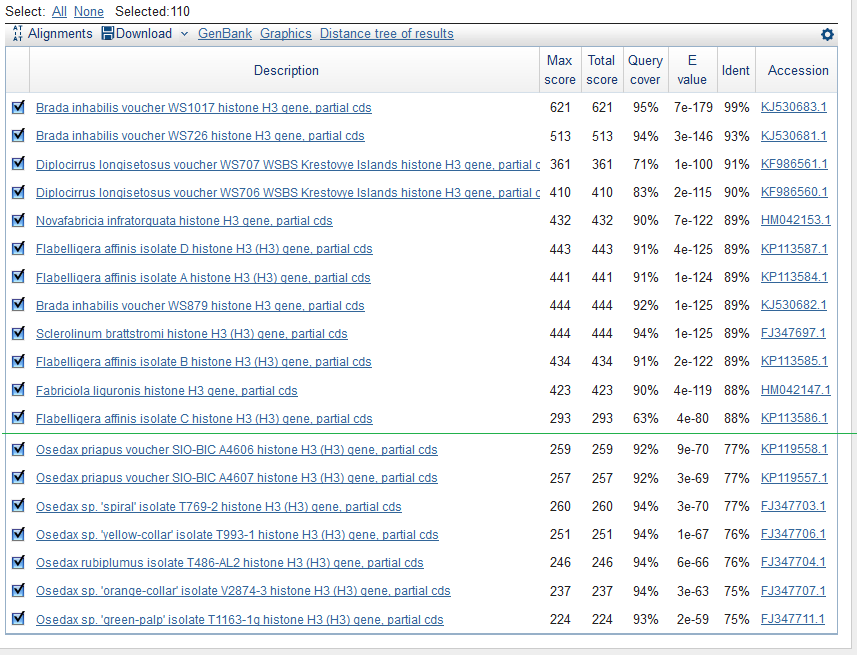
и 125-ю результатами выдачи по алгоритму blastn:
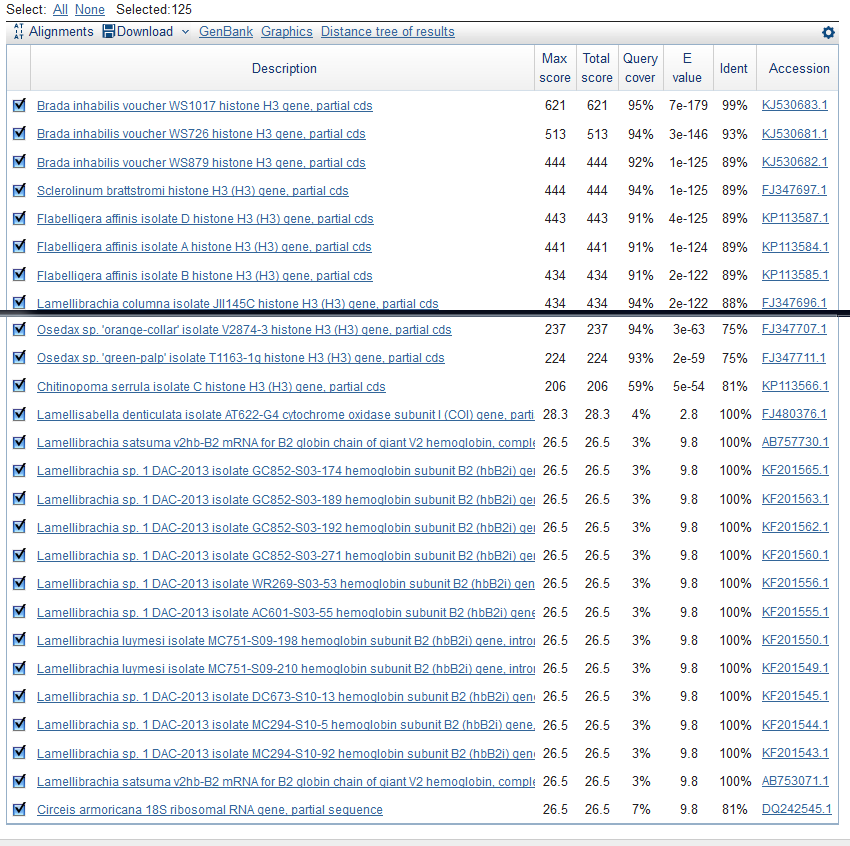
Различия в выдаче искались методом сопоставления подсвеченных CTRL+F находок на страничке каждого алгоритма. К примеру, поиск по роду "Novafabricia" выясветил 5 находок в выдаче blastn и discontiguous megablast и только 4 - в выдаче megablast.
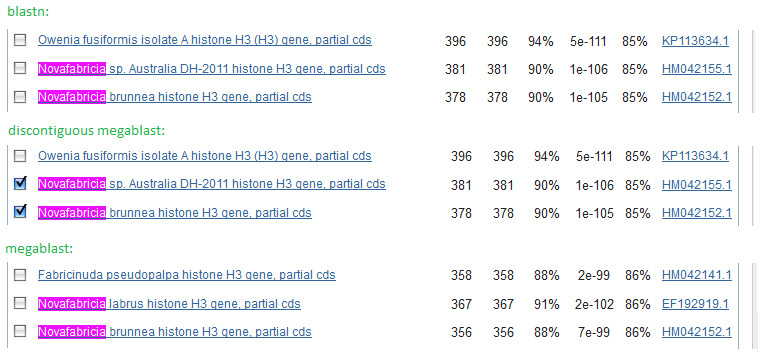
Разная длина слова привела к различающейся оценке качества выравнивания, поэтому в выдачу megablast не попал ген организма Novafabricia sp.
Также в выдачу blastn попал ген субъединицы B2 гемоглобина организма lamellibrachia sp.1, (это можно видеть на картинке с его выдачей). Причём со 100%-ной идентичностью запросу, который по пункту 4 в №7 кодирует гистон H3 в организме многощетинкового червя Brada %^) Вероятно, виноваты:
- неучтённое и слишком при том большое e-value, своим значением обязанное весьма скромному покрытию, взятому алгоритмом для анализа;
- маленькая длина слова.
Таблица результатов работы алгоритмов BLAST:
| Алгоритм |
Число результатов в выдаче: |
e-value (min/max) |
ident (max/min) |
| blastn |
125 |
7e-179 / 9.8 |
100% / 75% |
| discontiguous megablast |
110 |
7e-179 / 5e-54 |
99% / 75% |
| megablast |
43 |
0 / 2e-81 |
99% / 82% |
3. Поиск гомологов с помощью BLASTp
Используем tblastn, уточнив через uniprot.org AC белка, для которого ищем гомологи.
Поиск ведётся по базе RefSeq genomics, отбор результатов из выдачи ведётся по минимальному e-value и максимальной идентичности. С учётом того, что одна и та же последовательность может быть представлена в нескольких записях, не считаем дубликаты. Например, запись скэффолда, использованного при составлении генома, содержит тот же участок, что и запись этого генома.
Результаты поиска гомологов для 5 белков в задании сведены в тблицу:
Таблица результатов работы алгоритмов BLAST:
| Белок |
Лучшая находка |
Заключение |
| Функция: | Белок теплового шока |
| Масса, КДа: | 71 |
| ID: | HSP7C_HUMAN |
| AC: | P11142 |
| Файл: | P11142.fasta |
|
| среди | ID записи | % идентичных позиций | % покрытия | e-value |
| 7 | NC_006611.3 | 92 | 94 | 0 |
|
Гены, кодирующие гомологи HSP7C_HUMAN, найдены на 8-ой, 12-ой, 19-ой, 20-ой, 29-ой и Х-хромосоме. Лучшее соответствие было в скэффолде, который не смогли разместить в геноме (unplaced), поэтому данные приведены для 29-ой хромосомы.
Если понизить планку для e-value в соответствии с оценкой гомологичности TERT - следующего белка в таблице, то "хороших" находок получается 12,(на 2-ой, 4-ой,5-ой, 9-ой и 23-ей хромосомах). Но для окончательного убеждения в гомологичности требуется проверить аннотацию для каждой из них, что очень долго и не продиктовано заданием. |
| Функция: | Теломераза (обратная транскриптаза) |
| Масса, КДа: | 126,997 |
| ID: | TERT_HUMAN |
| AC: | O14746 |
| Файл: | O14746.fasta |
|
| среди | 1 |
| ID записи | NC_006616.3 |
| % идентичных позиций | 57 |
| покрытие | 87 |
| e-value | 8e-68 |
|
Находка только одна, и степень гомологичности гораздо ниже по всем параметрам. И хотя "взглянуть" на последовательность белка массой 126КДа не выходит, можно найти в аннотации (ссылка Gene в секции Related info, упорядочиваем по хромосоме и ищем по номеру начала гена, который отображается в выдаче tblastn в выравнивании найденных последовательностей), что гомолог также определён как TERT. |
| Функция: | Цитрат-синтаза |
| Масса, КДа: | 51,712 |
| ID: | CISY_HUMAN |
| AC: | O75390 |
| Файл: | O75390.fasta |
|
| среди | 2 |
| ID записи | NC_006599.3 |
| % идентичных позиций | 91 |
| покрытие | 96 |
| e-value> | 0 |
|
Гомологи найдены на 10-ой и 17-ой хромосоме. С учётом предыдущего опыта смотрим аннотацию для обеих находок, поскольку их оценки гомологичности гораздо лучше таковых у предыдущего белка. Обе оказались аннотированы как citrate synthase |
| Функция: | Субъединица RPB1 ДНК-направляемой РНК-полимеразы II |
| Масса, КДа: | 217,176 |
| ID: | RPB1_HUMAN |
| AC: | P24928 |
| Файл: | P24928.fasta |
|
| среди | 1 |
| ID записи | NC_006587.3 |
| % идентичных позиций | 69 |
| покрытие | 78 |
| e-value | 4e-130 |
|
Всего находок 3, однако уже у второй по качеству покрытие составило 26%, поэтому гомологом считаем только первую. |
| Функция: | Полиаденилат-связывающий протеин 2 |
| Масса, КДа: | 32,749 |
| ID: | PABP2_HUMAN |
| AC: | Q86U42 |
| Файл: | Q86U42.fasta |
|
| среди | 1 |
| ID записи | NC_006590.3 |
| % идентичных позиций | 93 |
| покрытие | 55 |
| e-value | 4e-29 |
|
Несмотря на высокое e-value, которое вполне ожидаемо с учётом размера белка, считаем гомологом первую находку. Аннотация это подтверждает: ген, начинающийся с 3580427-ого нуклеотида, обозначен как poly(A) binding protein |
4. Сходство геномов вирусов
Для анализа взята ДНК пяти разновидностей (то есть разноштаммностей) герпес-вируса человека (Viruses; dsDNA viruses, no RNA stage; Herpesvirales; Herpesviridae; Alphaherpesvirinae; Simplexvirus):
- Human herpesvirus 1 strain KOS 152011 bp , AC: JQ673480
- Human herpesvirus 1 strain KOS(2) 151024 bp, AC: JQ780693
- Human herpesvirus 1 isolate KOS 151974 bp, AC: KT899744
- Human herpesvirus 1 strain KOS79 135472 bp, AC: KT425109
- Human herpesvirus 1 strain 17 152222 bp, AC: NC_001806
Последовательность действий для получения сходства:
ls > HVir1Lst //создаём список файлов с последовательностями, подлежащими объединению
//не очень удачно: пришлось удалять первую строку списка, содержащую имя файла списка, поскольку вопреки моим ожиданиям сначала созаётся выходной файл, а уже потом выполняется команда ls - ассоциативность справа налево налицо. Надо было фильтровать имена для списка.
seqret @HVir1Lst hvir1_5in1.fasta //объединяем 5 последовательностей в один файл
makeblastdb -in hvir1_5in1.fasta -dbtype nucl //для tblastx создана база данных
tblastx -query hvir1_5in1.fasta -db hvir1_5in1.fasta -out hvir1_5in1_blast.out -outfmt 7 //поучаем вывод tblastx, содержит 10024 строки
python revise_blast_7.py --infile hvir1_5in1_blast.out --identity 50 --max_exp 5 --min_bits 20 --outfile hvir1_similar //обрабатываем скриптом, любезно предоставленным для выполнения работы (лежит здесь), число строк сокращается почти вдвое (5352 строки)
После вставки содержимго hvir1_similar в excel-таблицу получаем файл: HVir1_table.xls. Упорядочиваем совпадения по имени последовательности, поскольку ниже, чем 81% идентичности, у разных штаммов не наблюдается, да и тех, околовосьмидесятипроцентных, всего 16. Получим группы совпадений, в первой из которых будут совпадения первого штамма со семи остальными, во второй- совпадения второго штамма со всеми остальными, кроме первого, в третьей - со всеми, кроме первого и второго, и так далее. По числу совпадений оценим различия между штаммами.
| id штаммма запроса | id ответного штамма | число совпадений | средняя длина |
| JQ673480 | JQ780693 | 453 | 285,75 |
| JQ673480 | KT425109 | 576 | 270,1 |
| JQ673480 | KT899744 | 429 | 287 |
| JQ673480 | NC_001806 | 557 | 272,1 |
| JQ780693 | KT425109 | 582 | 268,9 |
| JQ780693 | KT899744 | 446 | 285,37 |
| JQ780693 | NC_001806 | 561 | 273,9 |
| KT425109 | KT899744 | 583 | 269 |
| KT425109 | NC_001806 | 539 | 264,9 |
| KT899744 | NC_001806 | 557 | 273 |
Наибольшее число совпадений - 583- у isolate KOS и KOS79, однако и средняя длина совпадения у них наименьшая, и идентичность худшая, а по длине последовательности сильно разнятся. Наименьшее число совпадений - 429 - у штаммов KOS и isolate KOS, однако средняя длина совпадения наивысшая, и идентичность почти на всех участках 100%. Поэтому схожесть штаммов KOS и isolate KOS представляется более высокой.
|



