Сборка de novo
1.Сборка контигов
Сборка из чтений 20-й хромосомы, подчищенных триммером в предыдущем практикуме, состоит из 2х этапов. На первом надо выделить k-меры - все возможные последовательности длины k. То есть двигаем рамку в k-позиций для символов на один символ, таких сдвигов возможно на k-1 меньше, чем символов в последовательности.
k-меры выделялись с помощью velveth:
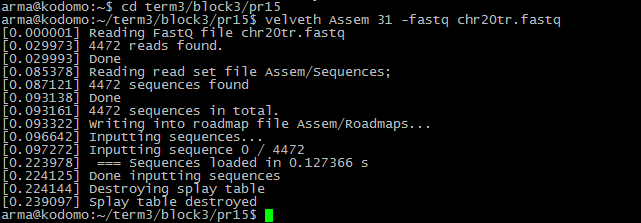
Далее k-меры выравниваются, и полностью совпадающие соседи заносятся в список вершин графа де Брёйна, соединённых ориентированным ребром. Маршрут обозначает контиг.
Сборка контигов по графу де Брёйна посредством velvetg:
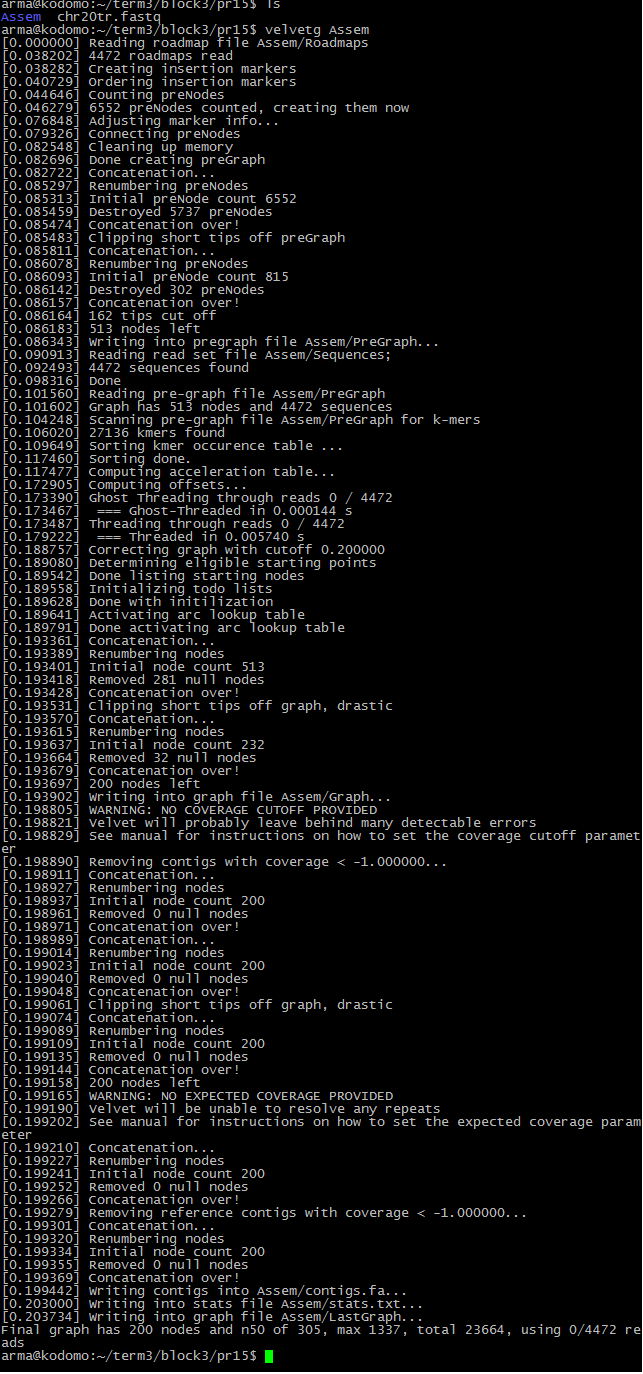
Сводная таблица:
| k-мер | контигов(=узлов графа) | n50 | Максимальная длина | Всего картировалось |
| 27136 | 200 | 305 | 1337 | 23664 |
Контигов много, и они короткие, пол-генома(20ой хромосомы) покрывается длинами начиная от 305. Сборка плохого качества и достоверно картироваться не будет.
2.Картирование контигов
Для картирования используем blastn. Создаём базу по 20 хромосоме и используем контиги из Assem/contigs.fa. Найденные картирования отражаем в файлах rslt(просто данные) и rslt_hdrs(данные с подписями):
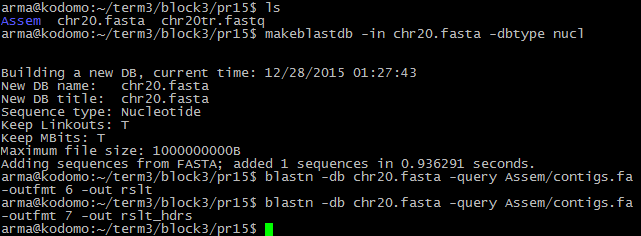
Данные заносим в Excel-файл для подсчёта частоты встречаемости контига и длины непокрытых участков. Сортируем по возрастанию столбца s.start и вычитаем соответствующие значения конца предыдущего контига из начала следующего. Там, где встречаются отрицательные значения, имеет место перекрытие контигов. Большая длина разрыва - скорее всего, интронная вставка, поскольку сборка состоит из экзонов.
Итоги: рекордсменом по частоте стал контиг NODE_194_length_282_cov_6.202127 - 2199 вхождений, только 68 контигов вошли однократно, причём многие - с перекрытиями. Перекрывающиеся контиги не были посчтаны за единый, потому что они позиционируются не по 100%-ному совпадению, а по лучшему качеству выравнивания, а граф строился по 100%ному совпадению.
|


