Трансмембранные белки, занятие 6Задание 1Белок из первого семестра, UDP-N-ацетилглюкозамин1-карбоксивинилтрансфераза бактерии Hippea maritima (cellular organisms; Bacteria; Proteobacteria; delta/epsilon subdivisions; Deltaproteobacteria; Desulfurellales; Desulfurellaceae; Hippea; Hippea maritima) не является трансмембранным (проверила на TMHMM заранее, чтоб не тратить время впустую). Поэтому был взят другой белок той же бактерии: транспортёр магния, magnesium transporter (найден в списке белков Hippea maritima по слову "transporter", поскольку транспортёр почти наверняка трансмембранный) (mag_trans_hipma.fasta). Поиск гомологов проводился BLAST'ом ресурса EMBL-EBI с границей идентичности (SCORES) 50% по базе UniProt Clusters 50% с ограничением количества выдаваемых результатов (ALIGNMENTS) не более 200. Мой белок длиной 457 а.о. оказался среди 200 найденных первым, остальные результаты шли с большим отрывом по идентичности: лучший имел length=448, Score=396.4, Identities=46.4, Positives=67.9, p-value=9.0E-131. Наихудший p-value был 6.1E-68.Редактирование выравниванияQ20K00 был выбран за слишком длинный левый N-хвост. Во-первых, по предсказанию TMHMM трансмембранные участки находятся справа, в последней сотне АК. Во-вторых, у белка, для которого существует pdb, на месте этого хвоста ничего нет, то есть разметить там ничего не удастся. Но удалён был за не очень хорошее совпадение с консенсусом. После чего Edit->Remove empty columns сильно облегчило вид. Последовательности с 154 по 174ую были удалены, потому что лучше совпадали между собой, чем с общим выравниванием, что привносило неясность в консенсус. Остальное удалению не подлежало: UPI00042A454B не имела хвоста, но трансмембранная часть совпадала хорошо. Что показывает, что хвост не влияет на способность транспортировать Mg2+ ВизуализацияДля визуализации структуры был взят pdb-файл гомолога из списка, поскольку в PDB моего белка нет. Вообще вероятность существования pdb-файла для случайно взятого белка очень низка, поэтому я просто по запросу "magnesium transporter" на uniprot.org в таблице результатов поиска брала id белков, для которых pdb-файл есть, и искала по CTRL+F этот id среди своих 200 гомологов. Наличие pdb-файла для белка в списке на uniprot показано в столбце "pdb cross-reference". Столбец надо включить в настройках отображаемых стобцов "columns". В итоге нашёлся pdb-файл для гомолога Thermus thermophilus, id = Q5SMG8. Его id в PDB: 4U9L. В строке выравнивания в JalView щёлкаем на Q5SMG8 и в контекстном меню связываем с ним 3d-структуру, вводя её id в PDB. JalView скачивает структуру, открывает её в JMol-окошке и сопоставляет буквы аминокислот белка в выравнивании с участками белка в окошке. Теперь можно определить трансмембранные участки визуально. Разметка трансмембранных участковНа левой белой части в JalView кликакм контекстное "Add row", вписываем имя строки. Выделяем по верхним цифрам сплошняком участок с нужными номерами остатков (трансмембранный), приписываем строку аннотации "М" (контекстное->label). Разметка по TMHMM не совпала с разметкой по 3d-модели, TMHMM не определил трансмембранность участка 311-360. В проекте JalView можно посмотреть разметку, выглядит примерно так: (участок) 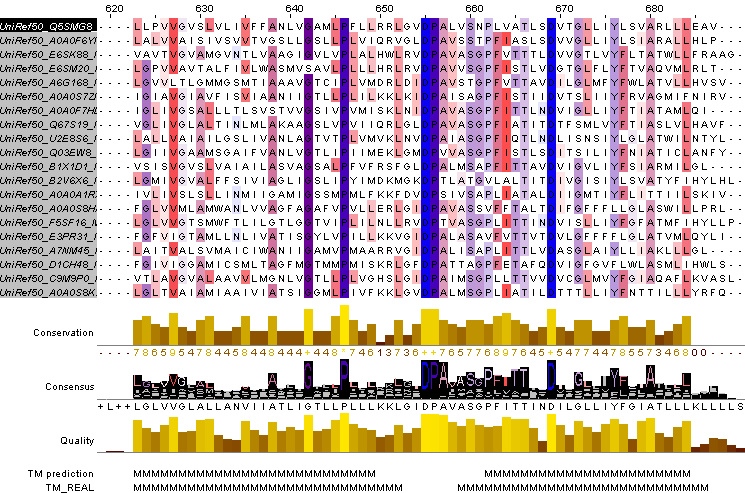 Остальные участки совпадают. В JMol можно наблюдать консервативные по гидрофобности и по идентичности участки, создающие трансмембранную структуру белка (оставлена одна субъединица для наглядности, красные = гидрофобные, синие = гидрофильные заряженные, фиолетовые = гидрофильные незаряженные): 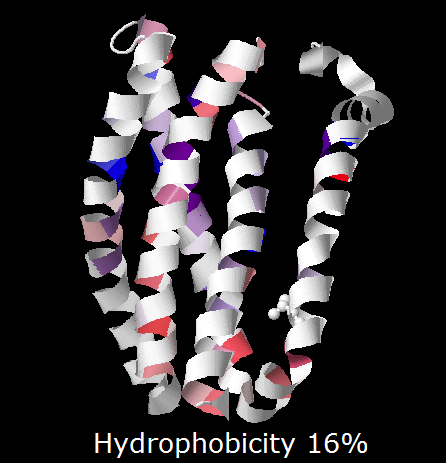 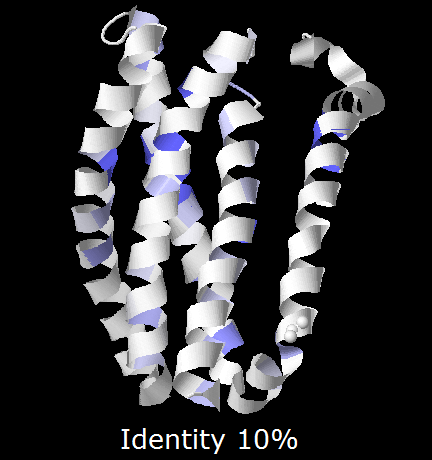 Для исходного белка Hippea maritima TMHMM выдал похожую структуру: можно сравнить два предсказания: 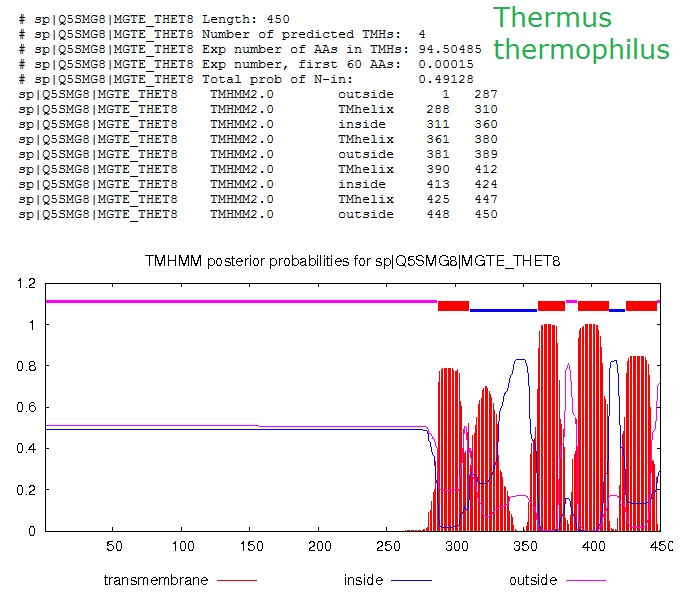 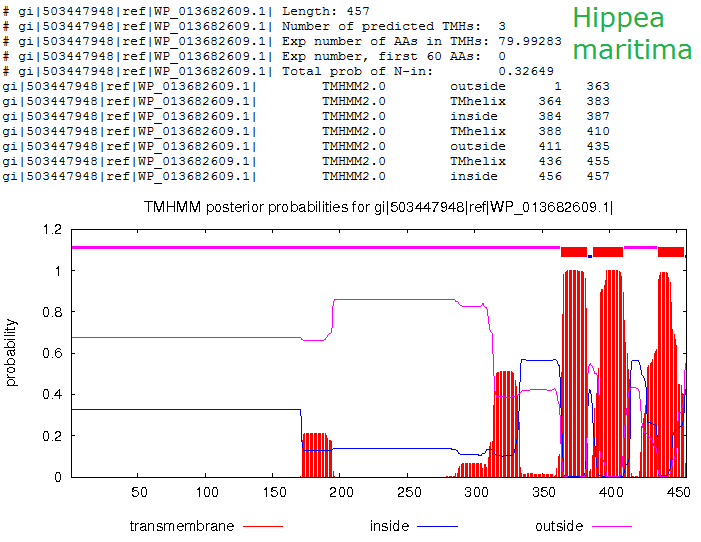 Ориентацию белка можно посмотреть на OPM, она выглядит так: 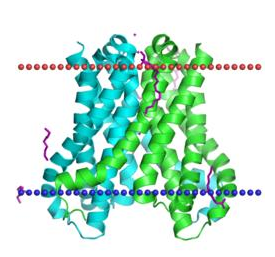 Участки по обе стороны мембраны довольно консервативны, чаще всего встречаются Pro, Arg, Lys, Asp, Glu и Asn. Участки в мембране консервативны только по гидрофобным частям, в целом консервативность их низкая. |