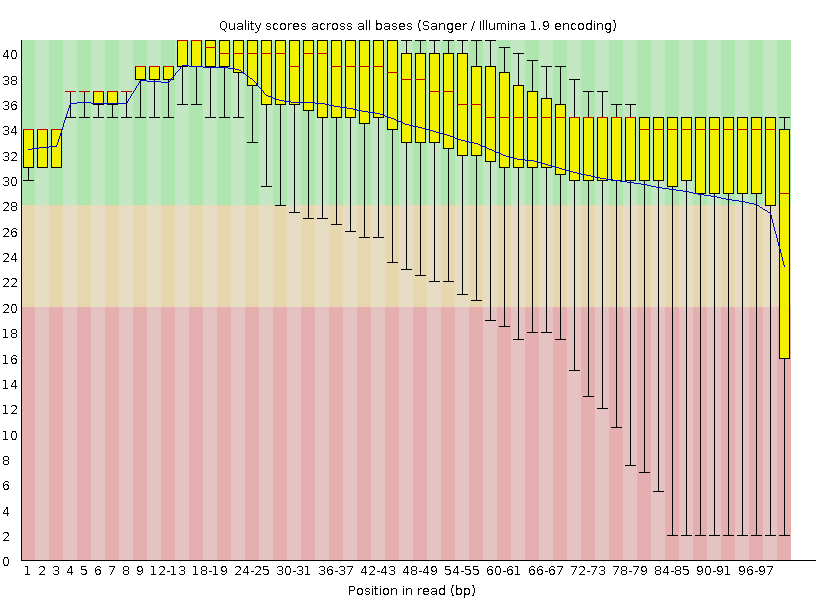
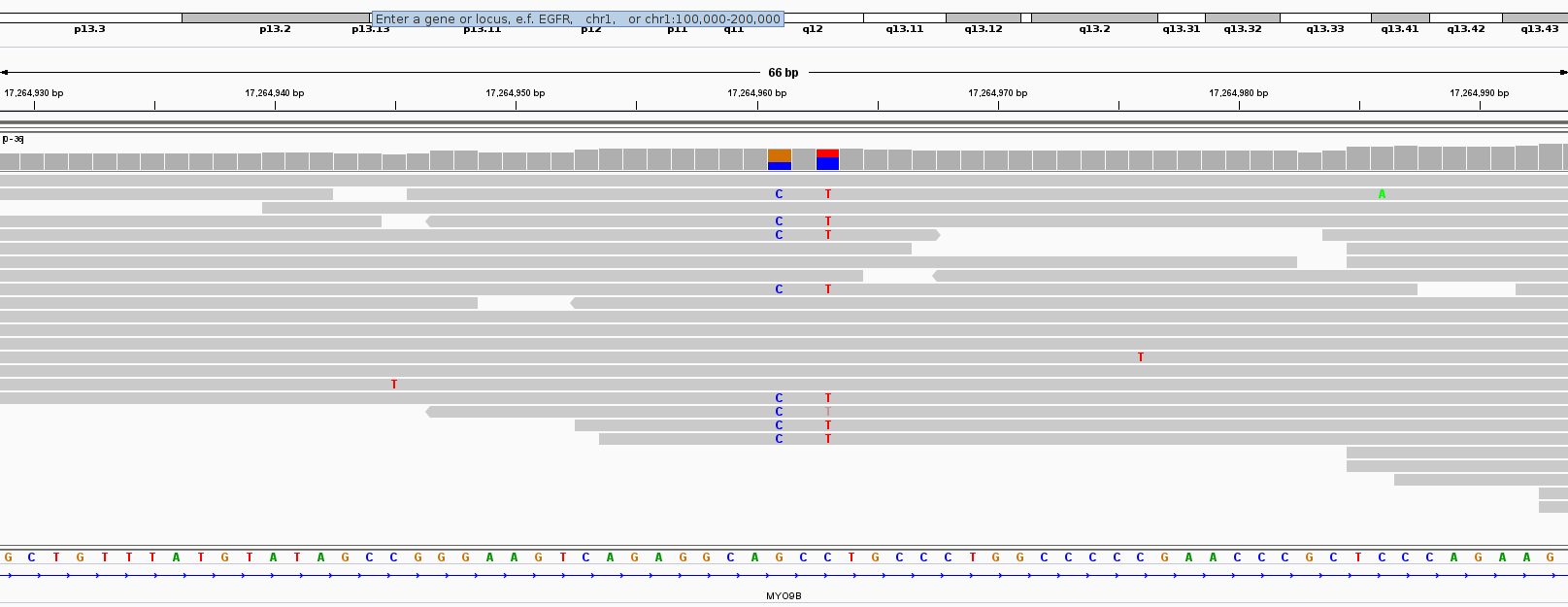
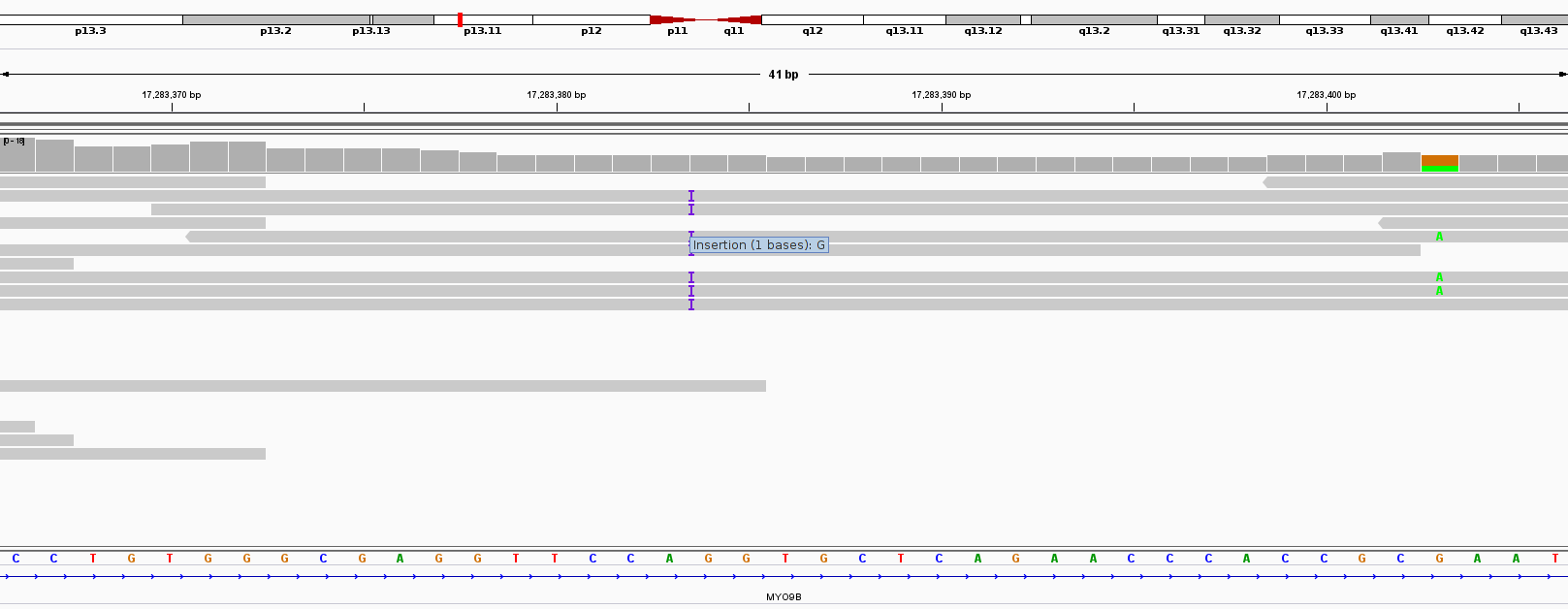
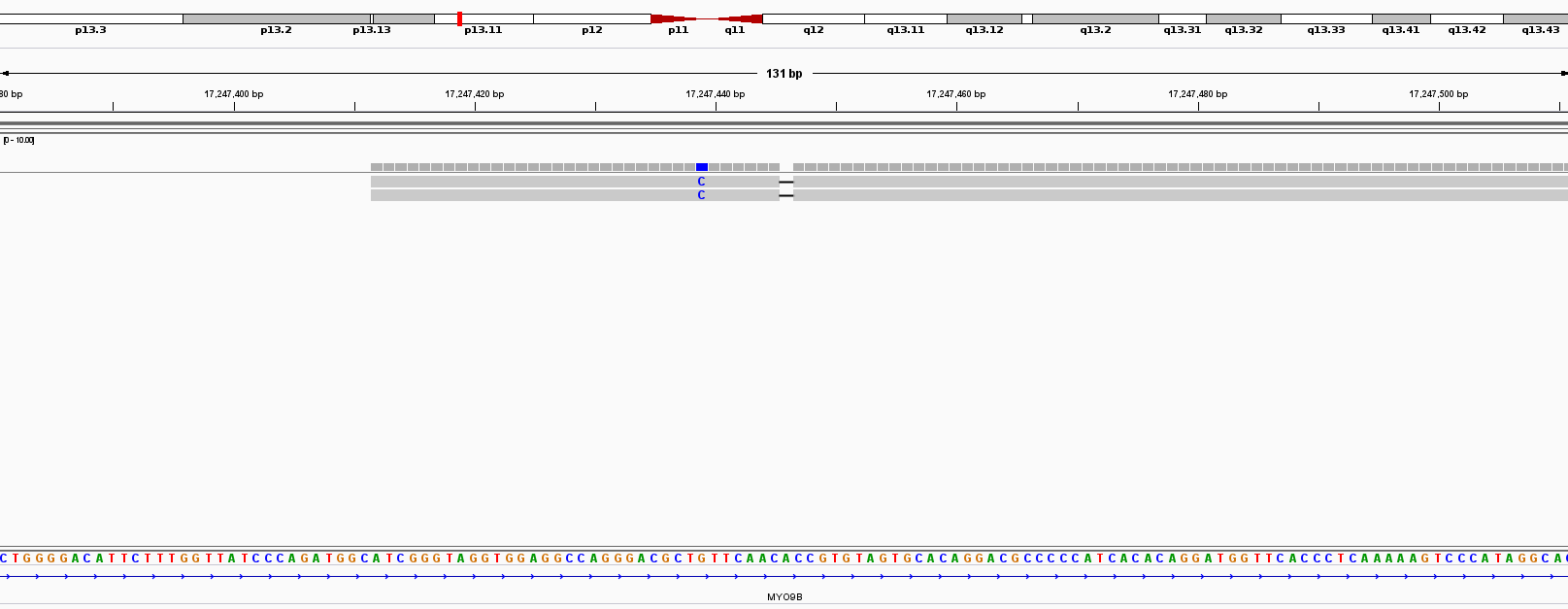
Число чтений до чистки: 5524;
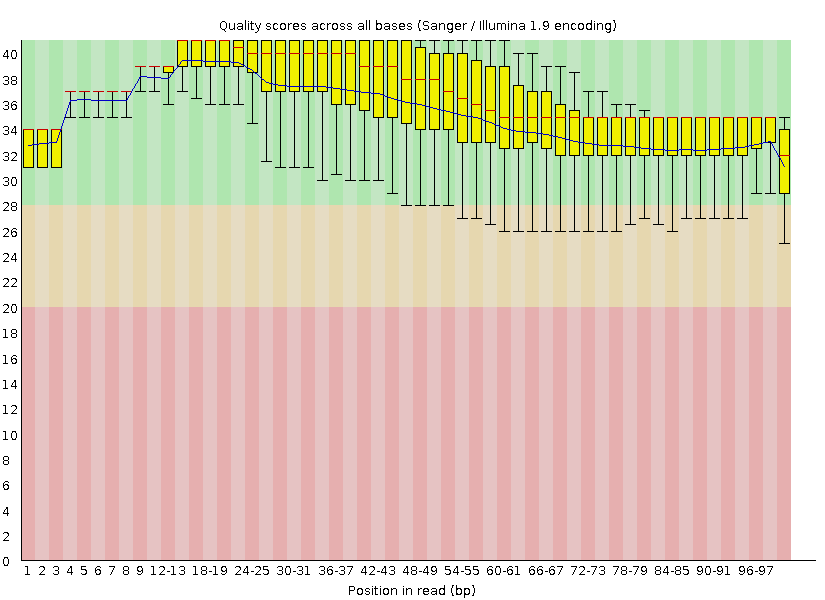
Число чтений после чистки: 5227;
Отсеялись все чтения длиной качественной части менее 50.
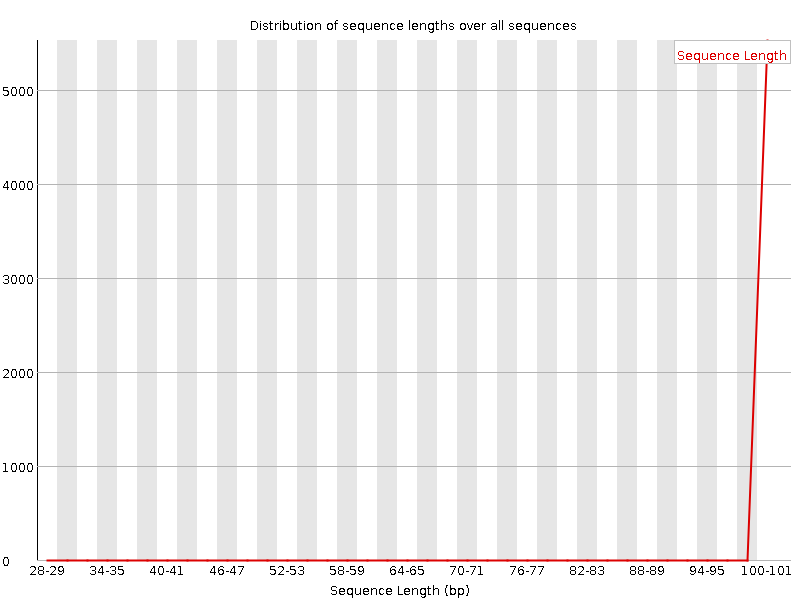
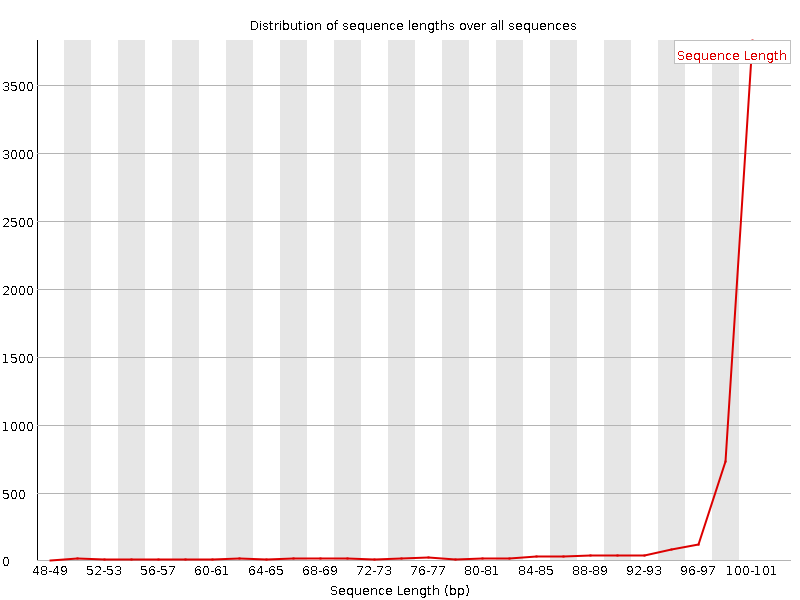
Остальная выдача FastQC имеет незначительные различия; упоминания стоит лишь распределение последовательностей по длине:
Из этой выдачи видно, что последовательности длиной более 100 часто укорачивались из-за низкокачественных концевых участков, а последовательности длиной менее 50 нуклеотидов выбрасывались.