Задание 1. Сравнение предсказаний трансмембранных участков в бета-листовом белке
Type: Transmembrane (Трансмембранные белки)
Class: Beta-barrel transmembrane (Белки с β-листами в трансмембранной части)
Superfamily: Ligand-gated protein channels (Лиганд-зависимые ионные каналы)
Family: Outer Membrane Receptor (OMR) (Рецепторы внешней мембраны)
Protein Name: Colicin I receptor (Рецетпор колицина I)
Species: Escherichia coli
Localization: Bacterial Gram-negative outer membrane (Наружная мембрана грам-отрицательных бактерий)
PDB ID: 2hdf
Uniprot: CIRA_ECOLI
Function:
Рецептор колицина I - рецепторный белок, который связывает белок Колицин Ia.
Колицины (специфичные для E. coli бактериоцины) представляют собой токсичные молекулы, выделяемые некоторыми штаммами бактерий
для уничтожения других бактерий во время стресса. Рецептор колицина I представляет собой TonB-зависимый транспортер,
экспрессируемый многими штаммами E. coli для связывания и транспорта Fe 3+ в комплексе с линейными катехолатами через внешнюю мембрану.
Когда колоцин I связывается с белком-рецептором колоцина I на поверхности бактериальной клетки, он вызывает изменение конформации рецептора, и он становится ионным каналом в клеточной мембране.
Это, в свою очередь, приводит к выходу ионов и других молекул из клетки, нарушению клеточного гомеостаза и, в конечном итоге, к гибели бактериальной клетки.
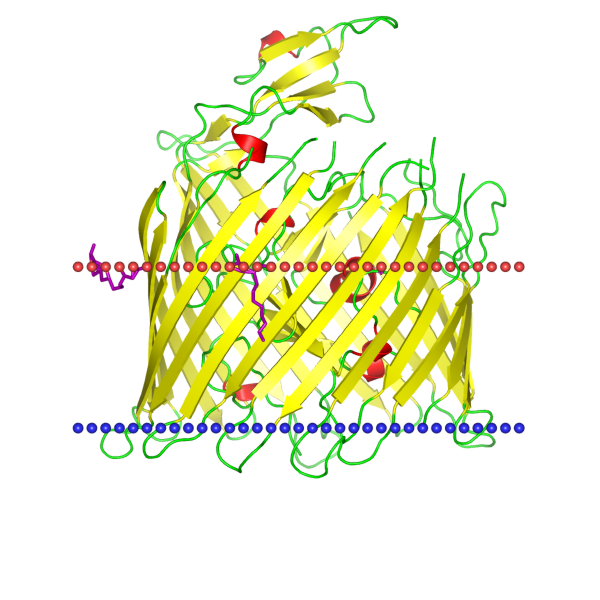
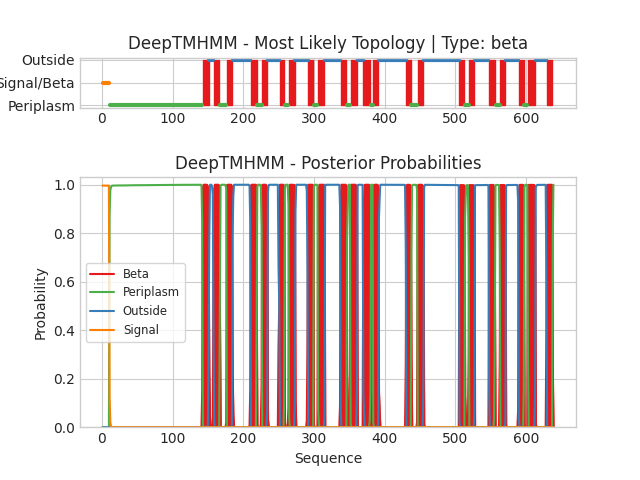
Из записи PDB был скача файл последовательности и загружен в DeepTMHMM. Согласно б/д OPM белок содержит 22 трансмембранных участка (далее - ТМ участки). Выдача DeepTMHMM тоже показала наличие 22 ТМ участков (cсылка на файл выдачи DeepTMHMM). Координаты ТМ участков по данным OPM и DeepTMHMM представлены в Таблице 1 в том порядке, в котором они были обозначены в этих источниках.
| Номер Петли | OPM | DeepTMHMM |
|---|---|---|
| 1 | 164-173 | 143-151 (*) |
| 2 | 184-192 | 158-166 |
| 3 | 199-208 | 177-184 (*) |
| 4 | 235-243 | 211-219 (*) |
| 5 | 250-258 | 227-234 (*) |
| 6 | 275-283 | 252-258 |
| 7 | 288-296 | 265-273(*) |
| 8 | 315-323 | 292-299 |
| 9 | 330-338 | 306-315 (*) |
| 10 | 362-371 | 338-346 (*) |
| 11 | 376-384 | 351-361 (*) |
| 12 | 395-403 | 370-380 |
| 13 | 408-415 | 384-391 (*) |
| 14 | 455-463 | 431-437 (*) |
| 15 | 471-477 | 447-455 |
| 16 | 531-538 | 506-513 (*) |
| 17 | 543-550 | 520-527 (*) |
| 18 | 573-579 | 548-556 |
| 19 | 587-593 | 564-571 (*) |
| 20 | 615-624 | 590-597 |
| 21 | 627-634 | 603-613 (*) |
| 22 | 655-662 | 630-637 |
Мы видим, что координаты OPM и DeepTMHMM сильно отличаются. DeepTMHMM определил 14 уникальных ТМ участка, которые не имеют пересечения с ТМ учатсками OPM. Делаем вывод, что данные не консистентны. Почему так могло произойти? Ответить точно на этот вопрос довольно сложно, потому что из за специфики моделей глубокого обчения мы не можем точно знать, на основании они выделили эти уникальные участки. Посмотрев на 3D структуру этого белка было замечено, что у него есть некая специфика в структуре: он представляет из себя группу из β-листов, вложенных в цилиндр из β-листов, то есть его ТМ участок имеет 2 слоя (рис). Мы так же знаем, что координаты ТМ участков в базе данных OPM были получены исходя из экспериментальной стрктуры, а DeepTMHMM использует информацию только о послежовательности. На основании этого выдвинуто 2 гипотезы: либо была получена некачетсвенная структура, либо модель DeepTMHMM плохо работает с такими белками.
Для проверки этой гипотезы были выбраны другие два белка которые тоже имеют большой вложенный участок из β-листов и для них был запущен DeepTMHMM. Эти белки это 4EPA (Pesticin receptor FyuA) и 5FR8 (Ferric enterobactin receptor PirA). Выдача DeepTMHMM для 4EPA и 5FR8. Так же DeepTMHMM был запущен для белка 6TZK (Cellulose synthase operon protein C) без большой вложенной структуры из β-листов (выдача). Конечно на основании пары белков однозначные выводы делать сложно, но мы видим, что погрешность коррдинат ТМ учатсков для белков 4EPA и 5FR8 немного больше чем для 6TZK. Однако такого координального сдвига, как для выбранного мною белка нет.
Задание 2. Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Поскольку на сервере PPM 3.0 возникли неполадки, и задача не смогла досчитаться за неделю, то по согласованию с преподавателем мною был взят другой белок: SAP_MYCTU (ID: O07802) (экспортер сульфолипида-1). Он необходим для транспорта через внутреннюю мембрану сульфолипида-1, который является основным липидом клеточной стенки патогенных микобактерий.
Последовательность была взята из UniProt. Скачать файл выдачи DeepTMHMM
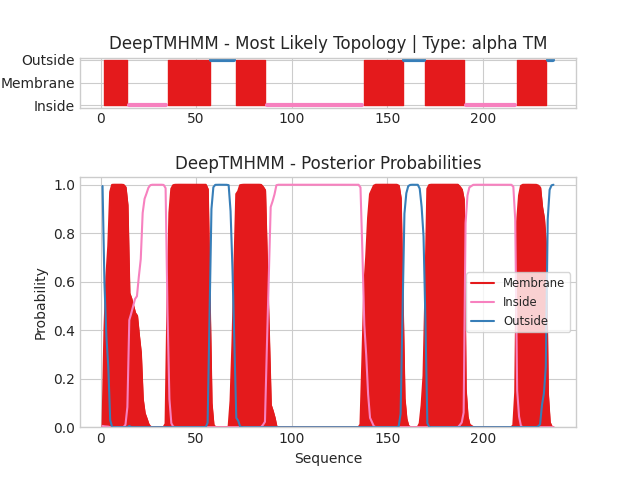
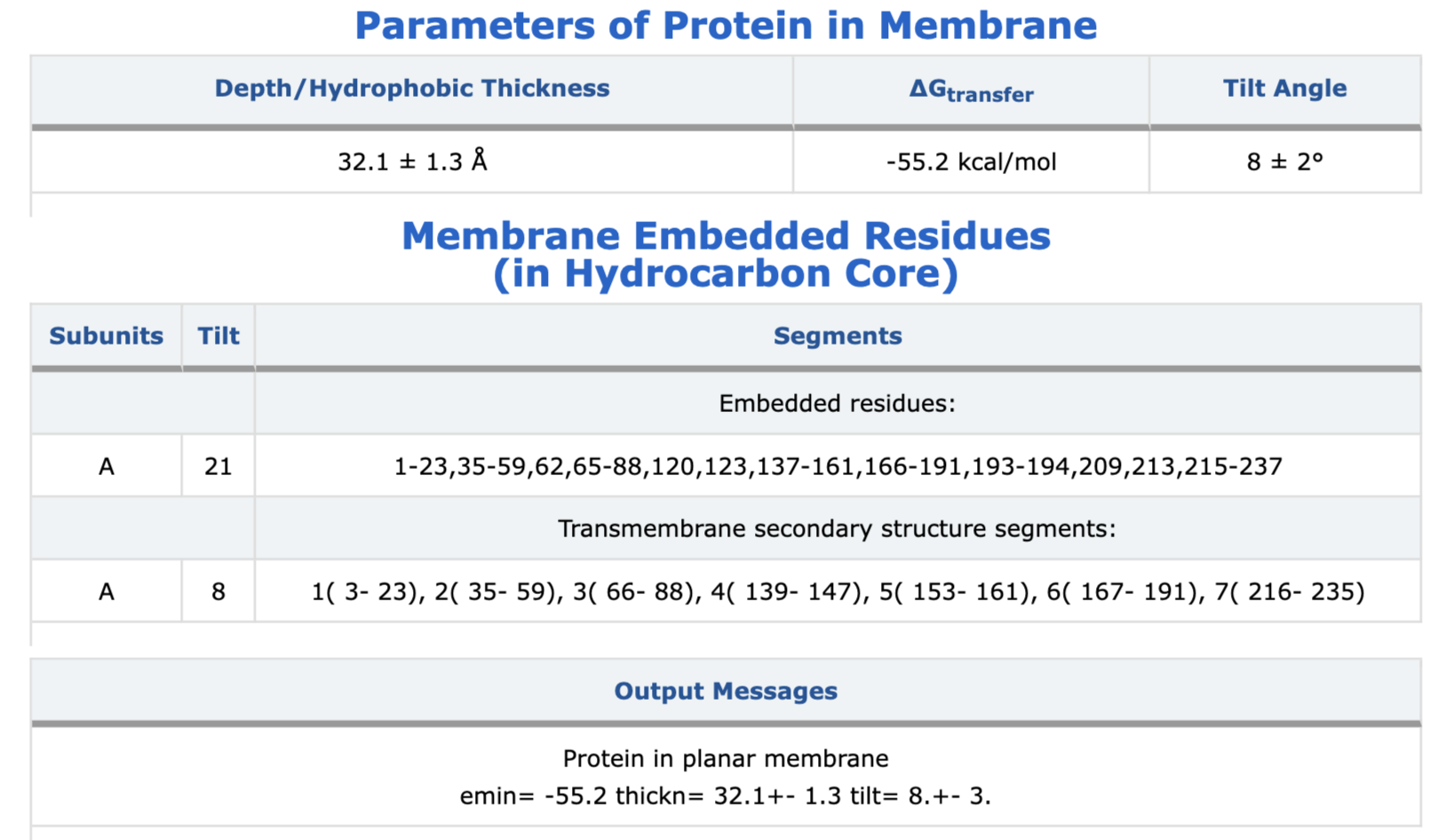
Для запуска алгоритма PPM использовались параметры, приведённые на Fig 3. Для запуска PPM было использовано предсказание AlphaFold, скачанное с UniProt с раздела "Structure". Параметр "Topology (N-ter)" был выставлен в соответствие с выдачей DeepTMHMM, который показал, что N-концевой домен располагается внутри.
DeepTMHMM предсказал 6 трансмембранных альфа-спиралей, а PPM - 7. Можно заметить, что DeepTMHMM слил 5 и 6 альфа спирали в выдаче PPM. Это можно попробовать объяснить сложным строением этого учатска или поргешностью работы алгоритмов.