назад
Знакомство с терминологией GO:
Описание функции белка с помощью аннотации Gene Ontology:
Описание функции белка PURT_Ecoli в соответствии с GO-аннотацией:
| |
Онтология GO (название словаря) |
Количество разных ассоциированных терминов GO |
Функция белка
(краткое описание, близкое к тексту определения терминов GO) |
| Где |
Cellular Component |
0 |
|
| Зачем, для чего |
Biological Process |
2 |
GO:0009152: процесс биосинтеза пуриновых рибонуклеотидов (химические реакции, в результате которых
получается пуриновый рибонуклеотид, состоящий из рибонуклеозида с пуриновым основанием (аденином или гуанином), эстерифицированного фосфатом на 3' или 5'-гидроксиле)
GO:0006164: процесс биосинтеза пуриновых нуклеотидов (аналогично GO:0009152, в качестве сахара выступает не только рибоза, но и дезоксирибоза)
|
| Молекулярный механизм |
Molecular Function |
4 |
GO:0016740: трансферазная активность (перенос функциональных групп или молекулярных остатков от одного соединения (донора) к другому (акцептору); трансферазы относятся ко 2-ому классу ферментов (EC) )
GO:0003824: каталитическая активность (катализ биохимических реакций при физиологических температурах; ферменты специфически связываются с субстратом для осуществления катализа реакции; ферменты чаще всего - белки, однако существуют РНК с каталитической активностью (рибозимы))
GO:0043815: фосфорибозилглицинамидформилтрансверазная 2 активность (фермент катализирует реакцию: муравьиная кислота + АТФ + 5'-фосфорибозилглицинамид = 5'-фосфорибозил-N-формилглицинамид + АДФ + дифосфат)
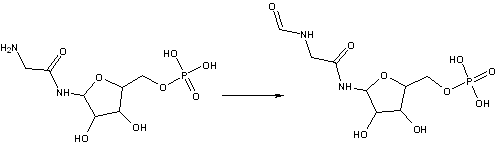
GO:0016742: гидроксиметил-, формил- зависимая трансферазная активность
|
| Специфичность |
Molecular Function |
4 |
GO:0005524: связывание АТФ (селективное, нековалентное взаимодействие с АТФ)
GO:0000287: связывание иона магния (селективное, нековалентное взаимодействие с катионом магния)
|
Описание термина GO:
На сайте консорциума Gene Ontology проведем поиск описаний для 3 терминов GO (т.к. не нашлось ни одного термина из словаря Cellular Component, возьмем 2 термина из Molecular Function и 1 - из Biological Process).
Описание терминов GO
| GO ID выбранного термина |
Список синонимов |
Список ближайших родительских терминов GO с указанием типа связи |
Список ближайших дочерних терминов GO с указанием типа связи |
| GO:0009152 (словарь Biological Process) |
exact: purine ribonucleotide anabolism
exact: purine ribonucleotide biosynthesis
exact: purine ribonucleotide formation
exact: purine ribonucleotide synthesis |
GO:0006164 - биосинтез пуриновых нуклеотидов (связь GO:0009152 is a GO:0006164, т.е. GO:0009152 - частный случай GO:0006164), GO:0006164 ассоциирован с нашим белком PURT_ECOLI
GO:0009150 - метаболизм пуриновых рибонуклеотидов (связь GO:0009152 is a GO:0009150)
GO:0009260 - биосинтез рибонуклеотидов (связь GO:0009152 is a GO:009260)
Картинка графа |
GO:0009180 - боисинтез пуриновых рибонуклеозид-дифосфатов
GO:0009206 - боисинтез пуриновых рибонуклеозид-трифосфатов
GO:0009168 - боисинтез пуриновых рибонуклеозид-монофосфатов
дочерние термины можно увидеть на графе |
| GO:0043815 (словарь Molecular Function) |
exact: 5'-phosphoribosylglycinamide transformylase 2
exact: formate-dependent GAR transformylase activity
есть также связанные синонимы (неточные):
related: GAR transformylase 2
related: GART 2
|
GO:0016742 - гидроксиметил-, формил- заваисимая трансферазная активность (GO:0043815 is a GO:0016742), GO:0016742 представлен среди терминов GO, ассоциированных с белком PURT_ECOLI
Граф |
Дочерних терминов нет, GO:0043815 - младший термин GO, нет таких термнов GO, являющихся частным случаем GO:0043815. |
| GO:0000287 (словарь Molecular Function) |
exact: magnesium binding |
GO:0046872 - связывание иона металла (связь GO:0000287 is a GO:0046872), этот термин также представлен серди ассоциированных с белком PURT_ECOLI
Граф |
Дочерних терминов нет, GO:0000287 - младший термин GO, нет таких термнов GO, являющихся частным случаем GO:0000287. |
Оценка качества функциональной аннотации белков в UniProt:
Определение числа реальных и гипотетических белков из таксона Oryza (рис, rice):
Ранг таксона: род (genus)
Идентификатор таксона (NCBI_ID): 4527
Классификация:
Царство: Растения
Отдел: покрытосеменные
Класс: Однодольные
Порядок: Злакоцветные
Семейство: Злаки
Подсемейство: Ehrhartoideae
Род: Рис
В SRS поле ProteinExistence базы данных UniProt проиндексировано следующим образом:
1: evidence at protein level - существование белка доказано экспериментально (86150 записей, 0,774%)
2: evidence at transcript level - известны только соответствующие транскрипты (526547 записей, 4,729%)
3: inferred from homology - белок предсказан по гомологии (2505249 записей, 22,499%)
4: predicted - иные предсказанные белки (8014981 записей, 71,984%)
5: uncertain - сомнительные белки (1541 записей, 0,014%)
Выясним соотношение между реальными и гипотетическими белками из таксона Oryza (по данным UniProt):
| |
Количество в UniProt |
| Существование белка доказано экспериментально |
385 |
| Известны только соответствующие транскрипты |
16065 |
| Гипотетический белок, предсказан по гомологии |
9596 |
| Иные предсказанные гипотетические белки |
120394 |
Из результатов поиска по UniProt видно, что большинство белков риса - предсказанные, а количество реально полученных белков - невелико.
Возможно, если провести поиск по SwissProt, доля выделенных белков и транскриптов будет больше, ведь SwissProt - курируемая база данных (SwissProt - часть UniProt).
| |
Количество в SwissProt |
| Существование белка доказано экспериментально |
275 |
| Известны только соответствующие транскрипты |
2001 |
| Гипотетический белок, предсказан по гомологии |
724 |
| Иные предсказанные гипотетические белки |
13 |
И действительно, число предсказанных белков значительно уменьшилось, а большинству записей соответствуют транскрипты.
Определение качества функциональной аннотации в SwissProt:
Коды экспериментального доказательства функции ( Guide to GO Evidence Codes ):
Inferred from Experiment (EXP) - из эксперимента
Inferred from Direct Assay (IDA) - из прямого анализа
Inferred from Physical Interaction (IPI) - из физического взаимодействия
Inferred from Mutant Phenotype (IMP) - из мутантного фенотипа
Inferred from Genetic Interaction (IGI) - из генетического взаимодействия
Inferred from Expression Pattern (IEP) - из модели экспрессии
Определим с помощью одного запроса к SRS, сколько из реальных белков (выделены белки или транскрипты) таксона Oryza аннотированы по всем трем словарям GO, и у которых встречается хотя бы один раз один из кодов экспериментального доказательства функции.
Для этого обратимся к SRS с запросом: (([swissprot-Taxonomy:Oryza*] & ([swissprot-ProteinExistence:1:*] | [swissprot-ProteinExistence:2:*])) & (((([swissprot-DBxref_:GO:*] & [swissprot-DBxref_:P:*]) & [swissprot-DBxref_:F:*]) & [swissprot-DBxref_:C:*]) & ((((([swissprot-DBxref_:EXP:*] | [swissprot-DBxref_:IDA:*]) | [swissprot-DBxref_:IPI:*]) | [swissprot-DBxref_:IMP:*]) | [swissprot-DBxref_:IGI:*]) | [swissprot-DBxref_:IEP:*])))
По запросу найдено 56 белков ( файл с находками). Всего реальных белков Oryza в SwissProt - 2276 (из предыдущего задания),
найденные белки составляют 2,5%. Такой результат говорит о низком количественном уровне аннотации в SwissProt, однако я думал, что будет еще меньше.
Обращение с аналогичным запросом к UniProt трудно осуществимо, из-за большего размера БД UniProt по сравнению с SwissProt.
С помощью запроса SRS к SwissProt были получены 56 хорошо аннотированных белков из организмов рода Oryza. Подобная аннотация требует много времени, сил и средств, было бы интересно узнать белки с какими функциями были аннотированы.
(К тому же Oryza - рис - значимая сельскохозяйственная культура, все белки имеют идентификаторы *_ORYSJ - соответствуют рису посевному, интересно на какие белки прежде всего было обращено внимание исследователей)
Для этого обработаем полученный список белков с помощью программы GOstat (хотя выборка и не является большой) (см. ниже Использование GO для работы с массовыми данными).
Чаще других представлены термины GO:
GO:0009987 (P-value=5.57e-32, встречается 27 раз) - процессы протекающие на клеточном уровне, но значимые не для отдельно взятых клеток (например клеточное общение)
GO:0007275 (P-value=4.68e-20, встречается 10 раз) - биологические процессы, связанные с ростом многоклеточного организма
GO:0044237 (P-value=6.45e-20, встречается 19 раз) - клеточные метаболические процессы
GO:0043170 (P-value=1.92e-18, встречается 17 раз) - метаболизм макромолекул
GO:0050896 (P-value=1.92e-18, встречается 12 раз) - изменение клеточной активности в ответ на стимул
GO:0009791 (P-value=3.32e-12, встречается 6 раз) - постэмбриональное развитие
Таким образом среди терминов GO аннотированных белков часто встречаются термины посвященные росту и развитию организма, реакции клеток на внешние стимулы, что значимо для изучения риса как сельскохозяйственной культуры.
Использование GO для работы с массовыми данными:
Получение выборки последовательностей белков с заданной функцией:
Проведем поиск всех белков, выполняющих функции карбоксилирования, из организмов, принадлежащих к роду Oryza.
Для этого найдем на сайте консорциума Gene Ontology идентификатор GO, соответствующий этой функции.
Карбоксилирование (carboxylation) - введение в молекулу карбоксильной группы -COOH. По запросу carboxylation на сайте Gene Ontology найдено 10 идентификаторов GO, среди которых 4 относятся к декарбоксилированию, а остальные посвящены карбоксилированию аминокислот белков, наиболее общим среди них является GO:0018214 .
На мой взгляд, такой результат поиска очень странный, ведь карбоксилирование происходит не только при посттрансляционных модификациях белка, но у растений главным образом в цикле Кальвина, когда фермент рибулозобисфосфаткарбоксилаза
присоединяет углекислый газ к рибулозо-1,5-бифосфату.
Идентификатор GO:0018214 относится к словарю biological process.
По запросу к SRS, где в поле Taxonomy - Oryza, а в поле DBxref_ - GO:0018214 ничего не нашлось ни в базе данных UniProt, ни даже в TrEMBL.
В то время, как для идентификатора GO:0016984, означающего рибулозобисфосфаткарбоксилазную активность, найдено 49 записей (по таксону Oryza в базе данных UniProt).
Запрос: ([uniprot-Taxonomy:Oryza*] & [uniprot-DBxref_:GO:0016984*]). Файл с последовательностями находок в fasta-формате.
Определение главной функции в большом списке белков:
В файле - список белков (174 белка), полученный в результате массового эксперимента (в т.ч. и белок PURT_ECOLI). Определим белки с какими функциями доминируют в этом списке, используя программу GOstat .
В выборке чаще других представлены термины GO:
GO:0005515 (P-Value=1.74e-50, встречается 48 раз) - селективное нековалентное взаимодействие (связывание) с белками и белковыми комплексами (словарь - molecular function)
GO:0005886 (P-Value=8.65e-49, встречается 55 раз) - мембрана, отделяющая клетку от внешней среды (словарь - cellular component)
GO:0044464 (P-value=1.26e-08, встречается 111 раз) - любой элемент клетки, базовая структурная и функциональная единица организма (словарь - cellular component)
GO:0006164 (P-value=3.48e-15, встречается 22 раза) - процесс биосинтеза пуриновых нуклеотидов (словарь - biological process). Этот термин ассоциирован с белком PURT_ECOLI (P33221).
GO:0006163 (P-value=4.69e-15, встречается 22 раза) - пуриновый метаболизм (словарь - biological process)
GO:0009152 (P-value=2.55e-14, встречается 20 раз) - биосинтез пуриновых рибонуклеотидов (словарь - biological process). Этот термин ассоциирован с белком PURT_ECOLI (P33221).
GO:0022857 (P-value=4.95e-06, встречается 29 раз) - участвует в транспорте веществ с одной стороны мембраны на другую (словарь - molecular function)
GO:0009259 (P-value=7.89e-14, встречается 20 раз) - метаболизм рибонуклеотидов (словарь - biological process)
В выборке доминируют белки, вовлеченные в метаболизм пуриновых рибонуклеотидов, что согласуется с функцией белка PURT_ECOLI. Возможно, также есть белки, обеспечивающие транспорт необходимых веществ через клеточную мембрану.
В выдаче GOstat первые 2 находки (GO:0005515 и GO:0005886) имеют очень низкие значения P-value (1.74e-50 и 8.65e-49 соответственно) (вероятность того, что они попали в выдачу случайным образом мала), далее P-value довольно резко увеличивается (для GO:0006164 P-value=3.48e-15), но все же еще остается достаточно низким, чтобы считать полученные термины GO достоверными.
