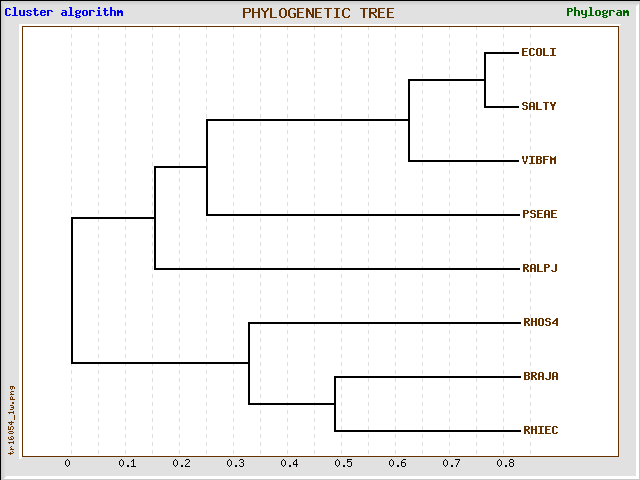
Реконструкции филогенетического дерева бактерий:
| Название | Мнемоника |
| Bradyrhizobium japonicum | BRAJA |
| Rhizobium etli | RHIEC |
| Ralstonia pickettii | RALPJ |
| Escherichia coli | ECOLI |
| Salmonella typhimurium | SALTY |
| Rhodobacter sphaeroides | RHOS4 |
| Pseudomonas aeruginosa | PSEAE |
| Vibrio fischeri | VIBFM |
Для реконструкции использовались последовательности белков семейства омега-субъеденц ДНК-зависимой РНК-полимеразы, файл с последовательностями. Множественное выравнивание программы muscle.
В результате получено правильное дерево (см. филогенетическое дерево ):

Матрица расстояний:
1 2 3 4 5 6 7 8
1 ECOLI 0.000 0.063 0.205 0.716 0.573 0.753 0.803 0.825
2 SALTY 0.063 0.000 0.205 0.716 0.573 0.753 0.803 0.825
3 VIBFM 0.205 0.205 0.000 0.722 0.587 0.749 0.811 0.832
4 RALPJ 0.716 0.716 0.722 0.000 0.637 0.848 0.905 0.925
5 PSEAE 0.573 0.573 0.587 0.637 0.000 0.745 0.802 0.837
6 RHOS4 0.753 0.753 0.749 0.848 0.745 0.000 0.487 0.521
7 BRAJA 0.803 0.803 0.811 0.905 0.802 0.487 0.000 0.345
8 RHIEC 0.825 0.825 0.832 0.925 0.837 0.521 0.345 0.000
Матрица расстояний отличается от матрицы , выданной программой fprotdist.
Однако как и в матрице fprotdist, расстояние от ECOLI до любой другой бактерии, кроме SALTY, равно расстоянию от SALTY до той же бактерии, расстояние же от ECOLI до SALTY - минимальное среди всех остальных расстояний. Поэтому также выполняется свойство ультраметричности для троек бактерий, включающих ECOLI и SALTY.
Программа TreeTop построила правильное филогенетическое дерево, даже на основании выравнивания по одному белку, что не удалось сделать программе fprotpars и fneighbor.