Выравнивание последовательностей белков
Глобальное парное выравнивание гомологичных белков
Примечание: Фосфофруктокиназа у Bacillus subtilis имеет рекомендуемое название "ATP-dependent 6-phosphofructokinase". Изоформа фосфофруктокиназы в названии не указана, так как для неё у Bacillus subtilis нет изоферментов.
| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels |
|---|---|---|---|---|---|---|---|
| ATP-dependent 6-phosphofructokinase isozyme 1 | PFKA_ECOLI | PFKA_BACSU | 926.0 | 55.8% | 74.1% | 3 | 3 |
| Glucose-6-phosphate isomerase | G6PI_ECOLI | G6PI_BACSU | 220.0 | 20.3% | 33.1% | 205 | 25 |
| Glucose-6-phosphate 1-dehydrogenase | G6PD_ECOLI | G6PD_BACSU | 931.5 | 39.4% | 59.8% | 26 | 9 |
Локальное парное выравнивание гомологичных белков
| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | Coverage 1 | Coverage 2 |
|---|---|---|---|---|---|---|---|---|---|
| ATP-dependent 6-phosphofructokinase isozyme 1 | PFKA_ECOLI | PFKA_BACSU | 928.0 | 56.3% | 74.8% | 1 | 1 | 99.4% | 99.4% |
| Glucose-6-phosphate isomerase | G6PI_ECOLI | G6PI_BACSU | 230.0 | 23.5% | 40.5% | 79 | 15 | 73.0% | 82.2% |
| Glucose-6-phosphate 1-dehydrogenase | G6PD_ECOLI | G6PD_BACSU | 935.5 | 39.7% | 60.3% | 25 | 8 | 99.2% | 99.4% |
Парное выравнивание негомологичных белков
| Protein Name 1 | Protein Name 2 | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | Coverage 1 | Coverage 2 |
|---|---|---|---|---|---|---|---|---|---|---|
| Глобальное выранивание | ||||||||||
| Pyruvate dehydrogenase E1 component | Transketolase | ODP1_ECOLI | TKT_BACSU | 172.0 | 17.0% | 29.4% | 330 | 37 | ||
| Локальное выранивание | ||||||||||
| Pyruvate dehydrogenase E1 component | Transketolase | ODP1_ECOLI | TKT_BACSU | 181.5 | 19.0% | 32.7% | 224 | 32 | 87.6% | 99.1% |
Как и ожидалось, у выравниваний негомологичных последовательностей Score, Identity и Similarity оказались ниже, чем для гомологичных последовательностей. Однако эти показатели близки к данным выравнивания гомологичных глюкозо-6-фосфат изомераз. Помимо случайного совпадения, близкое к 20% Identity у негомологичных белков можно объяснить тем, что оба фермента используют один и тот же кофермент (тиаминпирофосфат) и катализируют реакции с одинаковым механизмом[1] и, следовательно, могут иметь в своём составе схожие домены.
Множественное выравнивание гомологичных белков
Для множественного выравнивания была выбрана глюкозо-6-фосфат изомераза штамма K12 кишечной палочки (мнемоника функции белка: G6PI; рекомендуемое полное имя белка из E. coli: Glucose-6-phosphate isomerase), так как выравнивание её последовательности с гомологичным ферментом Bacillus subtilis показало для них низкую схожесть (и в локальном, и в глобальном выравниваниях Identity равна своему нижнему пределу для гомологичных белков: 20-25%). С помощью множественного выравнивания предполагалось проверить гипотезу о том, что низкая схожесть обусловлена быстрой эволюцией исследуемого фермента. Для выравнивания был предварительно получен список ID всех 584 белков с такой же мнемоникой функции в Swiss-Prot. Из них были выбраны белки родственных E.coli Haemophilus influenzae и Agrobacterium fabrum, родственной B. subtilis Clostridium acetobutylicum и менее родственных обоим бактерям Nostoc punctiforme и Mycobacterium tuberculosis, чтобы по полученному из выранивания филогенетическому дереву можно было оценить скорость эволюции белка. Выравнивание было получено путём создания файла с USA выбранных белков, создания файла с их последовательностями программой seqret и запуска на kodomo программы muscle для этого файла. По выравниванию видно, что белки E.coli, H. influenzae, A. fabrum и M. tuberculosis более похожи друг на друга, чем на другие три белка: примерно в половине столбцов 3-4 остатка из 4 одинаковы. Для всех семи белков выделяется только несколько крупных консервативных участков (по номерам столбцов): 158-174, 205-214, 280-298, 372-380, 511-518 и 533-542. Так как одинаковые остатки распределены не равномерно, а участками, то скорее всего все 7 белков гомологичны. По результатам дополнительного выранивания программой Clustal Omega было построено филогенетическое дерево выровненных последовательностей (Рис. 1). На нём выделяются группы Фирмикут (B. subtilis и C. acetobutylicum) и Гамма-протеобактерий (E.coli и H. influenzae), но не выделяется более крупный тип Протеобактерий (E.coli, H. influenzae и A. fabrum). Это означает, что глюкозо-6-фосфат изомераза эволюционирует настолько быстро, что между родство дальними видами по выраниванию её последовательностей установить невозможно, поэтому в парных выраниваниях Identity для неё низкий.
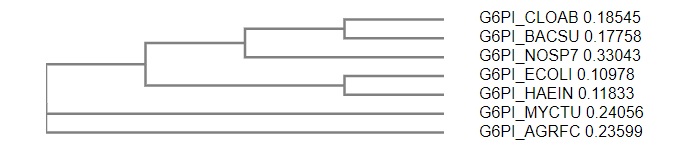
Рисунок 1. Филогенетическое дерево, построенное по выраниванию семи последовательностей глюкозо-6-фосфат изомеразы.
Выравнивание TrmB Thermococcus litoralis с его гомологом
| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | |||
|---|---|---|---|---|---|---|---|---|---|---|
| Глобальное выранивание | ||||||||||
| HTH-type sugar sensing transcriptional regulator TrmB | TRMBR_THELN | TRMBR_PYRFU | 1722.0 | 100.0% | 100.0% | 0 | 0 | |||
Выравнивание последовательностей репрессора TrmB археи Thermococcus litoralis и близкородственного вида Pyrococcus furiosus помимо гомологии показало полную их идентичность. Это подтверждает факт недавнего горизонтального переноса гена этого белка между этими археями [2].
Выравнивание при различных параметрах программ needle и water
| ID 1 | ID 2 | Gap opening penalty | Gap extension penalty | Score | % Identity | % Similarity | Gaps | Indels | Coverage 1 | Coverage 2 |
|---|---|---|---|---|---|---|---|---|---|---|
| Глобальное выранивание | ||||||||||
| PFKA_ECOLI | PFKA_BACSU | 10.0 | 2.0 | 926.0 | 55.8% | 74.1% | 3 | 3 | ||
| PFKA_ECOLI | PFKA_BACSU | 10.0 | 0.5 | 926.0 | 55.8% | 74.1% | 3 | 3 | ||
| PFKA_ECOLI | PFKA_BACSU | 1.0 | 2.0 | 981.0 | 54.4% | 72.1% | 41 | 39 | ||
| PFKA_ECOLI | PFKA_BACSU | 1.0 | 0.5 | 1002.5 | 53.4% | 70.1% | 69 | 40 | ||
| Локальное выранивание | ||||||||||
| PFKA_ECOLI | PFKA_BACSU | 10.0 | 2.0 | 928.0 | 56.3% | 74.8% | 1 | 1 | 99.4% | 99.4% |
| PFKA_ECOLI | PFKA_BACSU | 10.0 | 0.5 | 928.0 | 56.3% | 74.8% | 1 | 1 | 99.4% | 99.4% |
| PFKA_ECOLI | PFKA_BACSU | 1.0 | 2.0 | 992.0 | 54.0% | 70.9% | 64 | 43 | 99.7% | 99.4% |
| PFKA_ECOLI | PFKA_BACSU | 1.0 | 0.5 | 1004.5 | 53.8% | 70.7% | 66 | 39 | 99.7% | 99.4% |
Без опции -auto программы needle и water запрашивают значения двух параметров: "Gap opening penalty" и "Gap extension penalty" – соответственно штраф за первый гэп инделя и штраф за каждый последующий гэп инделя (по умолчанию равны 10 и 0.5 соответственно). Снижение штрафа за открытие увеличивает количество гэпов и инделей в выравнивании повышает вес выранивания. Покрытие в локальном выравнивании при этом увеличивается. Снижение штрафа за удлинение дополнительно увеличивает число гэпов.
Вес выравниваний с низкими штрафами выше, так как при возможности внесения большого числа гэпов негомологичные участки можно выровнять с высоким Identity. Например, в выравниваниях с наименьшими штрафами много гэпов было внесено в концевые части последовательностей, и там появились дополнительные идентичные остатки, хотя по выравниванию с наибольшими штрафами явная гомология в концевых участках не прослеживается.
Список литературы
- Д.Нельсон, М.Кокс. Основы биохимии Ленинджера: в 3 т. Т.1: Основы биохимии, строение и катализ. Пер. с англ. – 3-е изд, испр. – М.: Лаборатория знаний, 2017. – 694 с. ISBN: 978-5-00101-014-2
- Lee, S.-J., Surma, M., Hausner, W., Thomm, M., & Boos, W. (2008). The role of TrmB and TrmB-like transcriptional regulators for sugar transport and metabolism in the hyperthermophilic archaeon Pyrococcus furiosus. Archives of Microbiology, 190(3), 247256. DOI: 10.1007/s00203-008-0378-2.