Сборка de novo
Процесс сборки
Сначала были скачаны чтения SRR4240380, на основе которых предстояло составить сборку:
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/000/SRR4240380/SRR4240380.fastq.gz
Далее они были проанализированы с помощью fastqc:
fastqc SRR4240380.fastq.gz &> log/fastqc_SRR4240380.log
Был получен следующий отчёт: SRR4240380_fastqc.html. В чтениях fastqc нашёл три адаптерных последовательности. Они были скопированы в файл adapters.fa в формате fasta. Перепредставленный PolyA, также найденный fastqc, в файл с адаптерами включён не был. Далее в этот файл были загружены адаптерные последовательности из папки /mnt/scratch/NGS/adapters (на случай, если fastqc какие-то адаптеры не обнаружил):
cat /mnt/scratch/NGS/adapters/* >> adapters.fa
Далее чтения были триммированы:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240380.fastq.gz trimmed_SRR4240380.fastq.gz ILLUMINACLIP:adapters.fa:2:7:7 TRAILING:20 MINLEN:32 &> log/trimmomatic.log
# Одновременно удаляет адаптеры, обрезает чтения справа и фильтрует их по длине.
Как и для остальных программ, stdin и sderr программы trimmomatic были перенаправлены в log-файл: trimmomatic.log. Из него следует, что из 5217318 исходных чтений были удалены 352294 (6.75%). Триммированные чтения тоже были проанализированы с помощью fastqc:
fastqc trimmed_SRR4240380.fastq.gz &> log/fastqc_trimmed_SRR4240380.log
По полученному отчёту trimmed_SRR4240380_fastqc.html видно, что все адаптеры были удалены (fastqc их не находит). Из него следует, что из 5217318 исходных чтений были удалены 352294 (6.75%). Далее был составлен список содержащихся в чтениях k-меров:
velveth kmers 31 -fastq.gz trimmed_SRR4240380.fastq.gz -short &> log/velveth.log
На их основе была составлена сборка:
velvetg kmers &> log/velvetg.log
В log-файле velvetg.log был найден N50, равный 12042 bp. Также был получен файл contigs.fa с последовательностями собранных контигов и файл stats.txt со статистикой по ним. Всего собралось 402 контига. Помимо 162 (кол-во контигов с длиной больше 32 bp) информативных контигов с длиной порядка 100-10000 bp 340 было получено много мелких контигов с длиной вплоть до 31 bp (в файле stats.txt их длина отмечена как 1 – это означает, что длина контига равна длине в stats.txt плюс длина k-мера минус 1, то есть 31).
Среднее покрытие оказалось равным 9.43, максимальное – 130.48, минимальное – 2.42. Контиг с наибольшим покрытием – contig56, контиг с наименьшим покрытием – contig235. Каких-либо особенностей в их последовательности визуально не видно, их выравнивания на геном бактерии с помощью blast тоже принципиально не отличаются от приведённых ниже.
Анализ
Далее были рассмотрены 3 самых длинных контига (Табл.1). Для каждого из них было проведено выравнивание на геном Buchnera aphidicola (GenBank AC – CP009253.1) с помощью megablast (с модификацией blast2seq).
| Номер контига | Длина контига, bp | Покрытие |
|---|---|---|
| 3 | 25915 | 27.42 |
| 20 | 23850 | 24.76 |
| 23 | 23807 | 25.73 |
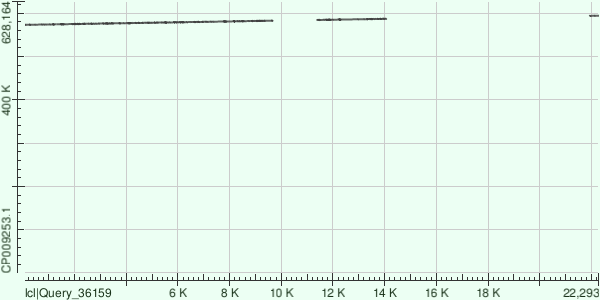
Самый длинный контиг – contig3 – имеет длину 25915 bp. Он выравнивается на 2004 – 11103, 613658 – 620926 и 621055 – 627104 координаты генома Buchnera aphidicola в прямой ориентации с 5019 однонуклеотидными заменами и 682 гэпами. Между указанными тремя участками генома находятся неконсервативные участки, на которых последовательности генома и контига сильно различаются. То же самое касается следующих двух контигов: они непрерывны, а между участками, на которых контиг выровнялся с геномом, находятся неконсервативные участки. Ссылка на выдачу blast: 3.txt.
Несмотря на то, что часть контига выранивается на конец хромосомы, а часть – на начало, контиг ложится на хромосому непрерывно, так как она кольцевая, и её конец переходит в начало, а контиг проходит через точку, принятую за начало хромосомы.
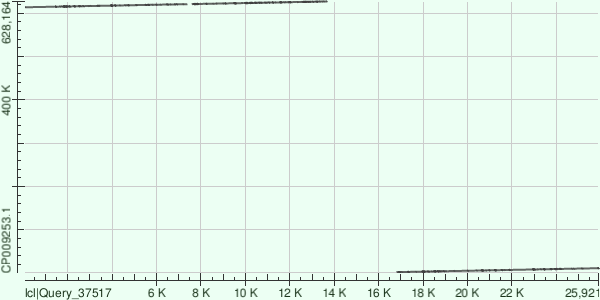
Второй по длине контиг – contig20 – имеет длину 23850 bp. Он выравнивается на геном Buchnera aphidicola с инверсией. Контиг выравнивается на 236918 – 247596, 232358 – 236859, 229411 – 232057 и 248967 – 252161 координаты генома с 5065 однонуклеотидными заменами и 686 гэпами. Ссылка на выдачу blast: 20.txt
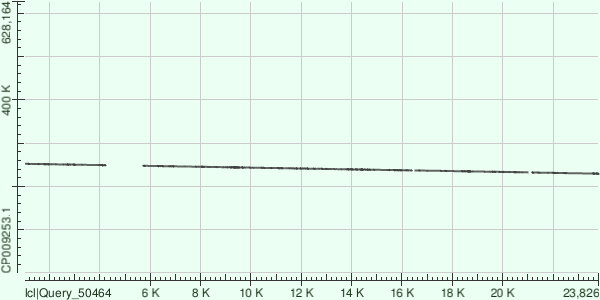
Третий по длине контиг – contig23 – имеет длину 23807 bp. Он выравнивается на 573092 – 582686, 584329 – 587055 и 593743 – 594099 координаты генома Buchnera aphidicola в прямой ориентации с 3357 однонуклеотидными заменами и 573 гэпами. Ссылка на выдачу blast: 23.txt