Сигналы и мотивы – 2
Особенности транскрипции архей
Поиск бактериальных промоторов – интересная задача, но в прошлых семестрах я изучал архею, термофила Thermococcus litoralis. Было решено найти сигнал инициации транскрипции в его промоторе. Особенность задачи состоит в том, что у архей РНК-полимераза похожа на эукариотическую РНК-полимеразу 2, соответственно в промоторе ожидается найти TATA-box. Из сходств с бактериями остаётся оперонная организация.
Извлечение последовательностей для обучения
Задача заключалась в поиске 30 участков длиной 150 bp, находящихся непосредственно перед началом оперона. Сначала с помощью сервиса operon-mapper были предсказаны опероны T. litoralis (на вход подавалась хромосома T. litoralis, добытая в NCBI). В качестве выборки для обучения было отобрано 30 оперонов c "+"-цепи, содержащих аннотированные гены и достаточно удалённых от конца предыдущих (в этом случае можно верить, что выбранные опероны существуют в реальности). Далее были получены последовательности с -150 по -1 позицию относительно начала каждого оперона следующим скриптом: cdk_operon.py. Они были поданы в веб-версию MEME в качестве входных данных. MEME был запущен со следующими параметрами: поиск 3 мотивов, встречающихся 0 или 1 раз в каждой последовательности, длина мотива от 6 до 10 нуклеотидов, поиск по обеим цепям (так как нельзя быть уверенным, что у архей нет сигналов транскрипции на "-"-цепи).
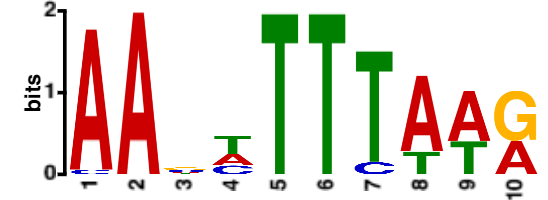
Один из найденных MEME мотивов (Рис. 1) был обнаружен во всех 30 последовательностях и оказался достаточно правдоподобным – с e-value = 9.9e-005. Найденный мотив имеет длину 10 bp (AANNTTTWWR) и не похож на TATA-box (TATAAA). Большой разброс в локализации (Рис. 2, в среднем на -60 позиции) мотива можно объяснить неточной аннотацией CDK (не тот ATG), а появление на "-"-цепи – тем, что согласно выдаче operon-mapper опероны на обеих цепях у T. litoralis располагаются в большинстве случаев практически вплотную друг к другу (в среднем несколько десятков нуклеотидов), и их промоторы могут перекрываться. Другие два найденные мотива недостаточно правдоподобны, имеют высокий e-value > 0.01, поэтому здесь не рассматриваются. Полная выдача MEME.

Рисунок 2. Выдача MEME.
Позиционная весовая матрица для найденного сигнала была получена по 30 отобранным выше последовательностям аналогично прошлому практикуму (с изменением GC-состава, определённого во втором семестре): pwm.csv.
Проверка PWM с помощью FIMO
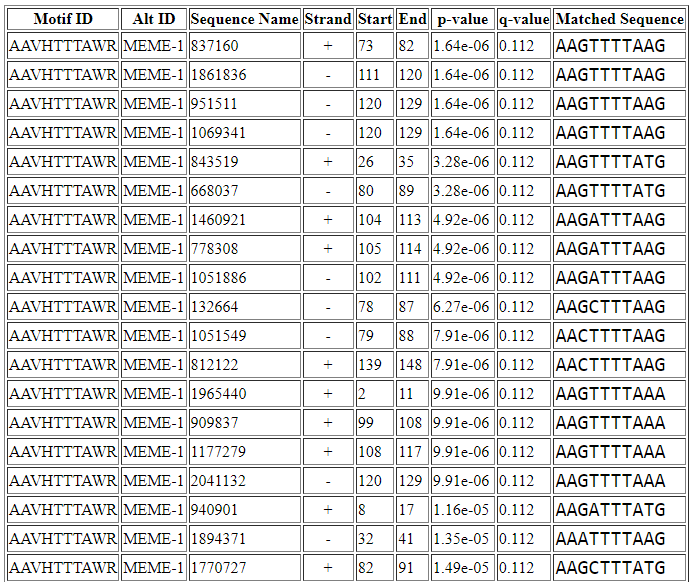
В качестве тестовых данных были взяты все остальные upstream-участки оперонов, кроме тех, которые были использованы для обучения: test_operon.py. Они были поданы на вход веб-версии FIMO вместе c PWM соответствующим файлом входными параметрами запуска: алфавит – ACGT, длина мотива до 10 нуклеотидов, порог p-value – 0.01.
FIMO выдал 8239 для уровня значимости 0.1, то есть около 10 мотивов на последовательность (обе цепи). Можно считать, что найденный мотив действительно является сигналом транскрипции. После обработке выдачи в Excel выяснилась ещё одна важная вещь: дисперсии координат мотива на "+"- и "-"-цепях приблизительно равны – это означает, что данный мотив может одинаково активировать транскрипцию, находясь на разных цепях (либо что соседние опероны на различных цепях всегда расположены на фиксированном расстоянии друг относительно друга). Средние значения координат мотива на разных цепях всё же различаются: (-64)-(-55) для "+"-цепи и (-71)-(-62) для "-"-цепи (свидетельствует в пользу второй гипотезы). Полная выдача FIMO.