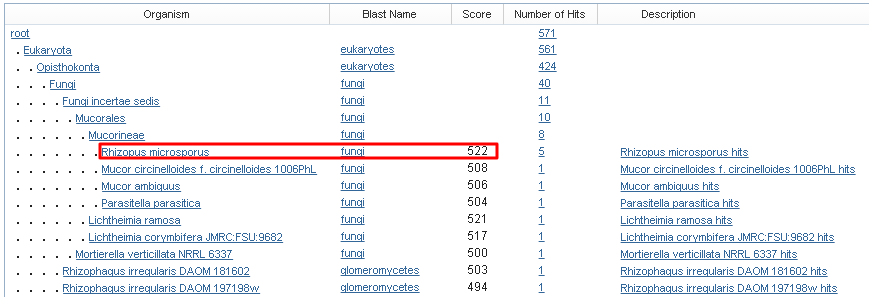
Рис.1. Таксономический отчет по результатам blastx для 318 скэффолда X5.
Cтраница со статусом задания AUGUSTUS.На этой же странице приведена команда для распаковки архива с результатами: tar -xzvf *.tar.gzПосле ее выполнения появляется папка augustus, в которой содержатся 6 файлов:
augustus.aa - аминокислотные последовательности предсказанных генов;
augustus.cdsexons - нуклеотидные последовательности(ДНК) предсказанных экзонов;
augustus.codingseq - кодирующие нуклеотидные последовательности(ДНК);
augustus.gbrowse - предсказанные гены в формате для геномного браузера GBrowse;
augustus.gff - таблица с нуклеотидными и аминокислотными последовательностями и координатами различных участков;
augustus.gtf - таблица с координатами различных участков контига;
*augustus.mrna - нуклеотидные последовательности(ДНК) предсказанных мРНК(в моем контиге их не было).
В 318 контиге AUGUSTUS предсказал 20 белков(Более, чем достаточно. Можно было взять контиг поменьше.) Поиск в blastp генов 5 и 20 не дал ни одной последовательности.
g2
Белок с наибольшим e-value(7e-107) на всем контиге. Но процент идентичности всего лишь 49%. Blastp определил, что он пренадлежит к суперсемье белков WD40, которые добавляют WD-повторы, состоящие из 40 аминокислот и заканчивающиеся триптофаном и аспаргиновой кислотой.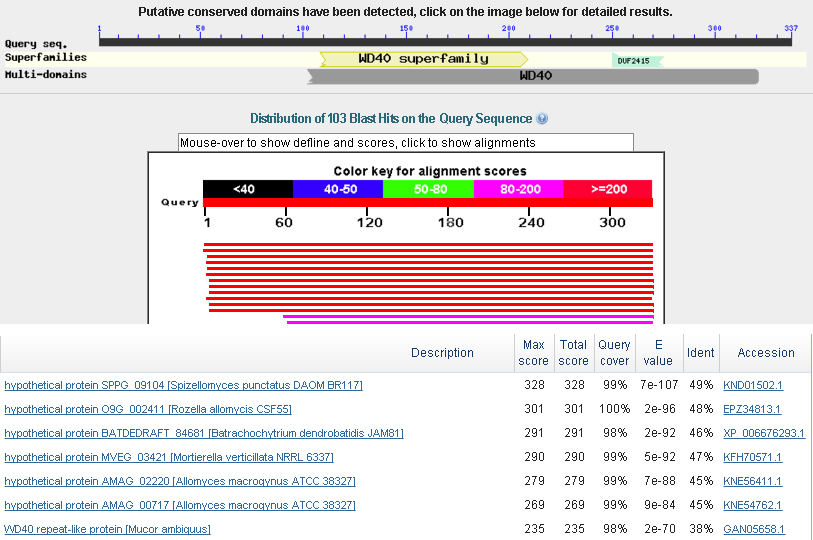
Рис.2. Результаты проверки g2 в blastp.
g13
Очень похожая на предыдущую ситуация. Только чуть повыше e-value - 2e-95. Гликозилтрансферазы переносят остатки моносахаридов от углевода-донора на молекулу-акцептор(спирт).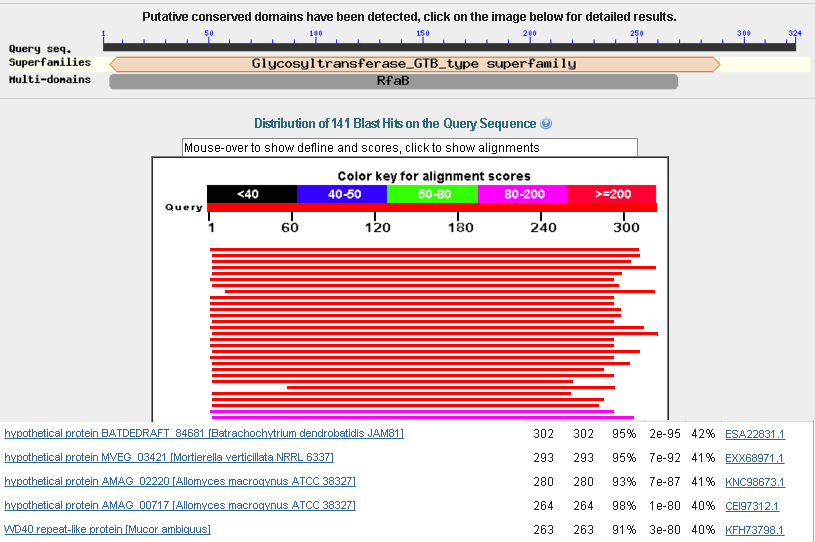
Рис.3. Результаты проверки g13 в blastp.
g6
Нашелся домен белков, относящихся к суперсмейству CHROMO. Находки все недостоверные. Самая лучшая находка и некоторые последующие содеражат больше аминокислот, чем искомая последовательность, поэтому в середине длинный гэп. Это могло произойти из-за неправильного предстказания AUGUSTUS экзон-интронной структуры. (Рис.4.)
Рис.4. Результаты проверки g6 в blastp: выранивание лучшей находки с искомой последовательностью.
Экспериментальных подтверждений этой находки нет. Процент идентичности низкий и высокий e-value, к тому же AUGUSTUS много ошибается. Скорее всего эта находка тоже ошибка AUGUSTUS.g8
Нашлось сразу два домена. E-value пониже, чем в предыдущем примере и процент идентичности слабо перевалили за 50. Но главное, что не все находки подтверждены только аннотацией.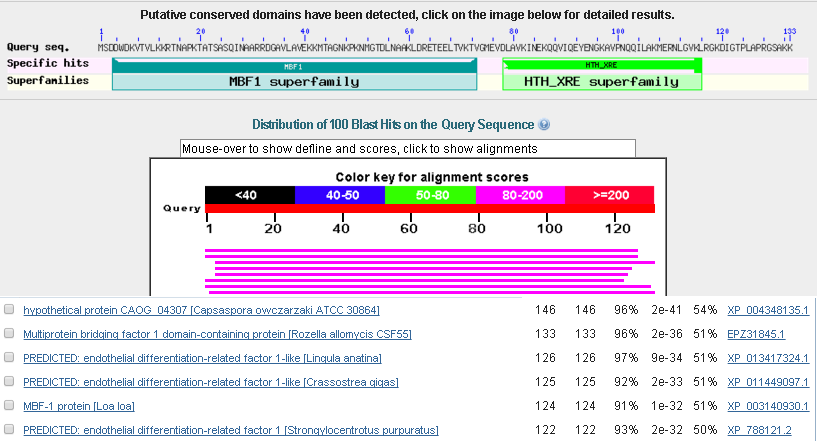
Рис.5. Результаты проверки g8 в blastp.
На рисунке 6 представлен большой гэп - следствие ошибки AUGUSTUDS в определении экзон-интронной стрекутуру.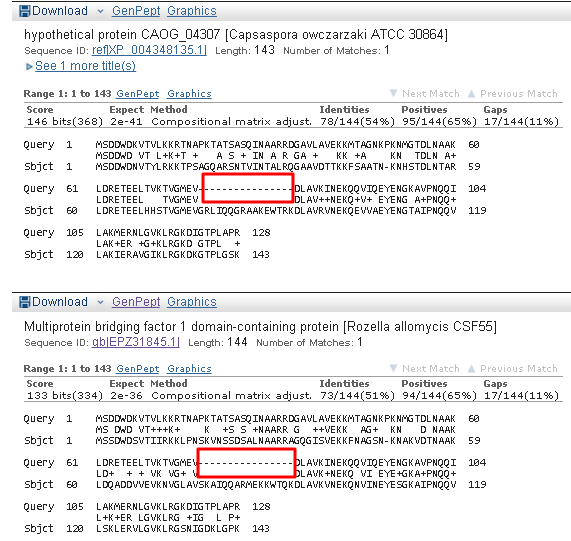
Рис.6. Выравнивания первых двух находок: гипотетической и нет - с искомой последовательностью.
g10
Если в предыдщих примерах можно было спорить о достоверности находок, то в этом определенно нет достоверных. Минимальное e-value равно 8,2.
Рис.7. Результаты проверки g10 в blastp.
g9
E-value высокое. Находки не достверны. Но для них есть подтверждение сильнее, чем "предполагаемое из аннотации"(IEA) - ISS("предполагаемое из последовательности и сходства структуры").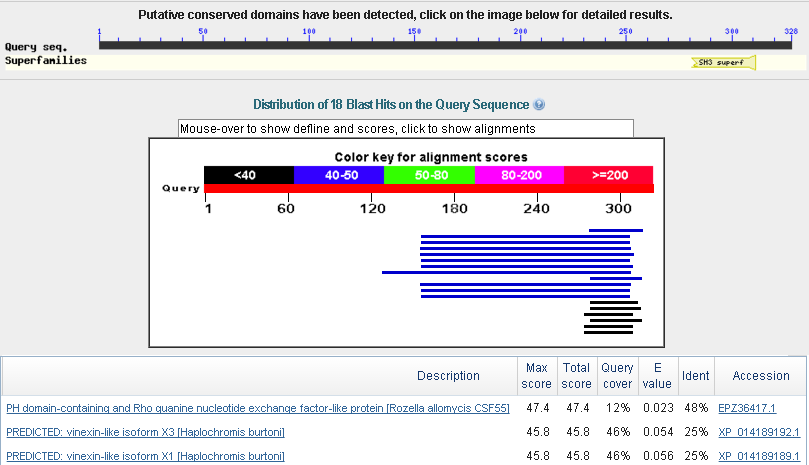
Рис.8. Результаты пррверки g9 в blastp.
Задание 2. Сравнение аннотаций Refseq и AUGUSTUS одного гена человека.
Для сравнения я выбрала ген GYS2. Белок, кодируемый этим геном, - гликоген синтаза печени - катализирует ограниченную по скорости ступень синтеза гликогена - перенос глюкозы с молекулы УДФ-глюкозы на гликоген, терминируя полимеризацию. Его координаты chr12:21,531,527-21,630,773, обратная цепь. На рисунке 9 белок GYS2 в геномном браузере с треками Base Position, RefSeq Genes и Augustus.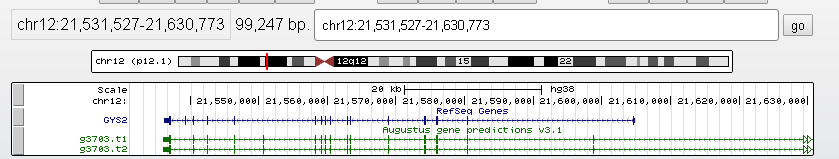
Рис.9. Окно геномного браузера с аннотациями для GYS2 из RefSeq и Augustus.
С помощью инструментов геномного браузера получены таблицы с экзонно-интронной структурой для аннотаций. Далее эти таблицы были обработаны в Excel. Ссылка на результат. На листе "таблица с экзонами и интронами" представлены экзоны и интроны без повторов. Их длины посчитаны на листе "eukaryotes"(c небольшими комментариями).В этом задании AUGUSTUS проявил себя куда лучше. Он смог правильно аннотировать 12 из 17 экзонов. Всего у него было 23 или 24 экзона - на картинке видно, что у обеих его аннотаций длинные хвосты по сравнению c RefSeq. Эти хвосты к белку GYS2 не относятся. Понять, какая из аннотаций лучше не получится, потому что различия между ними есть только там, где уже экзонов RefSeq. Но по суммарной длине CDS первая аннотация(1375 аминокислот) ближе к аннотации RefSeq(1044 аминокислоты) и в ней число нуклеотидов нацело делится на 3.