Задание 2. Поиск последовательности Шайна – Дальгарно в геноме бактерии из первого семестра
Последовательность Шайна-Дальгарно располагается перед старт-кодоном генов бактерий и архей на 3'-конце и участвует в стабилизации комплекса малой субъединицы рибосомы и мРНК[1].Из базы данных GenBank был скачан полный геном(хромосома) и future table для моей бактерии из первого семестра Thermus Thermophilus HB8. Из них скриптами features2CDSs.py и fragments2fasta.py были извлечены последовательности перед старт-кодонами "хороших" белков(длина 300-1000 п.н., в названии отсутствуют слова "hypothetical", "putative", "probable"). Т.к. консенсусная последовательность Шайна-Дальгарно AGGAGG состоит из 6 нуклеотидов, а располагается в среднем на расстоянии 10 нуклеотидов от стартого кодона, для поиска таких последовательностей в белках моей бактерии был взят интервал от старт-кодона в 16 нуклеотидов. На обратной цепи этот участок находится после последовательности белка, поэтому 16 не вычиталось из наименьшей координаты, а прибавлялось к наибольшей. И в итоге длина участка, на котором проводился поиск мотива составила 17 нуклеотидов.
В MEME было задана длина мотива 4-10 нуклеотидов и количество: три мотива(чтобы было, из чего выбрать). Первый мотив с наибольшим E-value(2.6e-190) похож на последовательность ШД(Рис.1).

Рис. 1. LOGO найденного мотива.
Далее с помощью мотива и матрицы вероятностей, построенной MEME по "хорошим" белкам в FIMO был проведен поиск по всем белкам с расширенными границами координат поиска(21 нуклеотид). Результаты поиска в файле fimo и на рисунке 2. Наиболее часто(77 из 282) в геноме Thermus Themophilis HB8 встречаются гены с последовательностью Шайна-Дальгарно, начинающейся за 13 нуклеотидов до старт-кодона. Из 1973 генов бактерии последовательность Шайна-Дальгарно обнаружена только в 282(14,3%), значит, у Thermus Thermophilus HB8 имеются другие механизмы стабилизации трансляционного комплекса.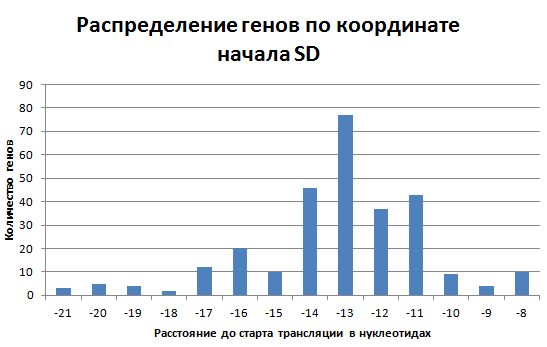
Рис. 2. Гистограмма, отражающая распределиние генов по координате начала последовательности Шайна-Далььгарно. (За ноль принят первый нуклеотид старт-кодона).
Ген argF(F цепи орнитинкарбомоил трансферазы, участвующей в биосинтезе L-аргинина) не содержит последовательности Шайна-Дальгарно, но содержит другую последовательность с 5'-конца, которая может стабилизировать трансляцию. Эта же последовательность была найдена в 64% генов из 130 генов Thermus, проанализированных Sanchez R et. al[2].Ссылки:
1. Shine J, Dalgarno L (1973). "Occurrence of heat-dissociable ribosomal RNA in insects: the presence of three polynucleotide chains in 26S RNA from cultured Aedes aegypti cells". Journal of Molecular Biology. 75: 57–72.2. Sanchez R, Roovers M, Glansdorff N. Organization and expression of a Thermus thermophilus arginine cluster: presence of unidentified open reading frames and absence of a Shine-Dalgarno sequence. J Bacteriol. 2000;182:5911–5.
Задание 3. Определение сайтов связывания данного транскрипционного фактора в данном участке хромосомы человека
Мне была выдан файл с чтениями с секвенатора №35. Командой fastq был проведен анализ чтений(Результат). Их качество хорошее, так что чистить Trimmomatic не надо. Далее чтения картировались следующими командами:samtools view -bSo chipseq_chunk35.bam chipseq_chunk35.sam
samtools sort chipseq_chunk35.bam -T chip_temp -o chipseq_chunk35.sorted.bam
samtools index chipseq_chunk35.sorted.bam
samtools idxstats chipseq_chunk35.sorted.bam > chipseq_chunk35.idxstats
samtools view -c chipseq_chunk35.sorted.bam
На геном откартировались все чтения 5957 штук. Больше всего чтений на одиннадцатой хромосоме. (Рис. 3.)
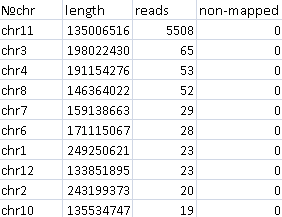
Рис. 3. Результаты картирования из файла *.bam
Командой macs2 callpeak были найдены 8 пиков. Визуализировали их с помощью геномного браузера(Рис.4).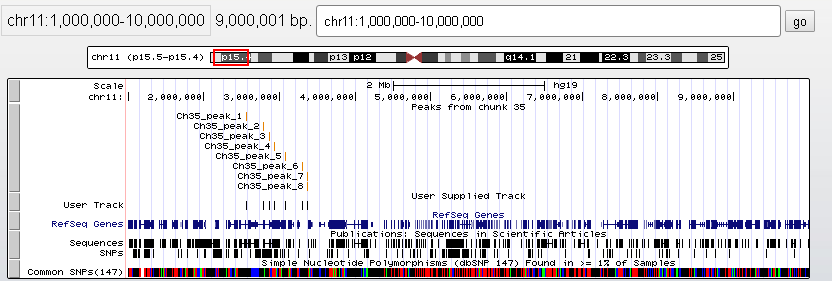
Рис.4. Пики в геномном браузере. Пики с именами Ch35_peak_№ из файла .narrowPeak, следующая дорожка User track из файла *_summits.bed.
Три пика 1, 2, 3 попали на интрон гена KCNQ1(Рис. 5). Чем больше отрицательный логарифм от p-value и q-value, тем достовернее пик. У первого пика они высокие.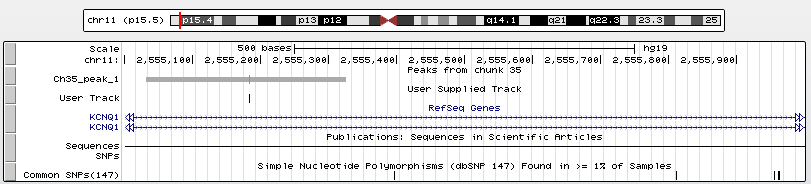
Рис. 5. Увеличенный первый пик. Локализация chr11: 2555031-2555325. Длина 294 п.н.
Самый широкий пик - седьмой(Рис.6) Его вершина сдвинута немного в право. На его месте нет генов, и значения логарифмов p-value и q-value меньше, чем у первого.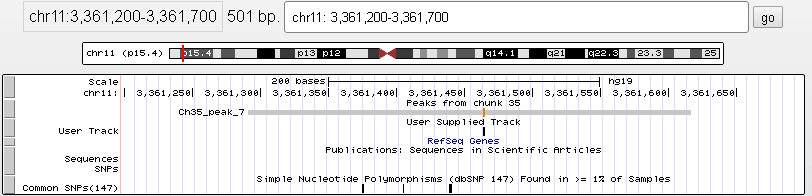
Рис.6. Увеличенный седьмой пик. Локализация chr11: 3361291-3361617. Lkbyf 326 п.н.
Задание 4. Поиск генов в геноме человека, транскрипция которых инициируется с помощью TATA-бокс связывающего белка и без сигнала TATA-бокса в промоторной области
TBP - архейный и эукариотический белок, узнающий восьминуклеотидный сигнал в ДНК, называемый TATA-box, с консенсусом TATAWAAR. Был выбран эксперимен ChIP-seq на клеточной линии K562(клетки лейкемии) с мышиными антителами к IgG. (Табл.1)1. CCDC80, обеспечивающий клеточную адгезию и поддерживающий форму клетки, локализация: chr3:112323233-112359990, цепь:-, длина: 36758
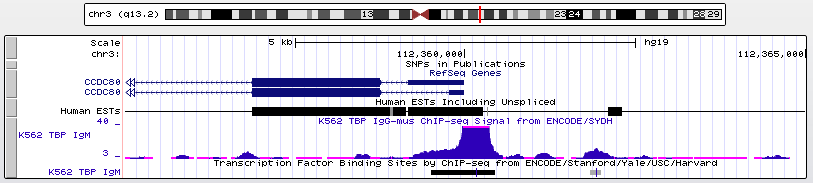
Рис. 7. Ген CCDC80, содержащий в промоторной области TATA-бокс.
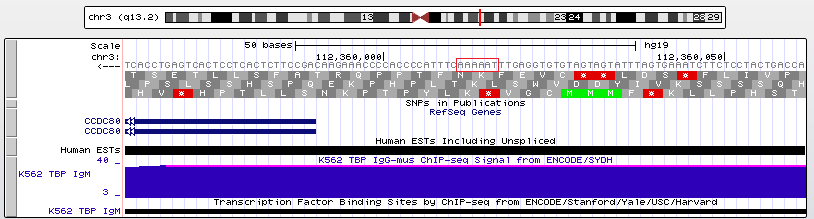
Рис. 8. Увеличенная промоторная область. TATA-бокс находится на расстоянии примерно 20 п.н.
2. ATP2C1, АТФ-зависимый транспортер ионов кальция, локализация: chr3:130569369-130722046, цепь:+, длина: 152678
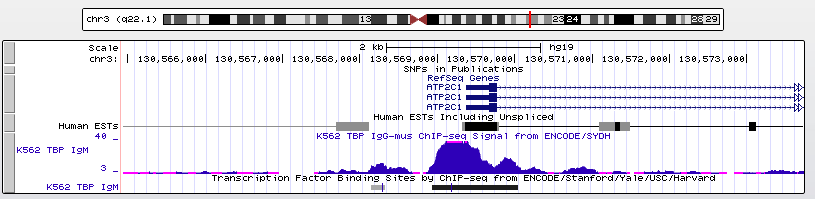
Рис. 9. Ген ATP2C1, содержащий в промоторной области TATA-бокс.
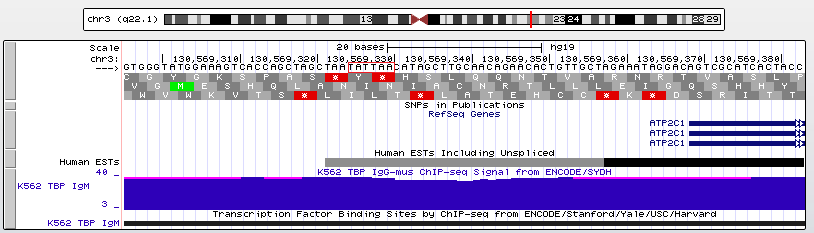
Рис. 10. Увеличенная промоторная область. TATA-бокс находится на расстоянии примерно 40 п.н.
3. EK11, преставитель семейства NIMA-киназ(никогда не входящих в митоз группы А), локализация: chr3:130745694-131069309, цепь:+, длина: 323616
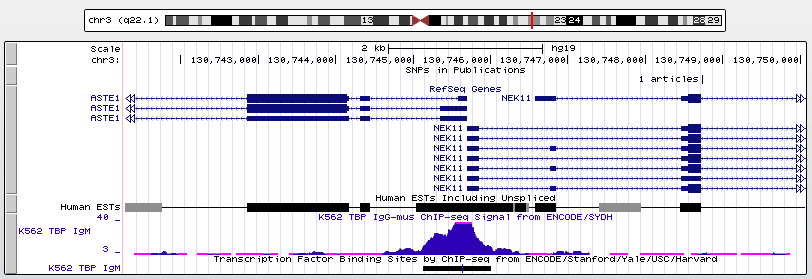
Рис. 11. Ген CCDC80, содержащий в промоторной области TATA-бокс.

Рис. 12. Увеличенная промоторная область. TATA-бокс находится на расстоянии примерно 40 п.н.
4. QTRT2, субъединица гуаниновой тРНК гликозилазы, локализация: chr3:113775582-113807268, цепь:+, длина: 31687
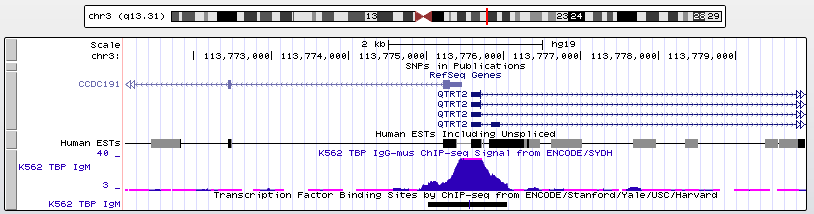
Рис. 13. Ген QTRT2, не содержащий в промоторной области TATA-бокс.
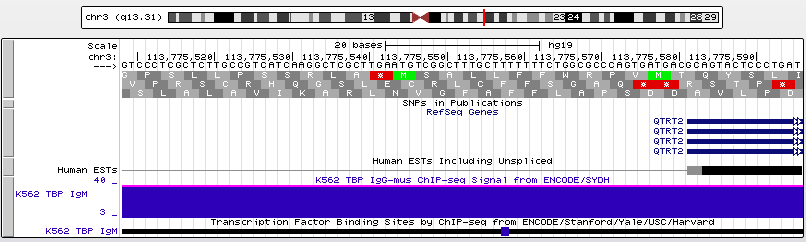
Рис. 14. Увеличенная промоторная область. TATA-бокс отсутствует.
Генов без TATA-бокса больше, чем с ним. Располагается он на расстоянии 20-40 п.н. от старта трансляции.