1. Составление списка белков целевого семейства из `SwissProt`
В качестве целевого семейства были выбраны белки Млекопитающих, содержащих домен TIR (PF01582), который ранее анализировался в практикуме 7 . Для этого в Uniprot был введен следующий поисковый запрос:database:(type:pfam id:PF01582) taxonomy:"Mammalia [40674]" AND reviewed:yes
Полученные данные были собраны в таблице excel. Скачать файл по ссылке, страница "all data". Для анализа было выбрано подсемейство белков, содержащее домены PF13895;PF01582 и PF13855;PF01582; (Первый содержит помимо TIR еще иммуноглобулиновый домен, а второй - лейцин-богатый домен). Список отобранных белков можно найти на второй странице ("selected_subfamily") того же файла.
2. Построение профиля
Затем по одной и той же схеме было построено два профиля. Один - рандомная выборка белков, содержащих домен PF01582 (в Jalview PF01582 seed), другая - на основе выравнивания 22 белков, принадлежащих к выбранному подсемейству (выравнивание получено с помощью скрипта как в практикуме 7). C помощью пакета HMM был получен профиль, откалиброван, а также проведен поиск с помощью этого профиля по базе данных uniprot. Сводная таблица, содержащая команды и результаты.| Команда | Исходный файл (seed) | Исходный файл (мой профиль) | Полученный файл (seed) | Полученный файл (мой профиль) |
| hmm2build -g output input | testalign.fasta | nadya_filtered.fasta | profile | profile2 |
| hmm2calibrate --num 5000 input | profile | profile2 | - | - |
| hmm2search -E 10000 -T 50 input /srv/databases/emboss/data/uniprot/uniprot_sprot.fasta > output |
profile | profile2 | result | result2 |
3. Анализ полученных результатов
На 3 и 4 листе файла excel находится информация о находках доменов для двух построенных профилей. Профиль, составленный из рандомной выборки нашел 15 из 30 последовательностей указанного подсемейства и 47 из 99 последовательностей, принадлежащих млекопитающим. Профиль, составленный из белков выбранного подсемейства нашел 29 из 30 последовательностей указанного подсемейства и 83 из 99 последовательностей белков млекопитающих. Уже при таком грубом анализе можно сказать, что профиль, построенный на основе подсемейства ищет белки подсемейства и млекопитающих лучше, чем профиль, построенный на рандомной выборке. Гистограммы весов находок, для данных профилей приведены на рисунке 1.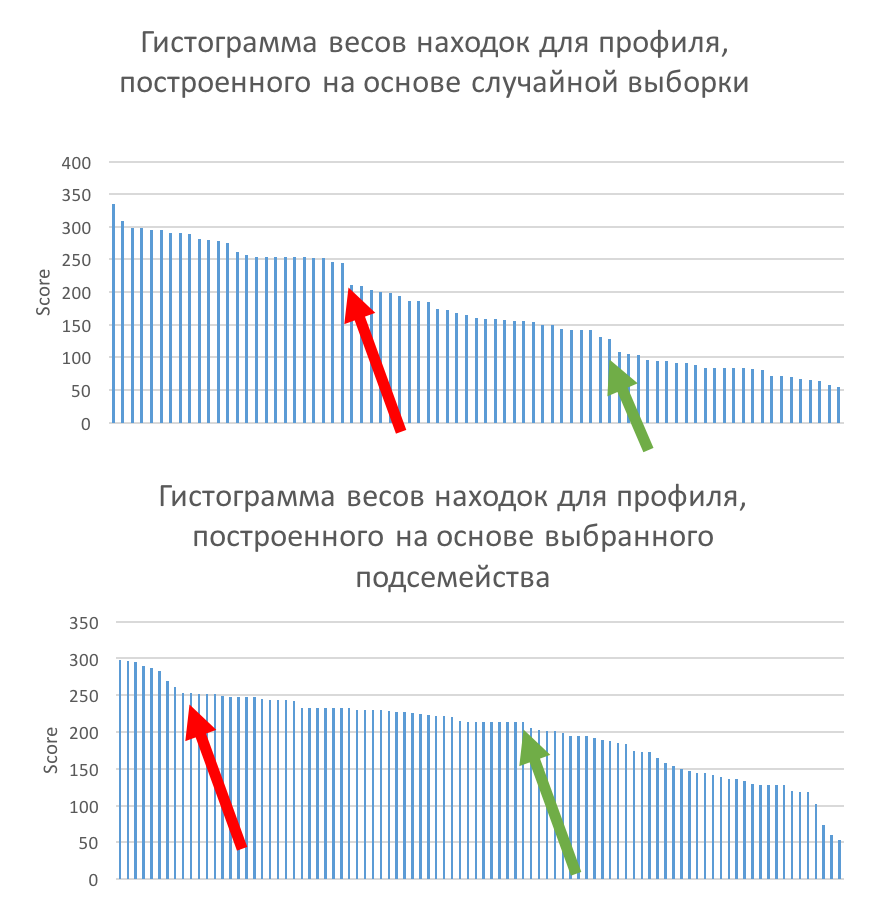
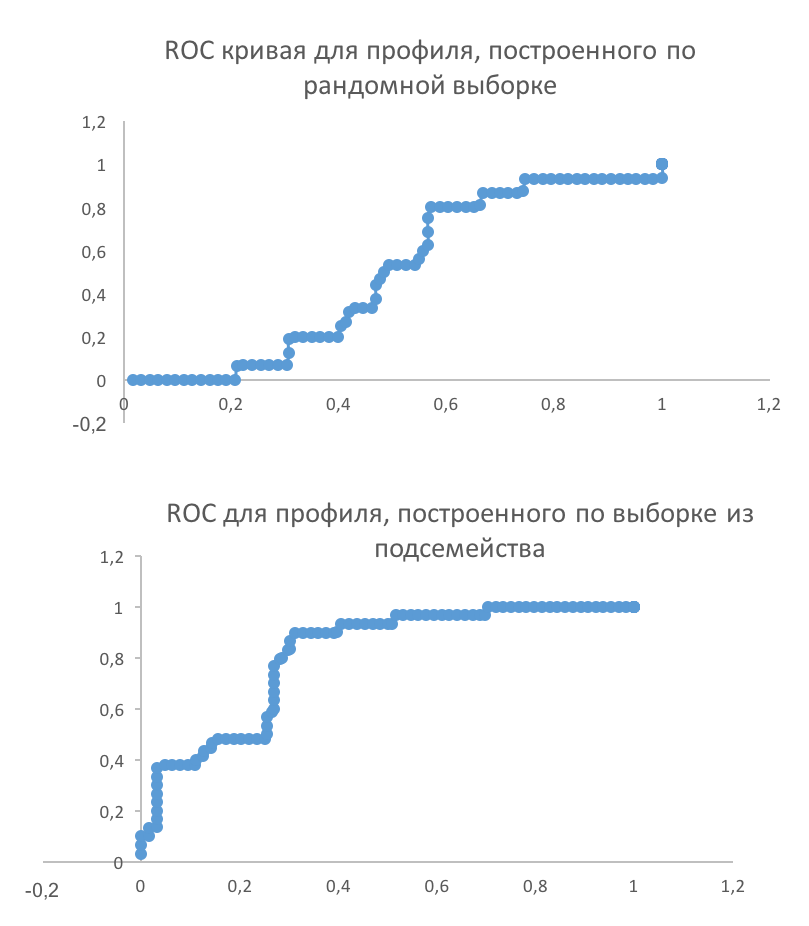
Затем для полученных данных были получены ROC-кривые (Receiver Operator Characteristic) (показывают зависимость количества верно классифицированных положительных примеров от количества неверно классифицированных отрицательных примеров). Для построения кривой были определены:
TP (True Positives) — верно классифицированные положительные примеры (так называемые истинно положительные случаи). Это количество последовательностей, расположенных выше порога и содержащие искомый домен;
TN (True Negatives) — верно классифицированные отрицательные примеры (истинно отрицательные случаи). Это количество последовательностей, расположенных ниже порога и не содержащих искомый домен;
FP (False Positives) — отрицательные примеры, классифицированные как положительные (ошибка II рода, ложно положительные случаи). Это количество последовательностей, расположенных выше порога, но не содержащих домен;
FN (False Negatives) — положительные примеры, классифицированные как отрицательные (ошибка I рода, ложно отрицательные примеры). Это количество последовательностей, расположенных ниже порога и содержащих домен.
Кроме того необходимо определить еще несколько параметров: специфичность (SP) — доля истинно отрицательных случаев, которые были правильно идентифицированы моделью (т.е. доля предсказанных белков, не содержащих домен, от общего количества последовательностей, известно не содержащих этот домен) и чувствительность (SE) — доля истинно положительных случаев (т.е. доля предсказанных белков, содержащих домен, от общего количества последовательностей, известно содержащих домен). Эти показатели считаются по следующим формулам: SP = TN/(TN+FP), SE = TP/(TP+FN).
ROC-кривая представляет собой зависимость SE от 1-SP. Приведенные вычисления проведены на 5 и 6 страницах указанного выше excel-файла. Полученные ROC-кривые приведены на рисунке 2. Для выбора порога необходимо максимизировать SE и SP, то есть выбрать такую пару, для которой максимально значение (SE+SP-1). Для неспецифичного профиля это оказалось 0,229, соответствующее значение порога для веса: 143,6; для специфичного профиля - 0,585, вес - 213,8.
| TP | TN | FP | FN | |
| profile1 | 12 | 27 | 36 | 3 |
| profile2 | 26 | 44 | 20 | 3 |
На основе полученных результатов можно сказать, что профиль, построенный по выборке белков из подсмейста выделяет это подсемемейство лучше, чем профиль, выбранный из рандомных последовательностей, содержащих этот домен. Кроме того полученный профиль обладает специфичностью 68,8% и чувствительностью 89,7%, что позволяет его использовать для поиска белков млекопитающих, содежащих доменные архитектуры PF13895;PF01582 и PF13855;PF01582.