Blast
Поиск гомологов белка в Swiss-Prot
Для выполнения задания я взял белок из практикума #7 - A0A3Q8WHX8.
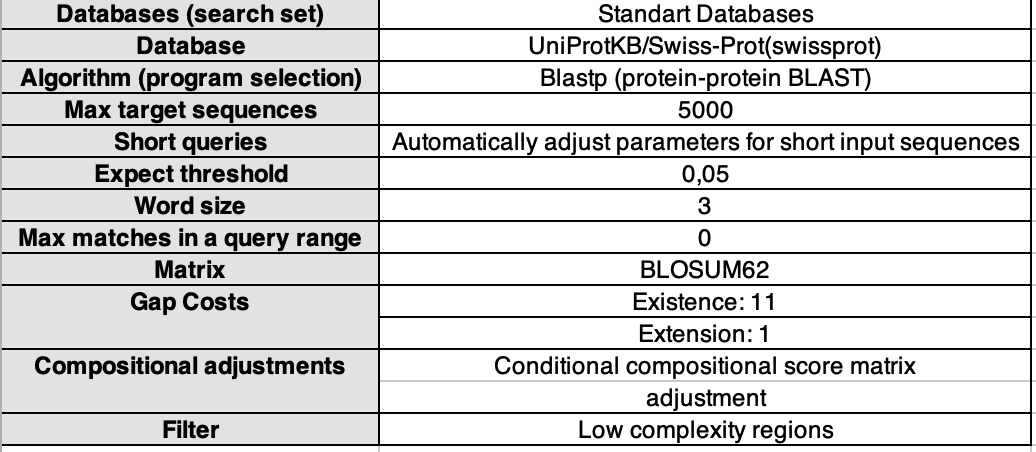
Параметры поиска гомологов:
По запросу было найдено 17 гомологов. Случайным образом я отобрал 6 белков (включая исходный) для множественного выравнивания. Выравнивание производилось в программе Jalview методом T-coffee. Исходя из результатов выравнивая, я сделал вывод, что белки гомологичны. Наиболее консервативные участки находятся на интервале 0-455.
Файл выравнивания в формате Jalview
Организмы, которые участвовали в выравнивании: Escherichia coli K-12, Haemophilus influenzae Rd KW20, Buchnera aphidicola str. Sg (Schizaphis graminum), Buchnera aphidicola str. APS (Acyrthosiphon pisum), Halomonas venusta, Buchnera aphidicola str. Bp (Baizongia pistaciae).
Поиск гомологов зрелого вирусного белка, вырезанного из полипротеина
Из базы данных Swiss-Prot я взял вирусный полипротеин Polyprotein pp62 (PP62_ASFB7), принадлежин он организму African swine fever virus (strain Badajoz 1971 Vero-adapted).

С помощью команды была получена цепь p15 полипротеина: seqret sw:PP62_ASFB7[2:158] segment.fasta
Fasta файл цепи полипротеинаВсе параметры поиска Blast, за исключением фильтра low complexity regions, остались неизменными. Результатом выполнения запроса было 4 находки гомологов.
В результате выравнивания этих последовательностей была установлена гомологичность.
Выравнивание полипротеина virus.jvpИсследование зависимости E-value от объёма банка
Число находок не изменилос, но E-Value для каждой изменился.
В первом поисковом запросе (без фильтров) для последовательности P0CA08.1 E-value равен 3e-111, во втором поиске (фильтр по вирусам) E-value равен 1e-112.
После произведения вычислений: E-value2/E-value1 * 100, доля вирусных белков составила 3.3%.