Выбор структуры белка
Структура гистидин-содержащего белка-переносчика фосфора в системе фосфотрансферазы Bacillus subtilis (PDB ID
2FEP, разрешение 2.45 ангстрем) была выбрана для дальнейшего анализа, также проверено, что структура удовлетворяет следующим требованиям: 387 результатов поиска, из которых при ограничении поиска параметрами RMSD (Root Mean Square Deviation) от 0.8 до 3.0 ангстрем и Nalgn от 140 до 247 -50%-90% числа остатков 2FEP) подходят следующие структуры:1DRJ,4KMR,3BIL,3HUU,4RKQ,3QK7,3E61
Структурные факторы
Из базы данных PDB были загружены модель
2FEP и файл структурных факторов. Число атомов в модели, а также число отражений (reflections) были найдены следующим образом:
grep -Eh ^ATOM\|^HETATM 2FEP.pdb | wc -l # 2885 = number of atoms
grep -Eh ^ATOM\|^HETATM 2FEP.cif | wc -l # 2885 = number of atoms
grep -Eh '[0-9]{3}' 2fep-sf.cif | wc -l # 13165 = number of reflections В файле 2fep-sf.cif присутствуют следующие данные для каждого отражения ( h, k, l- индексы рефлекса)
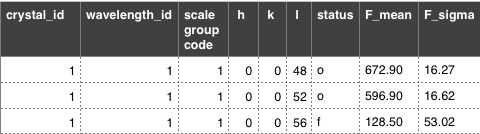 |
Изображение электронной плотности
Файл с картой электронной плотности для 2FEP был загружен с сайта EDS. Для визуализации структуры и электронной плотности была использована программа для визуализации молекулярных данных PyMol.
Изображения электронной плотности вокруг полипептидной цепи (для уровней изолиний 1.0 , 1.5 и 2.0 на указанных расстояниях от выбранного множества – параметр carve) были получены с помощью команд
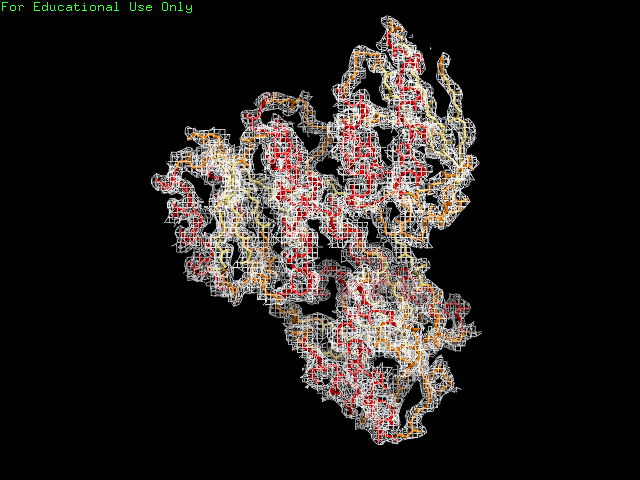 |
|
Рис. 1 Level 1 | |
 |
|
Рис. 2 Level 2 | |
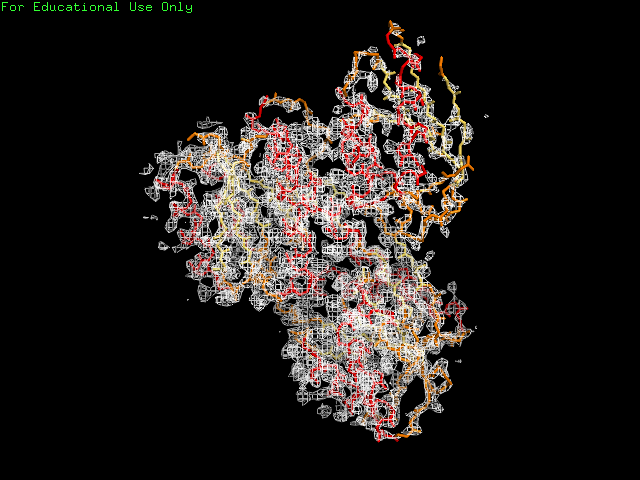 |
|
Рис. 3 Level 3 |
|
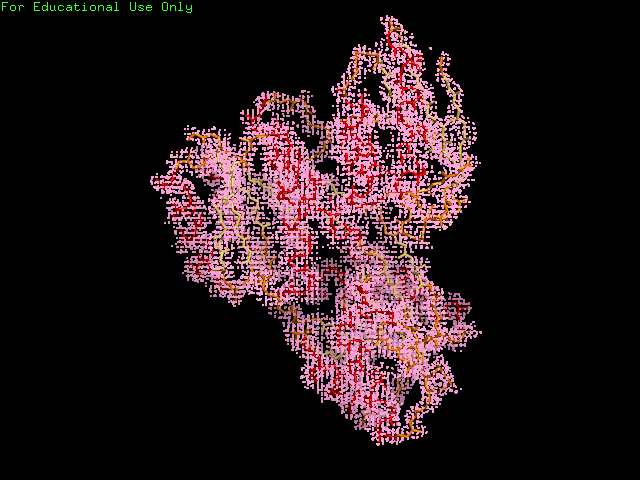 |
|
Рис. 4 Level 4 |
Код, с помощью которого были получены изображения электронной плотности вокруг аминокислотных остатков:
load 2fep.pdb
load 2fep.omap, 2fep_map
hide all
show sticks, all
set stick_radius, 0.05
select tyr, resi 76 and chain A
show spheres, tyr
set sphere_scale, 0.2, tyr
isomesh map, 2fep_map, 0.5, tyr, carve=2.5
color grey, map
set mesh_width, 0.5
center tyr
zoom tyr 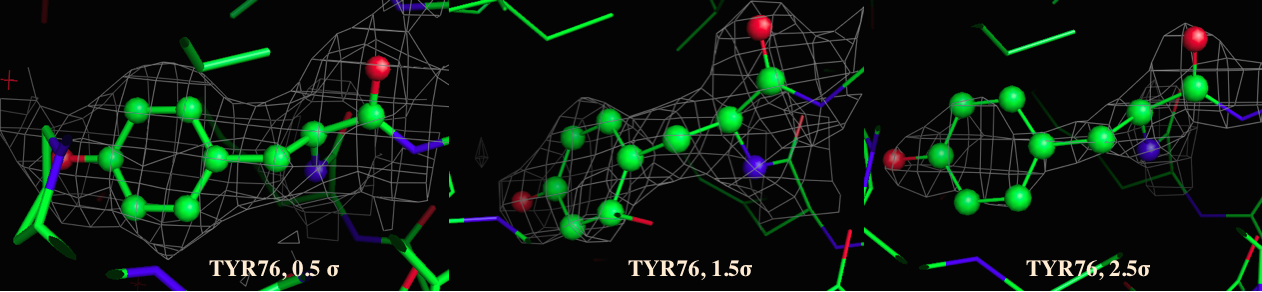 |
|
Рис. 5 Электронная плотность для участка структуры Tyr76 | |
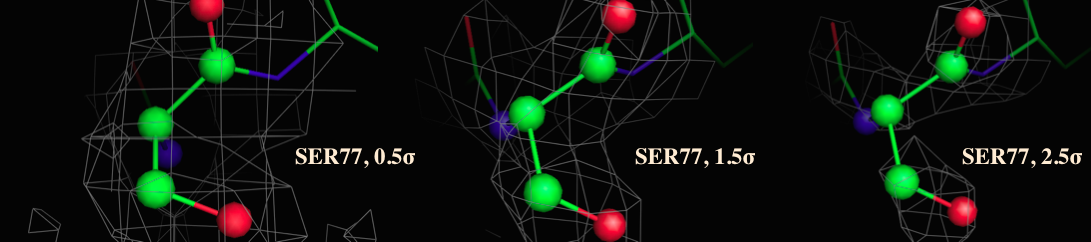 |
|
Рис. 6 Электронная плотность для участка структуры Ser77 | |
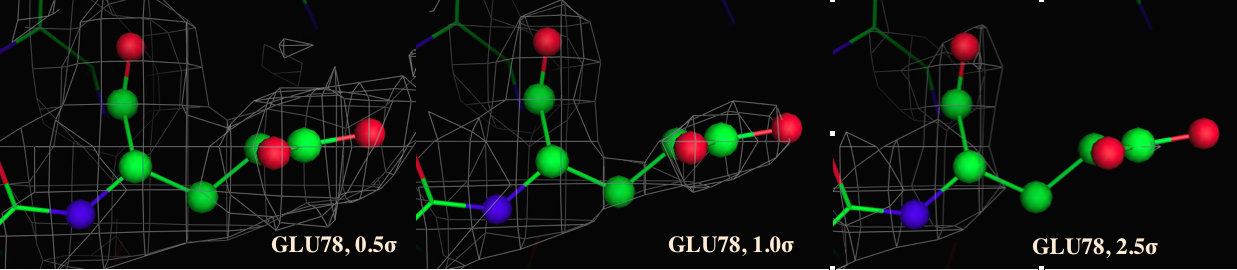 |
|
Рис. 7 Электронная плотность для участка структуры Glu78 |
|
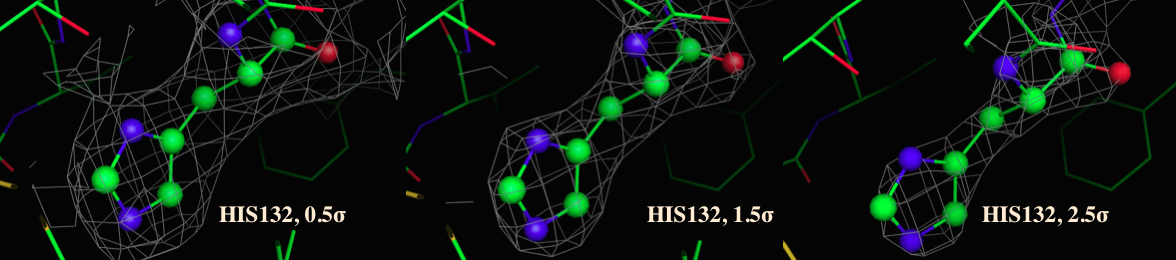 |
|
Рис. 8 Электронная плотность для участка структуры His132 |
© Boskhomdzhieva Baina, 2015