В качестве нуклеотидных последовательностей были взяты 16s рРНК бактерий из практикума 1, полученные из базы полных геномов NCBI. Последовательности генов рРНК и тРНК там записаны в файлах с расширением .frn. Все последовательности были собраны в единый fasta-файл, с помощью Muscle я построил их выравнивание. Затем в программе MEGA было построено филогенетическое дерево методом Maximum Likelihood. Полученное дерево представлено на рисунке 1.
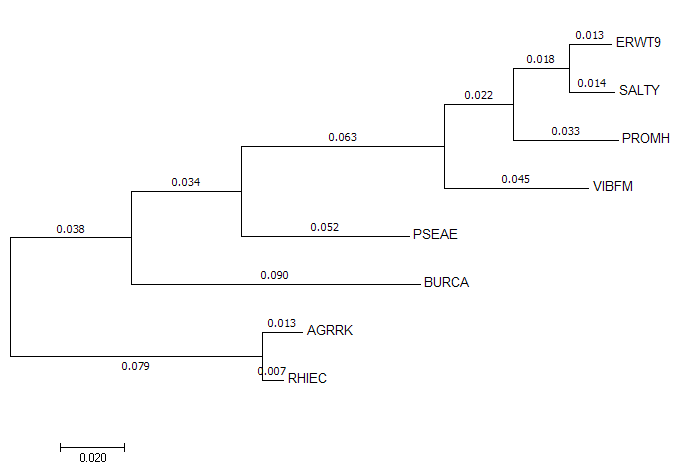
Рисунок 1. Филогенетическое дерево выбранных протеобактерий, построенное по выравниванию последовательностей 16s рРНК методом Maximum Likelihood.
Как видно, полученное дерево по топологии полностью совпадает с правильным деревом (из общего дерева протеобактерий). Стоит отметить, что всеми методами верхней тройки (ML, NJ, ME) получаются одинаковые деревья, поэтому можно считать их верными. Таким образом, деревья, построенные по нуклеотидным последовательностям оказываются более правильными, чем деревья, построенные по последовательностям белков.
С помощью blastp в полных протеомах отобранных бактерий были найдены гомологи белка CLPX_ECOLI (выдача blast на рисунке 2). С порогом E-value 0.001 найдено 28 белков, однако в дальнейшем использовалось только первые 17 находок (E-value < 7e-17), потому что эти белки имеют одинаковые функции и очень низкое E-value.
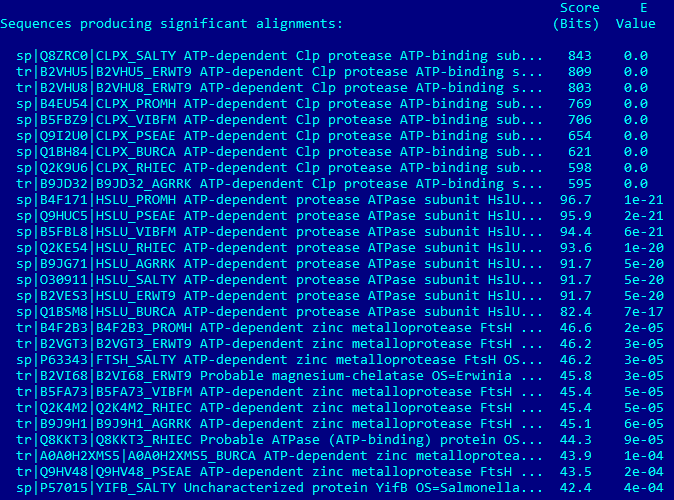
Рисунок 2. Выдача blastp при поиске гомологов белка CLPX_ECOLI в протеомах протеобактерий.
Последовательности гомологов были собраны в один файл и выровнены программой Muscle в JalView. Затем я построил филогенетическое дерево в программе MEGA методом Neighbour-Joining (рисунок 3). Ссылка на дерево в формате Newick
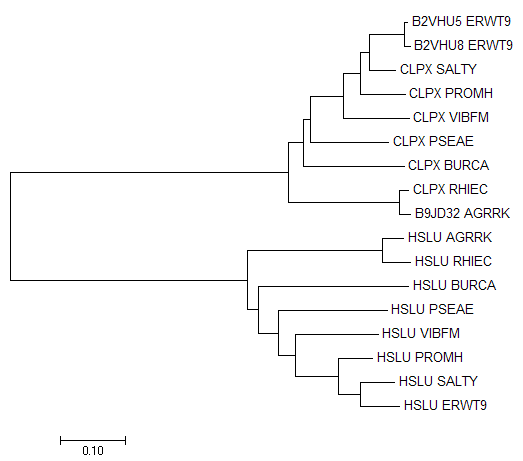
Рисунок 3. Филогенетическое дерево гомологов белка CLPX_ECOLI, построенное методом NJ. При наведении мышкой на изображение появляются обозначения: красной рамкой выделена группа попарно ортологичных белков, оранжевыми, синими и розовыми линиями - пары паралогов, зеленой рамкой отмечен пример дупликации гена в одном организме, голубыми линиями - разделение путей эволюции белков в результате видообразования.
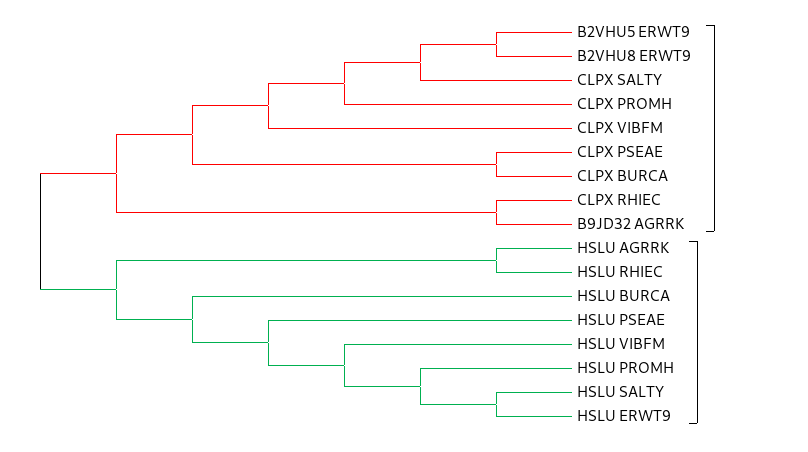
Рисунок 4. Филогенетическое дерево гомологов белка CLPX_ECOLI, построенное методом NJ. Группы ортологов покрашены разными цветами.

Рисунок 5. Филогенетическое дерево гомологов белка CLPX_ECOLI, построенное методом NJ, группы ортологов покрашены разными цветами и схлопнуты. Группы содержат соотвествующие по цвету белки, указанные на рисунке 4. Каждая группа содержит белки из всех рассматривавшихся бактерий, филогения белков соответствует филогении бактерий.