BLAST
Гомологи белка "Type IV secretion system protein" бактерии Bartonella krasnovii
В практикуме 7 мы рассматривали белок системы секреции IV бактерии Bartonella krasnovii,который участвует в бактериальной конъюгации и доставке бактериальных эффекторных белков в цитоплазму клеток-хозяев млекопитающих с целью нарушения клеточных функций хозяина. Поэтому ещё интереснее будет изучить гомологичные белки у других патогенных бактерий, это может продвинуть нас в понимании работы белков системы секреции IV и навести на идеи новых антибиотиков.
Было решено производить поиск по FASTA последовательности
Затем был установлен фильтр на процент идентичности от 30%, чтобы отсеять часть случайно гомологичных белков. Получилось 14 последовательностей. Уже можно сделать вывод, что этот белок довольно специфичен.
текстовый файл результатов поиска BLASTИнтересно, что наибольший процент идентичности - у неохарактеризованного белка из Methanocaldococcus jannaschii. Это термофильная метаногенная архея. Выглядит он следующим образом:
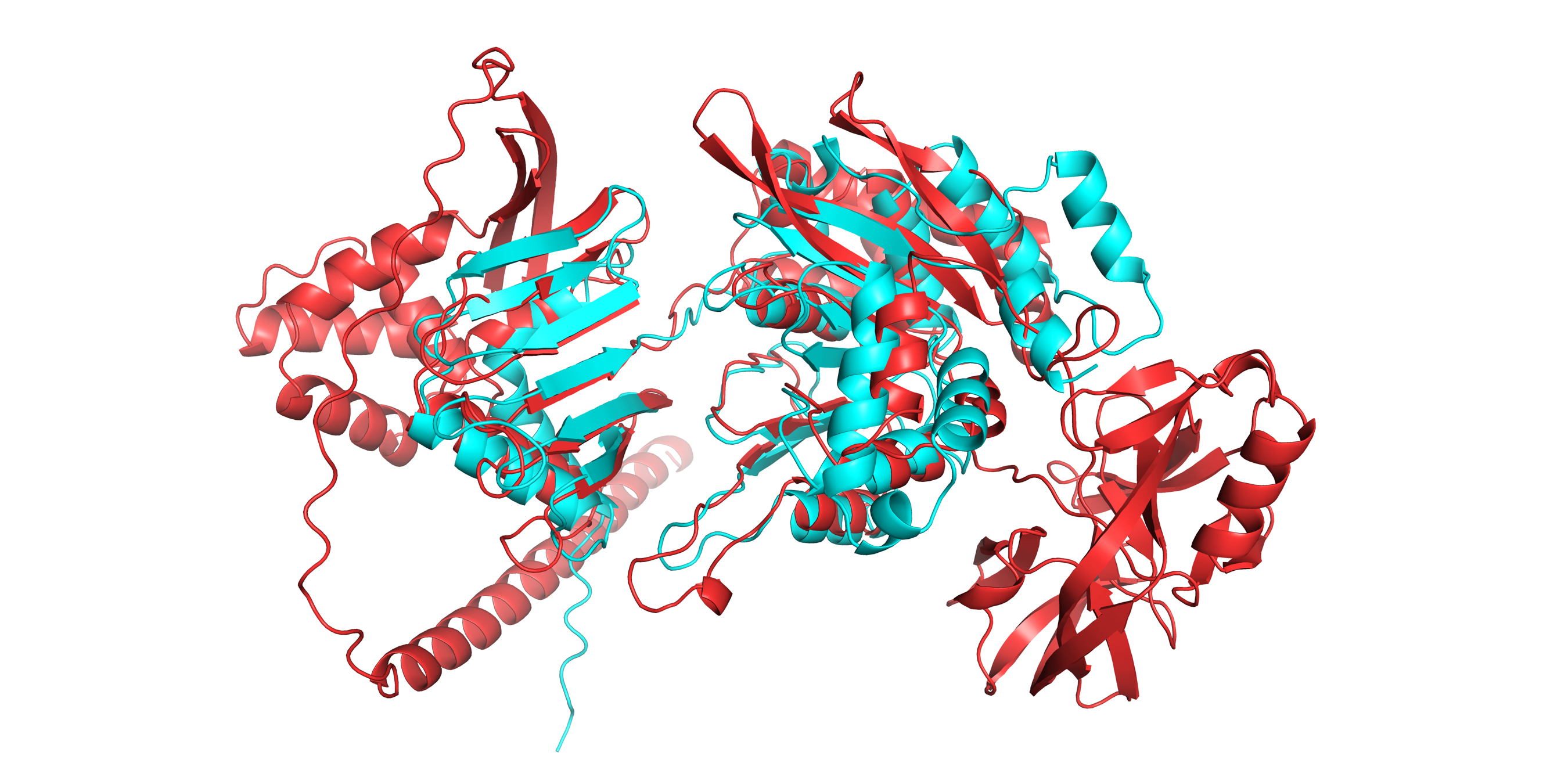
Для выравнивания мы выбрали: -- Q58191.1 RecName: Full=Uncharacterized protein MJ0781; Contains: RecName: Full=Mja klbA intein [Methanocaldococcus jannaschii DSM 2661] - чтобы оценить гомологию с наиболее идентичным белком -- Q6FYW0.1 RecName: Full=Type IV secretion system protein VirB11 [Bartonella quintana str. Toulouse] - чтобы сравнить с бактерией того же рода что и наша B. krasnovii -- Q9RNC7.1 RecName: Full=Type IV secretion system protein VirB11 [Bartonella henselae str. Houston-1] -- Q8FXK7.1 RecName: Full=Type IV secretion system protein VirB11 [Brucella suis 1330] - сравнить с бактерией другого рода -- Q7WDT5.1 RecName: Full=Type IV secretion system protein PtlH homolog [Bordetella bronchiseptica RB50] - сравнить с гомологом нашего белка
Выравнивание производим в Jalview с помощью Web Service - Alignment - Muscle with defaults
Белок из археи в два раза длинее - 721 аминокислота, в то время как у белков T4SS примерно 360 АА. Вероятно, такой разницей в длине и объясняется высокая степень идентичности. В выравнивании получилось очень много гэпов. Хотя есть и консервативные участки: 209-282 ; 322-327 ; 336-351; 364-374; 408-439. Удалим его из выравнивания, заменим белком бактерии из рода Brucella: Type IV secretion system protein VirB11 [Brucella abortus bv. 1 str. 9-941].
файл Jalview с выравниванием белков системы секреции IVВидим, что белок из Bordetella bronchiseptica RB50 действительно гомологичен, как и указано в названии. Эта бактерия может привести к инфекционному бронхиту, но редко заражает людей. Микроорганизм находится в эволюционном родстве с Bordetella pertussis, вызывающей коклюш у человека. Факт патагенности бактерии объясняет наличие сисемы секреции IV типа.
Самым длинным консервативным участком можно назвать 250-336. Ещё примечателен участок 178-225. На этом промежутке нет гэпов, много колонок identity=100% и функционально-консервативных колонок.
Гомологи вирусного белка
Мы выбрали New York virus (NYV):
ID: GP_NYV
AC: Q83887; Q83886; Q83888
выбрали отдельную цепь - Glycoprotein N (18..652)
Извлекаем последовательность в файл:
extractseq -sequence Q83887.txt -outseq Glycoprotein_N.fasta -regions 18-652текстовый файл результатов поиска BALST
При поиске в BLAST мы получили 18 последовательностей, для каждой E-Value 0, то есть выравнивание почти идеальное - высокая идентичность при стопроцентном покрытии выравнивания. 13 находок это такие же гликопротеины N, 5 оставшихся это Glycoprotein precursor, то есть предшественники гликопротеинов.
Выбрали следующие белки: Q83887.1 (наш NYV),Q9E006.1, P41265.1, P27315.1, P16853.1, P16493.1, P17880.1.
файл Jalview с выравниванием вирусных белковМожно уверенно заявить, что все белки гомологичны. Участки высокой идентичности располагаются на протяжении всей последовательности, самый длинный из них: 366-499. Почти все колонки на 100% идентичны, либо функционально-консервативны. Инделей также очень немного.
Исследование Е-value
Для всех найденных белков E-value 0.00, то есть находки сверхдостоверны. Для этого задания выберем другой белок. Например, Glycoprotein_C того же вируса NYV.
При обоих вариантах поиска нашлось 22 белка. После череды E-value = 0.00, то есть 18 почти идентичных белков, идут 4 менее похожих. Например, oдин из них - A6XIP3.1 идентичен на 25.21% нашему. Без указания среди каких организмов искать E-value = 1e-04, с указанием Viruses (taxid:10239) - 4e-06.
результат поиска в BLAST без указания таксона результат поиска в BLAST с указанием Viruses (taxid:10239)E-value = Kmne-λS
При указании таксона мы изменили только n - размер базы данных, по которой ищем, m - длина исходной последовательности мы не меняли, как и S - вес, K и λ вообще константы. Отношениие n(+viruses) к n(standart) будет показателем додли вирусных белков в базе:
n (+viruses)/ n (standart) = 4*10-6 / 1*104 *100%= 4%
Поиск "гомологов" бессмысленной последовательности
Мы взяли фразу рождённого в Российской империи американского биолога-эволюциониста Феодосия Григорьевича Добржанского: " Nothing in Biology Makes Sense Except in the Light of Evolution "
После удаления пробелов и букв B, J, O, U, X, Z получилась последовательность: NTHINGINILGYMAKESSENSEECEPTINTHELIGHTFEVLTIN - 44 "аминокислоты" длиной.
текстовый файл результатов поиска по базе swissprot и E-value<10При поиске по базе swissprot нашлось 10 белков с E-value ниже хотя бы 10. Один из них - Q20168.3 Probable coatomer subunit beta идентичен на 100%, но на участке всего лишь 8 аминокислот, что составляет только 18% от длины последовательности.
Наибольшее покрытие выравнивания у человеческого белка Q8WZ42.4 Rhabdomyosarcoma antigen (Titin). Это получилось из-за огромной разницы в длинах последовательностей - у титина длина аж 34350, а у нашей случайной последовательности 44. Из-за этого она выровнялась в 37 местах (Number of Matches: 37), и получилась соответствующая разница между Max Score=28.6 и Total score=631. Следующий по проценту покрытия белок из аскомицета - A4RD09.2 Pentafunctional AROM polypeptide - 52%. К нему наша последовательность выровнялась в трёх местах тоже по 4-14 аминокислот. У этого же белка наибольший вес выравнивания - 32.9 и наименьший E-value - 0.38. По совокупности этих параметров можно было бы сделать вывод что это лучшая из находок, но опять же, всё так сложилось из-за большой разницы в длине - 1590 против 44. Просто вероятностно что-нибудь схожее на таких длинах найдётся
Если сопоставлять нашу бессмысленную последовательность со схожими по длине, то тут лучшей будет находка B0B898.1 Small ribosomal subunit protein uS19 длиной 88 аминокислот. E-value: 0.8 , Identities: 55% , длина выравнивания: 20, вес: 31.6. Собственно, 6 из 10 находок бласта это вариации этого белка из разных видов бактерий рода Chlamydia.