Предсказание генов прокариот
1. Сравнение предсказаний генов в базе данных GenBank и по данным Prodigal для плазмиды
О плазмиде. Эта плазмида
пренадлежит кишечной палочке (Escherichia coli).
Кишечная палочка - известный модельный организм. Считается, что это самый изученный
организм из всех. Можно предположить, что и плазмиды этой палочки изучены достаточно хорошо.
Ее размер 49467 пар нуклеотидов. В плазмиде 63 гена и все 63
из них CDS, то есть кодирующие белки. Плазмида представляет из себя кольцевую ДНК.
Используемые команды. Для скачивания таблицы с аннотациями использовал следующую
команду EMBOSS: seqret embl:CP014524 gff:plasmid.gff -feature. Что значит эта команда: в базе данных embl
найти файл с названием CP014524 сохранить в формате gff файла plasmid.gff с аннотациями.
Prodigal запускал командой prodigal.windows.exe -i plasmid.fasta -f sco -o plasmid.sco.
-i задает входной файл. -f формат выходного файла. -o название выходного файла. -p
изменяет алгоритм программы, есть два значения single и meta, по умолчанию стоит single.
С помощью python-скрипта производил
сравнивание выдачи Prodigal и GenBank. Заранее я сделал файлы с .gen и .pro, в которых
информация о кодирующих участах представлена сходным образом: начало, конец, направление.
К этим файлам я применял свой скрипт. Учитывая направление, алгоритм записывает в переменные
данные по N- и C- концам кодирующих фрагметов из GenBank. Дальше происходит поиск среди
всех строк файла с выдачей Prodigal. В 4 переменные счетчика сохраняют информацию по
соответствиям выдачи. Если сумма значений переменных дает 63 (количество CDS в аннотации
GenBank), значит все работает верно, это своего рода критерий правильной работы.
Для последовательностей с длиной меньше 100000 пн Prodigal предлагает
использовать алгоритм meta, но сравнение с GenBank показало, что single сработал лучше.
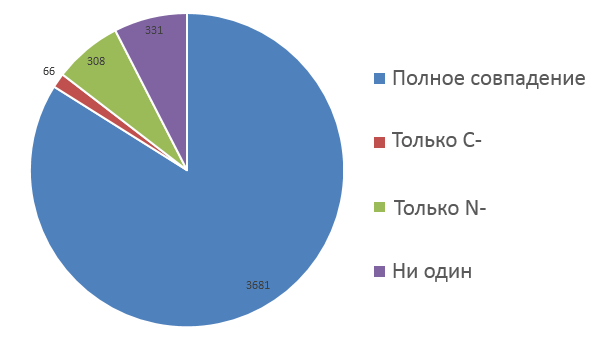
Посмотрим на диаграмах ниже сравнение аннотаций с предсказаниями. Синий цвет - все
совпало, красный - только C-конец, зеленый - только N-конец, фиолетовый - ничего не
совпало. Видим, что single дает более хорошие предсказания, чем meta.

Посмотрим два случая, когда предсказание не совпало с аннотацией.
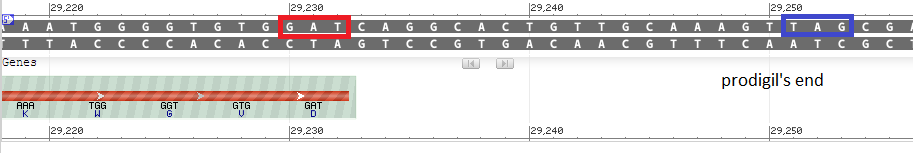
В первом случае не совпал C-конец. На приведенной ниже картинке видно, что стоп-кодоном
данного гена является GAT, Prodigal же закончил ближайшим каноничным стоп-кодоном (TAG), потому
что для работы мы ему предлагали стандартную таблицу генетического кода.
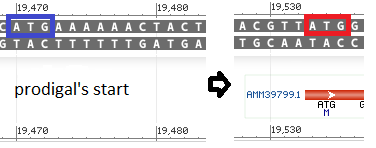
Во втором случае не совпал N-конец. И по предсказанию, и по аннотации
старт-кодоном является ATG-последовательность. Prodigal выбрал наиболее рано стоящий
вариант, что можно считать логичным. Картинка ниже иллюстрирует данный пример.
2. Сравнение предсказаний для геномной записи
Провел сравнение аннотации и предсказаний для своей бактерии
Deinococcus radiodurans R1. Результаты видны в диаграмме ниже. Общие тенденции сохраняются.

Кроме того сравнил выдачу Prodigal с GenBank для известного
модельного организма кишечной палочки. Здесь предсказания были самыми точными. Можно
предположить, что "настройку" Prodigal проводили на кишечной палочке, потому что ее
геном хорошо изучен. Но все прошлые тенденции сохраняются и тут.
Отметим общие тенденции и попробуем их объяснить. Для всех
рассмотренных нами случаев характерны следующие показатели. Процент полного совпадения
превышает 50% что говорит о том, что большая часть предсказаний верна и им можно
доверять. Количество верно предсказанных N-концов намного больше, чем С-концов. Это
связано с тем, что часто метионин кодируется не только ATG, и тем, что из нескольких
старт-кодонов он должен выбрать один, причем зачастую первый (мы видели это в примере).
В итоге, можно сказать, что Prodigal действительно можно использовать для предсказаний
белок-кодирующих участков.
P.S. В процессе выполнения у меня была небольшая путанница с
концами. Во всех местах, где написано про N- и C- концы имеется ввиду, что они
не совпадают.
P.P.S. Есть подозрение, что скрипт сработал не совсем верно, либо же проблема в разных аннотациях. В ближайшее время решу эту проблему с возможной заменой диаграмм.
P.P.S. Есть подозрение, что скрипт сработал не совсем верно, либо же проблема в разных аннотациях. В ближайшее время решу эту проблему с возможной заменой диаграмм.
© Maximov Vladislav, 2019.
