Секвенирование по Сэнгеру
Были даны 2 файла в формате .ab1 для
прямой
и обратной цепи ДНК.
Для прямой цепи длина нечитаемого участка в начале последовательности
составила 25 нуклеотидов, в конце - 49 нуклеотидов. Для обратной
цепи соответственно 21 и 24 (после перехода к комплементарной последовательности).
Сила сигнала практически не меняется при движении
вдоль последовательности, однако шум усиливается на краях
последовательности, а в середине он относительно слаб. Но если мы будем
сравнивать хроматограммы цепей друг с другом, то легко увидим, что
хроматограмма прямой цепи более качественная. Шумы даже на краях
читаемой области не превышают 1/5 силы сигнала (за очень редким
исключением), а в середине во многих
местах шумы вообще незаметны. Она имеет мало проблемных мест. В отличие
от нее, хроматограмма обратной цепи сделана хуже. Шумы хорошо заметны
даже в середине, где могут составлять до 1/6, редко 1/4 силы сингала.
По концам шумы ожидаемо усиливаются и могут составлять до половины силы
силнала. На концах многократно увеличивается количество проблемных
мест, возникают несуществующие пики. Суммарное количество проблемных
мест многократно превышает количество таковых в прямой цепи.
Разбор 4 проблемных случаев:
| 1. Здесь и далее будем рассматривать проблемные места только в прямой цепи. Проблемное место выделено красным прямоугольником, его аналог в обратной цепи также выделен красным прямоугольником. Видим небольшой зеленый пик примерно на одинаковом удалении от соседних "хороших" пиков. Под этим пиком видим чуть смещенный черный пик. Проверяем по обратной цепи. В этом месте не видим никакого черного пика, только относительно сильный синий "шум", значит, не полиморфизм, а просто аденин. | 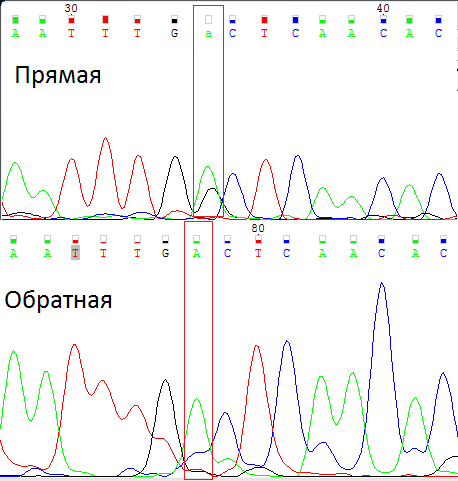 |
| 2. Зеленый пик, эквивалентный по силе соседним хорошим зеленым пикам и равноудаленный от них, имеет смещенный черный пик в половину "зеленого" сигнала. Смотрим на обратную цепь. до сих пор видим черный "шум", но его сила не превышает 1/8 силы сигнала. Делаем вывод, что это просто аденин. |  |
| 3. Синий пик равноудален от соседних пиков. Черный "шум" составляет примерно половину силы сигнала, но соседних черные пики многократно превышают этот шум. Смотрим на обратную цепь. Шумы у синего пика здесь не превышают четверти силы сигнала. Шум "становится" красным. Делаем вывод, что это цитозин. | 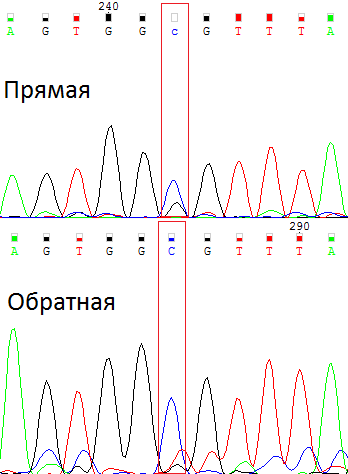 |
| 4. Этот случай очень наглядно демонстрирует важность секвенирования обоих цепей. В одном месте у цепей есть ошибки, но разного рода. Рассмотрим вначале прямую цепь. Черный пик еле превышает минимум зеленого сигнала соседних пиков, хотя примерно равноудален от них. Находим на комплементарной цепи этот кусочек. Видим добротный черный пик, но правее него между пиками вклинились сильные шумы, на которых даже намека не было на прямой цепи. Делаем вывод, что на прямой цепи у нас гуанин, а на обратной - нет никакого нуклеотида между G и A (правее G). |  |
Полученный исправленные последовательности
прямой
и обратной цепи
сохранил в формате fasta. Создал
проект
jalview, где произвел выравнивание прямой и обратной
последовательности. А проблемные места отмечал на дорожке снизу.
Причем если проблемной место в прямой цепи ставил lable - S,
в обратной - R.
Разбор нечитаемого фрагмента:

Программа выдает какие-то осмысленные
результаты, хотя их достоверность вызывает сильные сомнения.
Расстояния между пиками сильно варьируют, "шумы" по силе
часто эквавалентны "сигналу". Хотя некоторые пики и сильно
выделяются на фоне шумов, но в целом весь фрагмент не вызывает
доверия. Его научная ценность стремится к нулю.
© Maximov Vladislav, 2019.
