Нуклеотидный BLAST
1. Определение таксономии и функции прочтенной нуклеотидной последовательности из практикума 6
В ходе выполнения практикума 6 была получена
последовательность (прямая цепь).
Запустил megaBLAST, задав ему эту данную последовательность. Первые несколько
результатов указаны ниже.
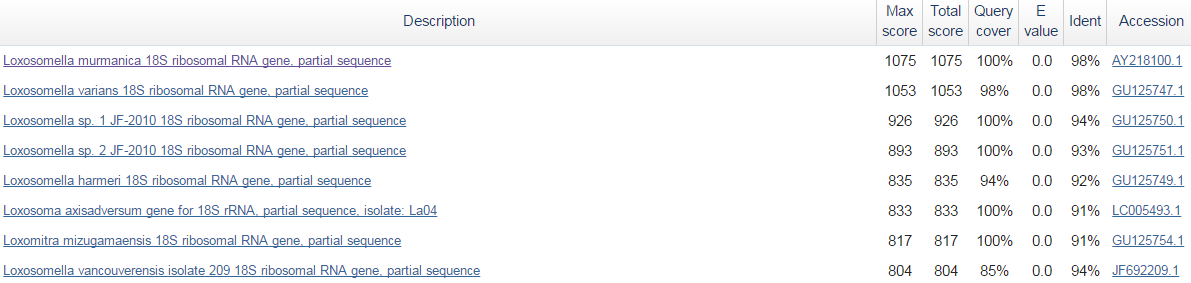
Данные с высокой долей вероятности говорят, что наша последовательность
кодирует рибосомальную РНК 18S субъединицы. С указанной ниже таксономией:
- Eukaryota
- Metazoa
- Lophotrochozoa
- Entoprocta
- Loxosomatidae
- Loxosomella
- Loxosomella murmanica
Чтобы убедиться в правильности выдачи BLAST и правильном выборе организма, решил
провести выравнивание вручную.
Скачал 5 последовательностей из 15 лучших находок. Сделал их выравнивание с исходной
с помощью алгоритма muscle в jalview. Кусок
выравнивания
из проекта
jalview представлен ниже.
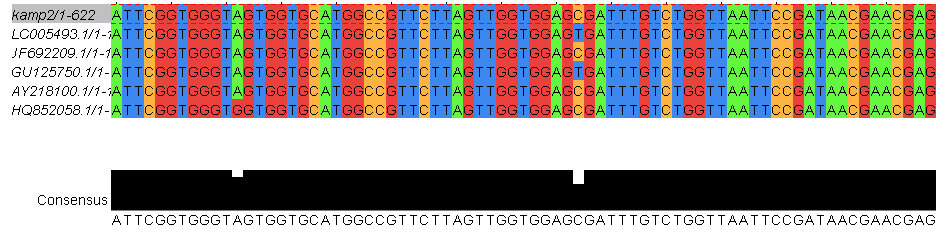
Исходная последовательность сверху. Видим большие участки сходства
в середине выравнивания. Сходство последовательностей очень велико.
2.Сравнение списка находок нуклеотидной последовательности тремя разными алгоритмами BLAST
Запустим выравнивание нашей последовательности тремя различными
алгоритмами BLAST: BLASTn, discontiguous megaBLAST, megaBLAST. Со следующими общими
параметрами:
-максимальное количество последовательностей: 1000
-база данных: Nucleotide collection
-поиск только среди: царства Metazoa
-исключая: семейство Loxosomatidae
-максимальное количество последовательностей: 1000
-база данных: Nucleotide collection
-поиск только среди: царства Metazoa
-исключая: семейство Loxosomatidae
Все остальные параметры - типичные для алгоритмов. Все данные
по алгоритмам внесены в таблицу 1.
| Таблица 1. Сравнение алгоритмов BLAST | |||||
| Алгоритм | Количество находок (из 1000) | Лучший счет | Хучший счет | Лучшее сходство, % | Хучшее сходство, % |
| BLASTn | 1000 | 821 | 740 | 90 | 87 |
| disc. megaBLAST | 1000 | 821 | 742 | 90 | 87 |
| megaBLAST | 1000 | 798 | 699 | 90 | 87 |
E-value и степень покрытия не менялись и составляли соотвественно
0 и 99% для всех 1000 лучших находок для всех алгоритмов. Но мы видим, что
megaBLAST не нашел много последовательностей с хорошим счетом: его лучший score меньше,
чем у остальных алгоритмов. Из-за этого и хучшая находка имеет меньший счет, т.к.
не было найдено много хороших последовательностей. Разница между BLASTn и
disc. megaBLAST небольшая, наименьший счет у BLASTn выше, следовательно
disc. megaBLAST нашел одну или несколько последовательностей с хорошим счетом. Он
более лояльно подходит к поиску последовательностей, но длина слова у него такая же
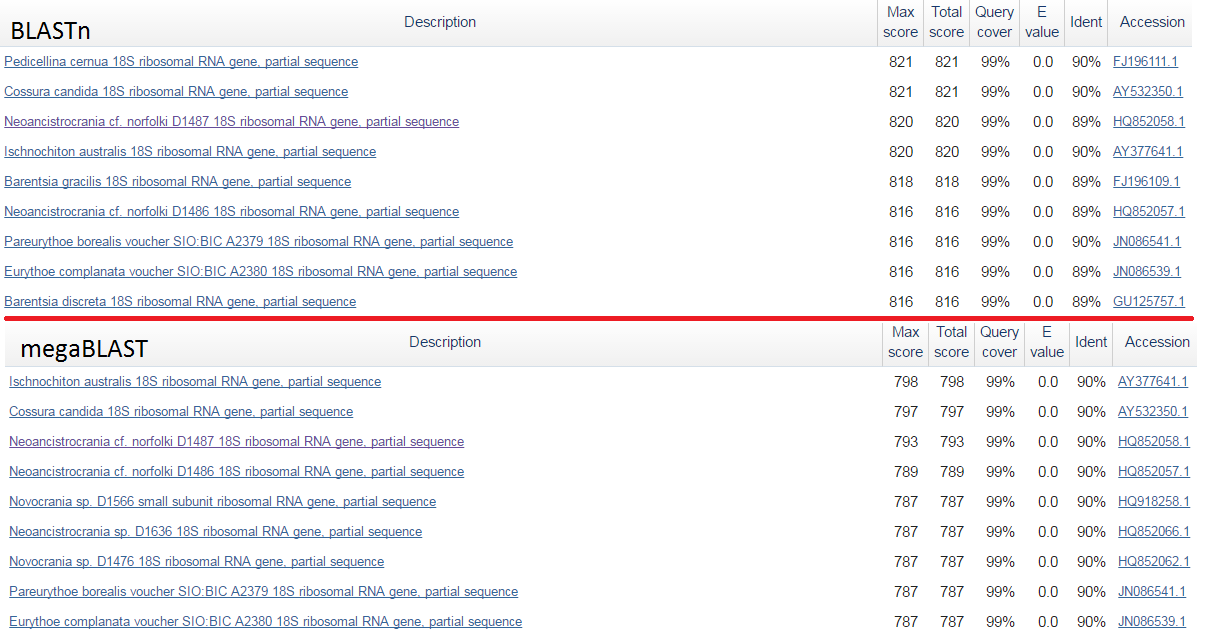
как и у BLASTn. Посмотрим различные лучшие выдачи BLASTn и megaBLAST на картинке ниже.
Видим различные находки в топе этих алгоритмов, точнее BLASTn нашел последовательности,
которые не нашел megaBLAST.
3.Поиск гомологов трех белков в геноме одного организма
Для поиска гомологов пользуемся алгоритмом tblastn,
установленным на сервере. Наш организм - Amoboaphelidium protococarum,
сборка генома X5. E-value я ограничил 0.0001, чтобы заведомо убрать
нерепрезентативные выравнивания. Все данные по выравнивание в
Excel-таблице.
Начнем с белка теплового шока, шаперона HSP7C_HUMAN. Его
длина 646 аминокислот. Лучшее локальное выравнивание имеет длину 607 аминокислот
(94% покрытие),
78% сходство и 0 e-value. Я считаю, что это гомолог человеческого шаперона.
Шапероны - белки, главная функция которых - восстановление правильной третичной или
четвертичной структуры белков. Они есть почти во всех живых организмах, механизм
их действия также консервативен. Многие шапероны являются белками теплового шока, их
экспрессия начинается в ответ на рост температуры или другие стрессы.
Второй белок - белок, связывающий поли(А) хвост мРНК, PABP2_HUMAN.
Его длина 306 аминокислот. Покрытие лучшей находки составляет 28%, совпадение - 63%,
e-value - 2Е-28. Маловероятно, что это простое совпадение. Возможно это гомолог
какого-то домена нашего белка. Для всех эукариот характерно полиаденилирование,
поэтому наличие белков ответственных за этот процесс является критичным.
Но что касается конкретных белков этот белкового комплекса, то они не обязательно
должны быть консервативными, напротив какие-то их части могут отличаться у разных таксонов.
Третий белок - часть ДНК-зависимой РНК-полимеразы 2. RPB1_HUMAN имеет
длину 1970 аминокислот. Лучшая находка длиной 1547 (79% покрытие) имеет сходство 52%,
e-value - 0. Есть еще одна находка меньшей длины и с сходством 53%. Скорее всего
это гомологи исходного белка. У эукариот три типа ДНК-зависимых РНК-полимераз. Наш белок
субъединица второй.
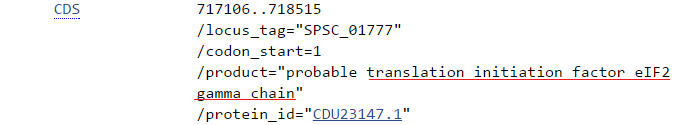
4.Поиск гена белка, закодированного в одном скэффолде организма
Взял один из
скэффолдов
организма. Запустил BLASTn. Поиск дал множество последовательностей схожей длины,
покрытие 5%.
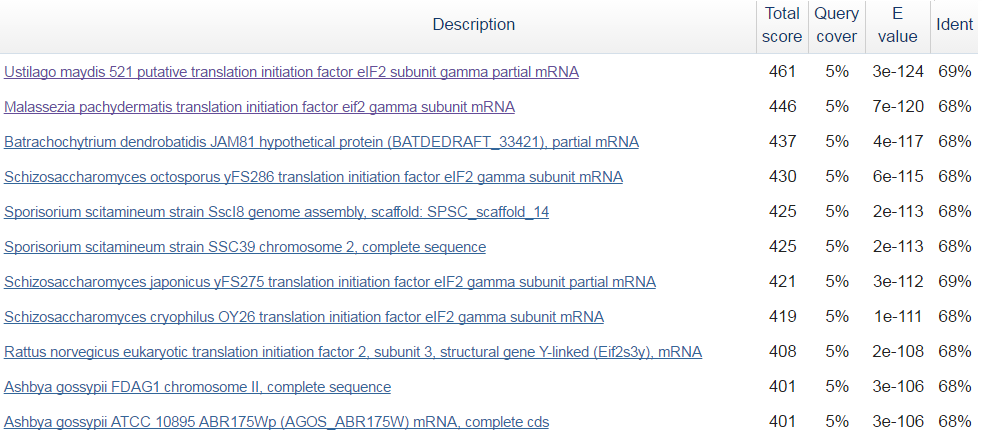
Удалось выяснить, что этот ген, вероятнее всего, кодирует гамма
цепь фактора инициации трансляции eIF2.
Факторы инициации трансляции - белки, обеспечивающие инициацию
трансляции, то есть синтеза полипептидной цепи на рибосомах.
© Maximov Vladislav, 2019.
