Определение сайта связывания транскрипционного фактора
В работе анализировался файл chipseq_chunk21.fastq. Файл содержит риды, полученные в ходе Chip-seq эксперимента. Для начала провели проверку качества чтений при помощи программы FastQC, при помощи команды (fastqc chipseq_chunk21.fastq). Базовую статистику можно видеть на картинке ниже.

Остальная статистика доступна по ссылке. Мы видим очень хорошее качество чтений. Какие-то отклонения в GC-контенте и частоте оснований можно объяснить особенностями нашего NGS эксперимента, мы ищем сайты связывания фактора, а не проводим полногеномное секвенирование. Следовательно, фильтрация данных не необходима.
Риды были картированы на проиндексированный геном при помощи команды (bwa mem ../../hg19/GRCh37.p13.genome.fa chipseq_chunk21.fastq > chipseq_chunk21.sam). Выясним, сколько ридов картировались на геном и на какие хромосомы. Для этого использовали пакет samtools:
samtools view -bSo chipseq_chunk21.bam chipseq_chunk21.sam - получаем бинарную версию нашего файла;
samtools sort chipseq_chunkX.bam -T chip_temp -o chipseq_chunkX.sorted.bam - сортируем риды по координате начала рида в референсе;
samtools index chipseq_chunk21.sorted.bam - индексируем;
samtools idxstats chipseq_chunk21.sorted.bam > chipseq_chunk21.idxstats - получение информации о количестве чтений на каждой хромосоме.
samtools view -c chipseq_chunk21.sorted.bam - показывает общее количество картированных ридов
samtools view -bSo chipseq_chunk21.bam chipseq_chunk21.sam - получаем бинарную версию нашего файла;
samtools sort chipseq_chunkX.bam -T chip_temp -o chipseq_chunkX.sorted.bam - сортируем риды по координате начала рида в референсе;
samtools index chipseq_chunk21.sorted.bam - индексируем;
samtools idxstats chipseq_chunk21.sorted.bam > chipseq_chunk21.idxstats - получение информации о количестве чтений на каждой хромосоме.
samtools view -c chipseq_chunk21.sorted.bam - показывает общее количество картированных ридов
Общее количество ридов - 4305. Из них 4305 были картированы. Из них 3999 - на 9 хромосому. Более подробная информация - в файле. Для поиска пиков воспользуемся MACS2 с командой вызова (macs2 callpeak -t chipseq_chunk21.sorted.bam --nomodel --ourdir chr9). Количества пиков для построения модели было недостаточно, поэтому использовался параметр --nomodel. --ourdir - название директории, содержащей выходные данные. Название было дано по исследуемой хромосоме. Было обнаружено три пика в 9 хромосоме. Информация о них на картинке ниже.
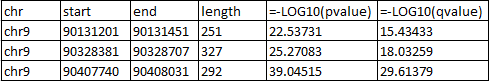
Последние 2 столбца показывают отрицательный десятичный логарифм p-value и q-value. Таким образом, чем больше это число, тем достовернее информация о пике. Пики визуализировали с помощтю Genome Browser (на картинке ниже).

Рассмотрим первый пик. Его ширина - 154, вершина отстоит от начала на 140, то есть ближе к левому краю пика. Пик имеет высокую достоверность и пересекается с геном DARK1. Это ген, кодирующий связанную со смертью протеинкиназу 1. Пересечение проходит в теле гена, однако, недалеко от старта. Картинка ниже.
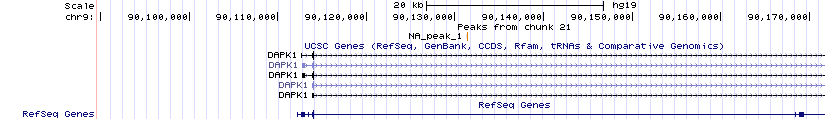
Рассмотрим второй пик. Его ширина - 180, вершина на расстоянии 140 от начала. У пика хорошая достоверность. Пик стоит между геном DARK1 и геном CTSL1. Второй ген тоже протеин кодирующий. Но пик ближе к концу гена DARK1, чем к началу CTSL1, и отстоит от него на приличном расстоянии.
© Maximov Vladislav, 2019.
