Выбор пары ортологических рядов
В KEGG открыл метаболизм аланина, аспартата и глутамина. В этом
метаболизме выбрал реакцию L-Alanine -> Pyruvat (EC:2.6.1.44). На картинке ниже эта реакция выделена
на указанном метаболизме черным фоном.
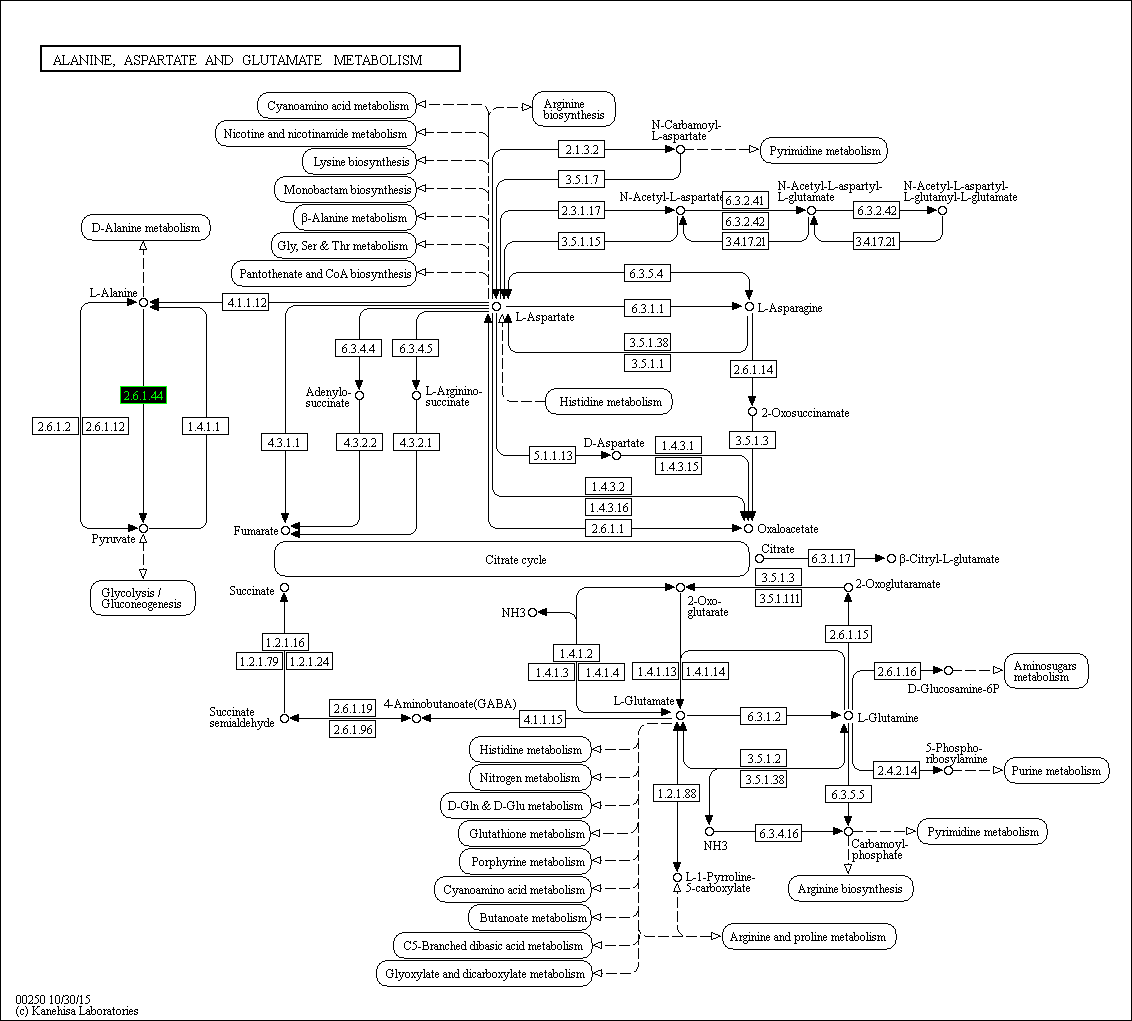
Общее число
ортологических рядов реакции - 3.
Для дальнейшей работы отобрал 2 их них:
K00827 с 147 белками
и
K14272 с 49 последовательностями в Uniprot.
Третий ряд содержит больше 700 последовательностей.
Эти ферменты проводят реакцию: L-Alanine + Glyoxylate <=> Pyruvate + Glycine (картинка ниже)

Построение совместного множественного выравнивания
С помощью сервиса "Retrieve/ID mapping" БД Uniprot получил последовательности
нужных мне белков в fasta-формате. Теперь
скриптом изменил названия всех
последовательностей таким образом, что в идентификаторе содержится информация о ряде, из которого был взят белок.
Полученный
файл был использован для построения множественного выравнивания.
Выравнивание было произведено сервисом "Muscle with Default",
проект этого выравнивания.
Проверка гомологичности белков в выравнивании
В выравнивании есть последовательности которые плохо выравнены с остальными или слишком коротки.
Все подобные последовательности были удалены. Они встречались в обоих рядах, однако в ряду K00827 таких было
намного больше, даже учитывая, что последовательностей там было изначально больше. После удаления плохо выровненных
последовательностей образовались блоки, состоящие только из гэпов. Они были убраны. Итоговая версия
проекта. Посмотрим, являются ли ряды гомологичными. Видна хорошая гомология внутри
каждого из рядов, но нет ни одного блока, охватывающего все последовательности. "Границу" между рядами
в выравнивании можно легко увидеть, даже не смотря на идентификаторы, т.к. разница между ними очень велика.
Пример на картинке ниже.

Деревья
На мой взгляд, дерево по данному выравниванию строить нельзя, т.к. выравнивания
как такового нет (см. прошлый пункт). Если бы мы все-таки решили построить дерево по этим данным,
то заметили бы строгое разделение на 2 группы, каждая из которых представляет собой ортологический ряд.
Точно такое же дерево мы бы увидили, если бы выравнивали 2 группы белков, которые между собой не
гомологичны, но внутри каждой группы есть хорошая гомология.
Отсутствие гомологии между рядами может говорить о том, что нет специализированного активного центра
для данной реакции/веществ, либо, что один из рядов специализируется больше на другой реакции, но может
катализировать и данную. Можно заметить, что ряд K14272 обладает только glutamate-glyoxylate aminotransferase
активностью (обеспечивает реакции EC:2.6.1.4 2.6.1.2 2.6.1.44), а ряд K00827 - alanine-glyoxylate transaminase и
(R)-3-amino-2-methylpropionate-pyruvate transaminase активностями (обеспечивает реакции
EC:2.6.1.44 2.6.1.40). Видим пересечение только по одному типу реакций, а по типу фермента пересечений вообще нет.
Соответственно, вторая гипотеза становится более вероятной.



