Геномное окружение. База данных GO
1. Получение информации о КОГе, к которому относится белок NP_295013
Белок описан на NCBI как RecQ ДНК хеликаза.
С помощью сервиса CDD и базы данных КОГов выбрал КОГ, к которому по моему мнению относится мой белок. Как и ожидалось нашлось несколько хитов (совпадений) (показано на рис.1). Диапазон e-value составляет от 0 до 8е-03, интервал перекрытия от нескольких десятков до 600 (у лучшего), при длине белка 824 аминокислоты.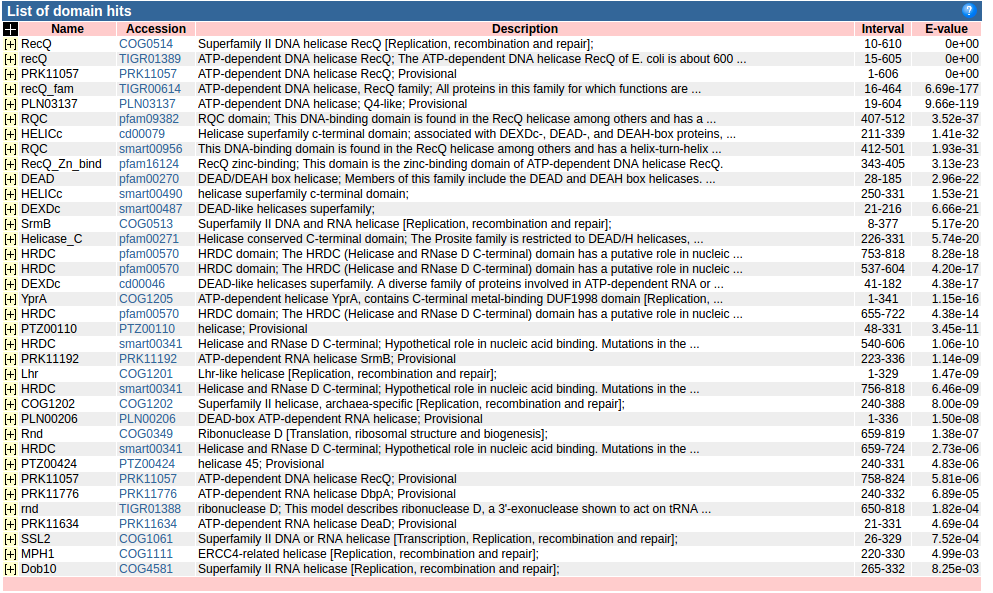
Идентификатор выбранного КОГа -
COG0514.
Его e-value = 0, КОГ встречается в белке с 10 по 610 аминокислоту (из 824). Название КОГа -
Superfamily II DNA helicase RecQ (Суперсемейство II ДНК-геликазы RecQ), COG0514, категория -
L:
Replication, recombination and repair (репликация, рекомбинация и репарация).
2. Визуализация геномного окружения
Пользовался сервисом
STRING,
идентификатор нашего белка DR_1289. Выдача представляет собой
граф взаимодействий,
где узлы - белки (большие - с известной 3D структурой, маленькие - с неизвестной),
стороны - различные взаимодействия (каждый цвет обозначает определенный тип взаимодействий).
Параметры:
- minimum required interaction score: 0.7;
- max number of interactors to show: 10 по первой оболочке, по второй - 0. На рис.2 показан изначальный граф со всеми видами взаимодействий, он загружен информацией, и понять что-либо очень сложно. Поэтому были выключены те взаимодействия, в которые наш белок не вступает: gene fusions и co-occurrence. На рис.3 (под рис.2) видим упрощенный граф.
- minimum required interaction score: 0.7;
- max number of interactors to show: 10 по первой оболочке, по второй - 0. На рис.2 показан изначальный граф со всеми видами взаимодействий, он загружен информацией, и понять что-либо очень сложно. Поэтому были выключены те взаимодействия, в которые наш белок не вступает: gene fusions и co-occurrence. На рис.3 (под рис.2) видим упрощенный граф.
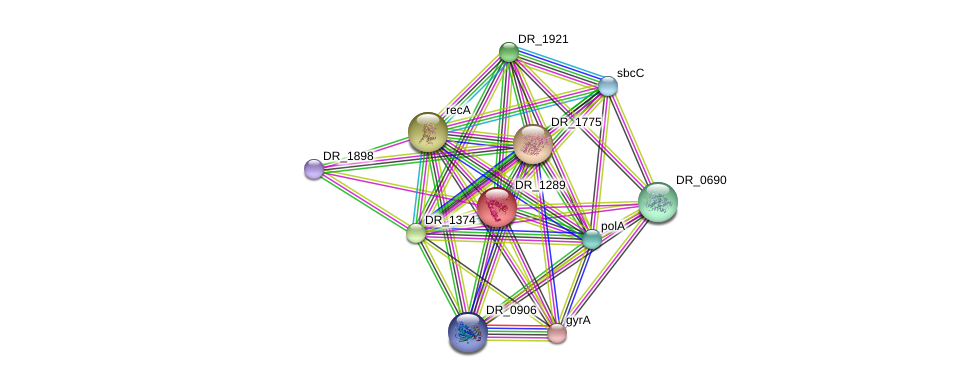
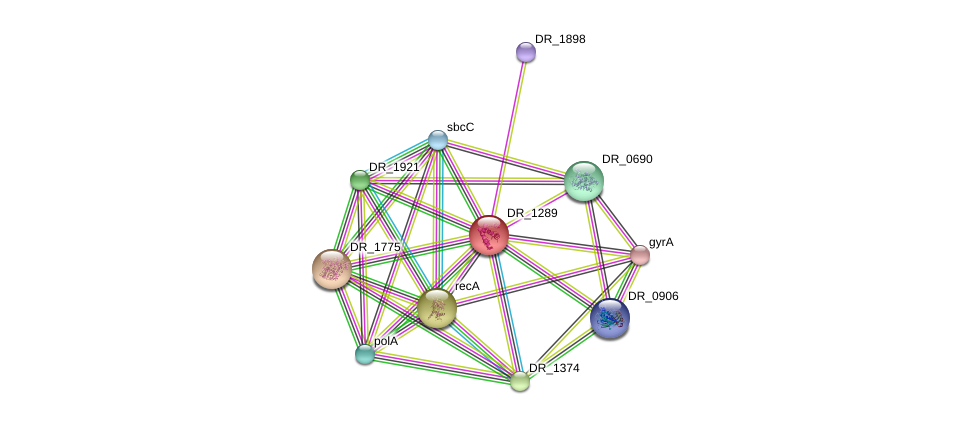
Из графа видно, что белок имеет большое количество как предсказанных, так и
экспериментально доказанных (что особенно важно) взаимодействий. Так же из таблицы на рис.4 видно, что
все(!) взаимодействия с 10 другими белками экспериментально подтверждены и имеют высокую достоверность.

Рассмотрим некоторые белки из окружения нашего. Например, рекомбиназа А, эндонуклеаза,
топоизомераза 1 типа, гиразы (относятся к группе топоизомераз). Все они участвуют в функциях категории
нашего КОГа: репликации, рекомбинации и репарации.
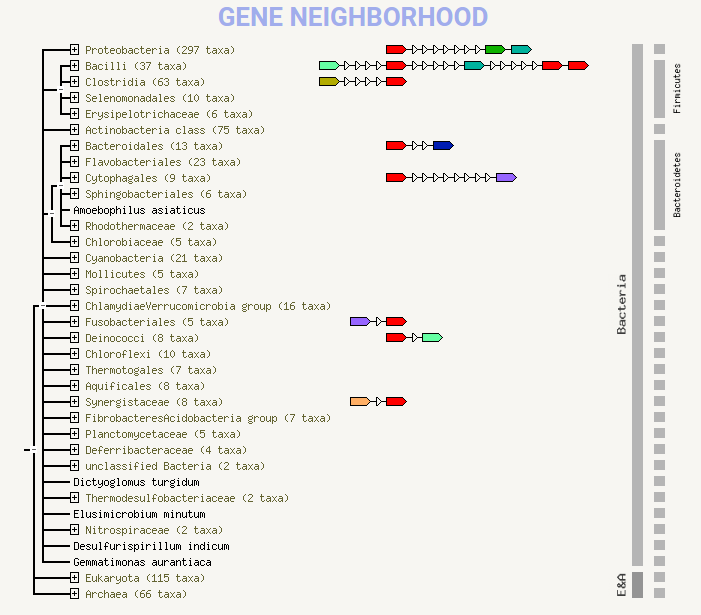
На картинке ниже отображено геномное окружение
нашего КОГа. Никаких закономерностей и паттернов не обнаружено.
3. Отнесение белка RecQ ДНК хеликазы из Deinococcus radiodurans R1 к терминам GO
Использовал поиск по fasta-последовательности белка сервиса
AmiGO.
Лучшая находка - ATP-dependent DNA helicase RecQ из Geobacter sulfurreducens PCA с e-value = 1.0e-167,
identities = 320/605 (52%), positives = 405/605 (66%). Из чего можно сделать вывод, что это не наш
белок, но очень близкий гомолог. Этот белок выполняет ту же функцию, что и наш, так что без проблем
будем использовать его в дальнейшем. Его идентификатор UniProt - Q74ER2. Он имеет
2 ассоциации
В таблице 1 указаны термины GO, отнесенный к нашему белку. В таблице 2 - описание встреченный кодов
типа достоверности.
| Таблица 1. Термины GO, отнесенные к белку с идентификатором UniProt Q74ER2. | ||||
|---|---|---|---|---|
| Аспект | Идентификатор GO | Название термина | Перевод названия термина | Код типа достоверности |
| biological process | GO:0006310 | DNA recombination | Рекомбинация ДНК | ISS |
| molecular function | GO:0004003 | ATP-dependent DNA helicase activity | АТФ-зависимая активность ДНК геликазы | ISS |
| Таблица 2. Описание кодов достоверности, использованных в Таблице 1. | ||
|---|---|---|
| Код типа достоверности | Расшифровка кода типа достоверности | Объяснение |
| ISS | Inferred from Sequence or Structural Similarity | Данный код присваивается в случае, если анализ был основан на последовательностях и обзор доказательств и аннотаций делается вручную. Так же может быть использован для структурного сходства с экспериментально охарактеризованными продуктами гена. |
Существует глобальный проект gene ontology, целью которого является систематизация
наших данных о белках и последовательностях. В этом практикуме мы знакомились с работой этого
механизма. Вначале мы искали КОГ, к которому относится наш белок. Мы заведомо знали функцию нашего
белка, поэтому не было ничего удивительного в том, что КОГ представлял собой суперсемейство
геликаз (наш белок - геликаза). Но если бы мы ничего не знали о белке, то эта информация была бы
ценной.
Далее с помощью STRING посмотрели связи нашего КОГа с другими белками. В выдаче STRING находятся только те белки, с которыми есть предполагаемое или доказанное взаимодействие, причем, мы можем знать, насколько достоверно данное взаимодействие.
В конце, с помощью сервиса AmiGO нашли ближайший гомолог нашего белка с терминами GO. К сожалению, это не был именно наш белок, но его гомология не вызывает сомнений и мы могли перенести термины GO с того белка на наш.
Gene ontology мощный механизм, позволяющий классифицировать белки по различным параметрам, либо узнавать свойства новых белков,
Далее с помощью STRING посмотрели связи нашего КОГа с другими белками. В выдаче STRING находятся только те белки, с которыми есть предполагаемое или доказанное взаимодействие, причем, мы можем знать, насколько достоверно данное взаимодействие.
В конце, с помощью сервиса AmiGO нашли ближайший гомолог нашего белка с терминами GO. К сожалению, это не был именно наш белок, но его гомология не вызывает сомнений и мы могли перенести термины GO с того белка на наш.
Gene ontology мощный механизм, позволяющий классифицировать белки по различным параметрам, либо узнавать свойства новых белков,
© Maximov Vladislav, 2019.
