Поиск сигналов. Часть 2.
Первая часть практикума доступна по ссылке. Программой featcopy были извлечены координаты генов в геноме Bacillus pumilus и по ним восстановлены координаты upstream regions наших находок. Использовали находки с лучшими p-value, так как q-value одинаковые. Эти мотивы находятся рядом с генами NP_375139.1 и NP_373827.1. Для дальнейшей работы необходим файл с известными последовательностями сайтов метилирования и последовательностью первого и второго мотива с окружением в 50 нуклеотидов, чтобы учесть пересечения с сайтами больше длины мотива. Далее использовали fuzznuc:
fuzznuc -sequence mX.fasta -pattern @MT_sites.txt -outfile fuzznucX.out
fuzznuc -sequence mX.fasta -pattern @MT_sites.txt -outfile fuzznucX.out
Для первого мотива было обнаружено 45 находок находок, для второго - 49. Основную часть составляют четырехбуквенные находки. Но есть и 7-буквенные, без N посередине или на концах, например, CAAAAAR или AAAAGRG.
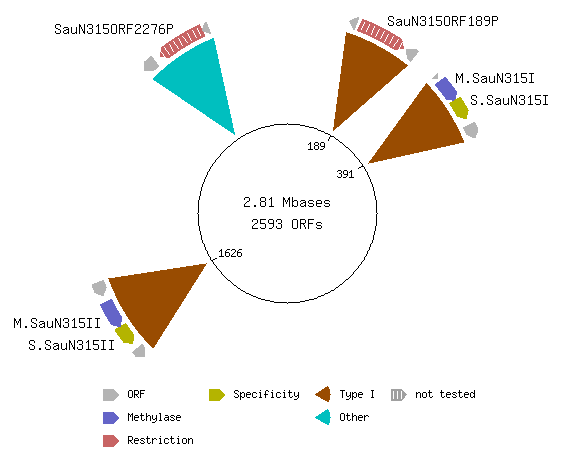
Чтобы проверить есть ли в геноме нашей бактерии ДНК метилтрансферазы с той же специфичностью, что у тех, для которых были найдены пересечения с мотивами, геном Staphylococcus aureus subsp. aureus N315 был найден в базе данных REBASE и была получена информация о системе рестрикции-модификации данной бактерии. Как видно на картинке ниже, было найдено 4 ДНК метилтрансферазы. 3 из них относятся к 1 типу, что понятно из расшифровки снизу картинки. И еще одна - к 4 типу.

© Maximov Vladislav, 2019.
