Предсказание вторичной структуры заданной тРНК и анализ НК-белкового комплекса
Предсказание вторичной структуры заданной тРНК
В результате применения команды
einverted -sequence trna.fasta -gap 12 -threshold 10 -match 3 -mismatch -3 -stdout
были предсказаны пары:
01: Score 15: 7/9 ( 77%) matches, 0 gaps
1 ggccccatc 9
|||| | ||
31 ccggcgcag 23
EMBOSS_001: Score 15: 10/15 ( 66%) matches, 0 gaps
34 ctcaaggccgaaacg 48
| | ||| ||| ||
69 ggggtcccccttagc 55
Такие пары не могут соответствовать стеблям тРНК. Получить адекватный результат подгонкой параметров не удалось.
Предсказание вторичной структуры тРНК алгоритмом Зукера
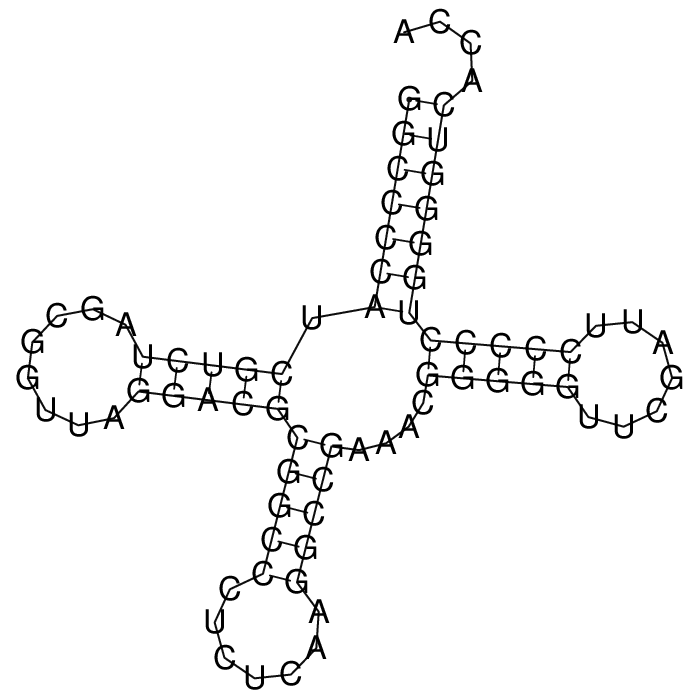
| Позиции в структуре(find pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера | |
| Акцепторный стебель | 1-7, 66-72 | - | 1-7, 65-71 |
| D-стебель | 10-14, 21-25 | - | 9-13, 22-26 |
| Антикодоновый стебель | 26-32, 38-44 | - | 27-31, 39-43 |
| T-стебель | 49-53, 61-65 | - | 48-52, 60-64 |
| Общее число канонических пар нуклеотидов | 24 | 24 | 24 |
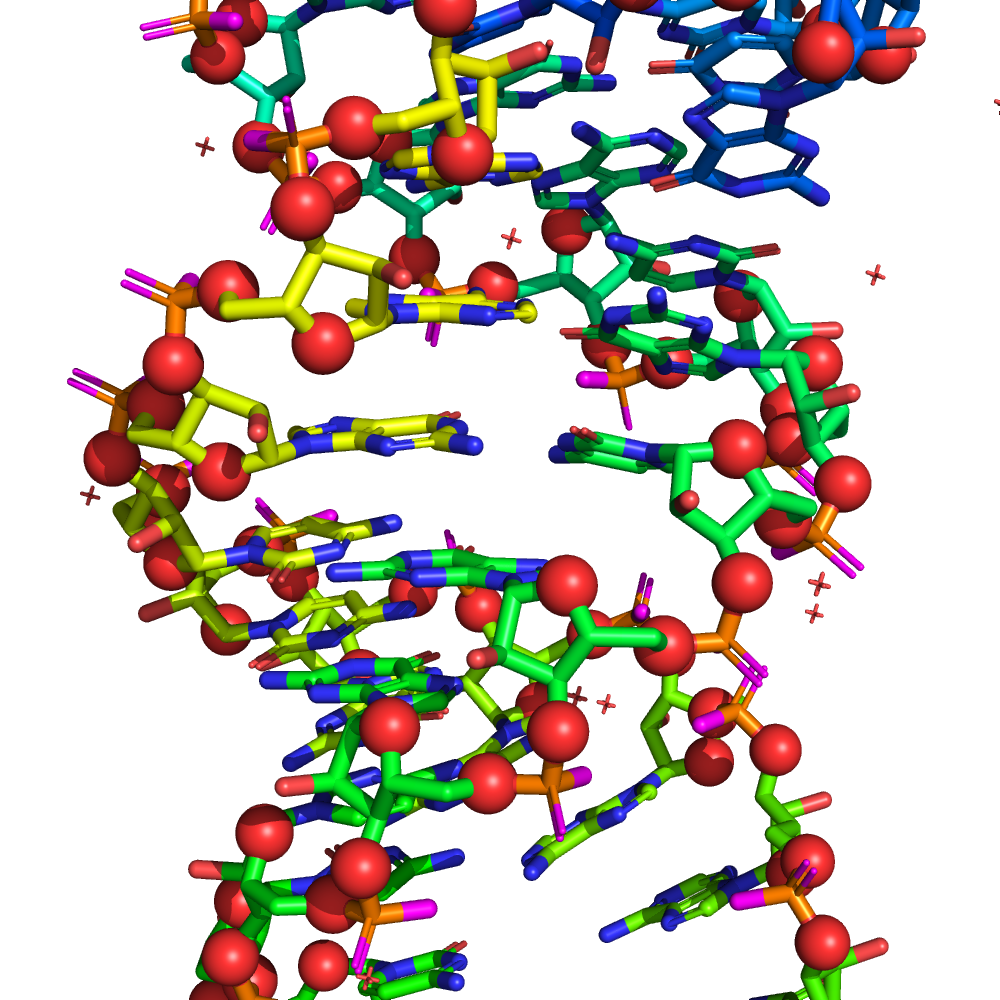
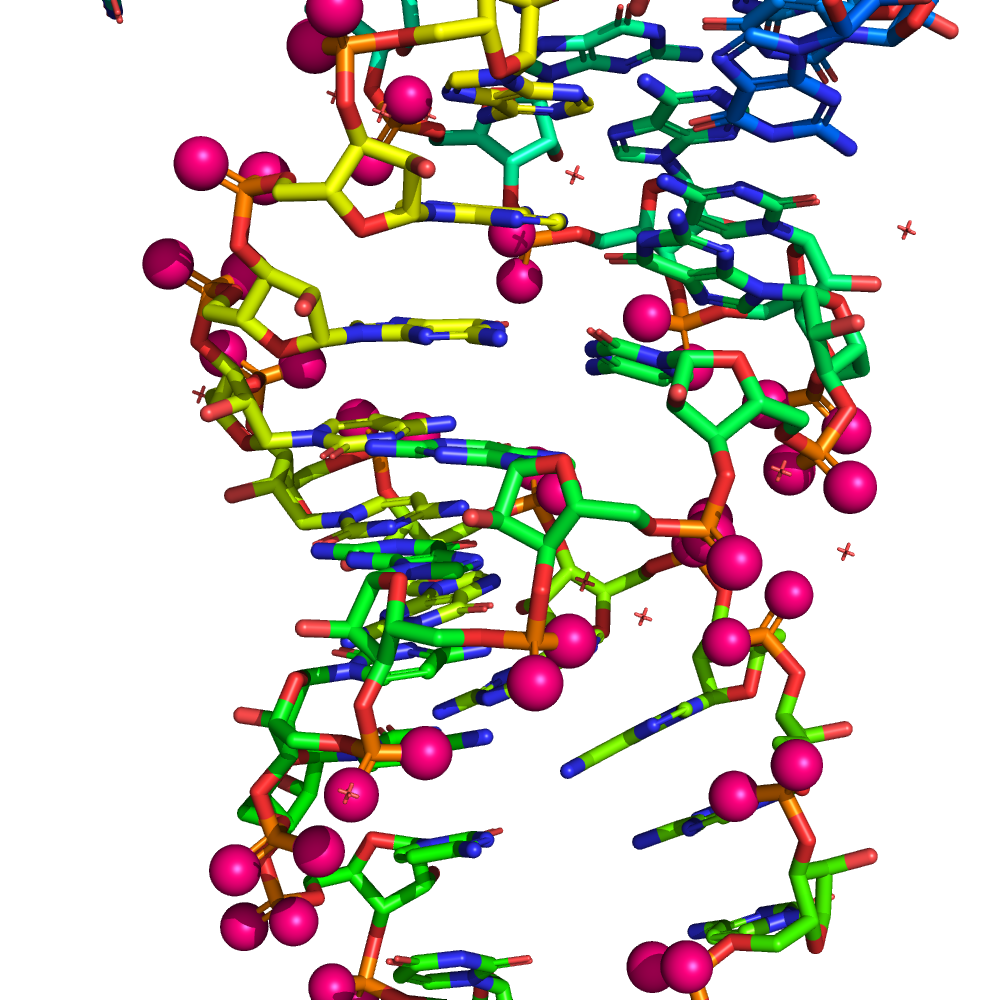
Поиск РНК-белковых контактов в заданной структуре
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-рибозы | 8 | 54 | 62 |
| остатками фосфорной кислоты | 13 | 11 | 24 |
| остатками азотистых оснований со стороны большой бороздки | 1 | 8 | 9 |
| остатками азотистых оснований со стороны малой бороздки | 4 | 7 | 11 |
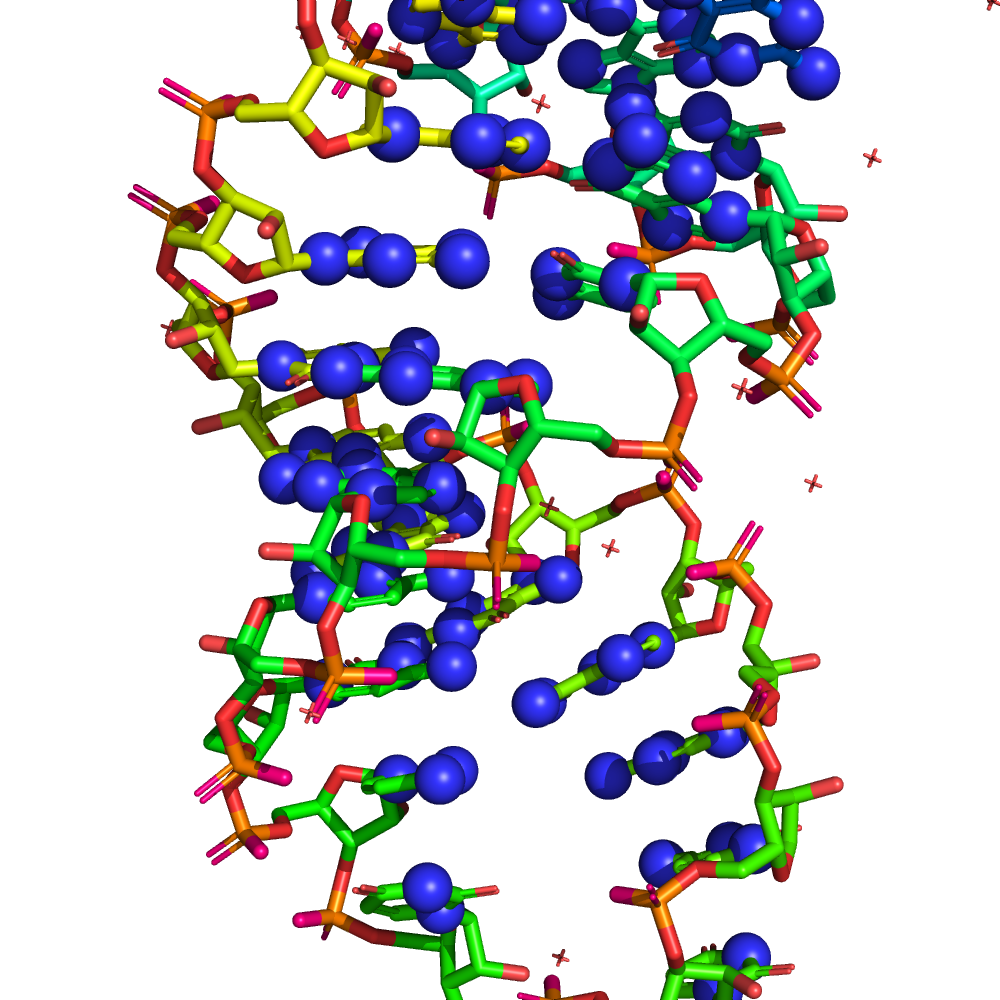
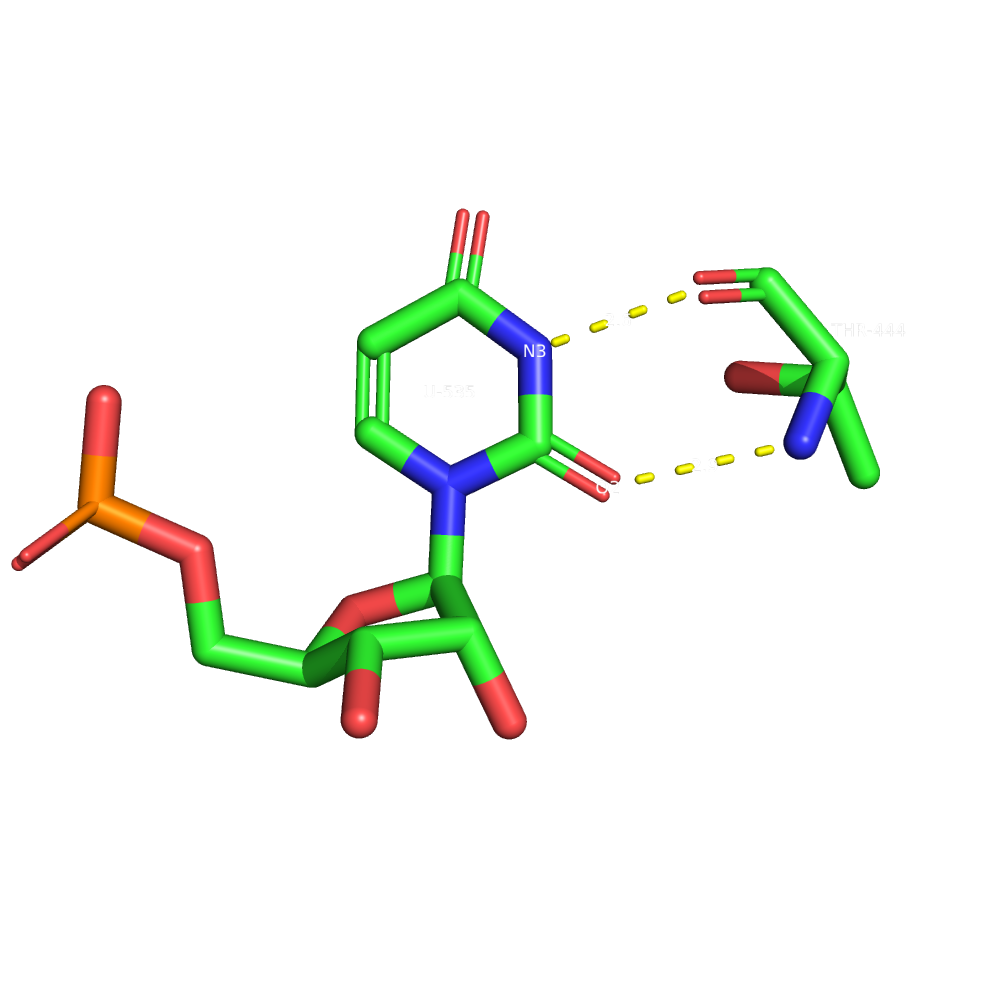
Визуализация аминокислотных остатков
Остатки выбирались вблизи антикодоновой петли - по ней(в том числе) идет узнавание тРНК.
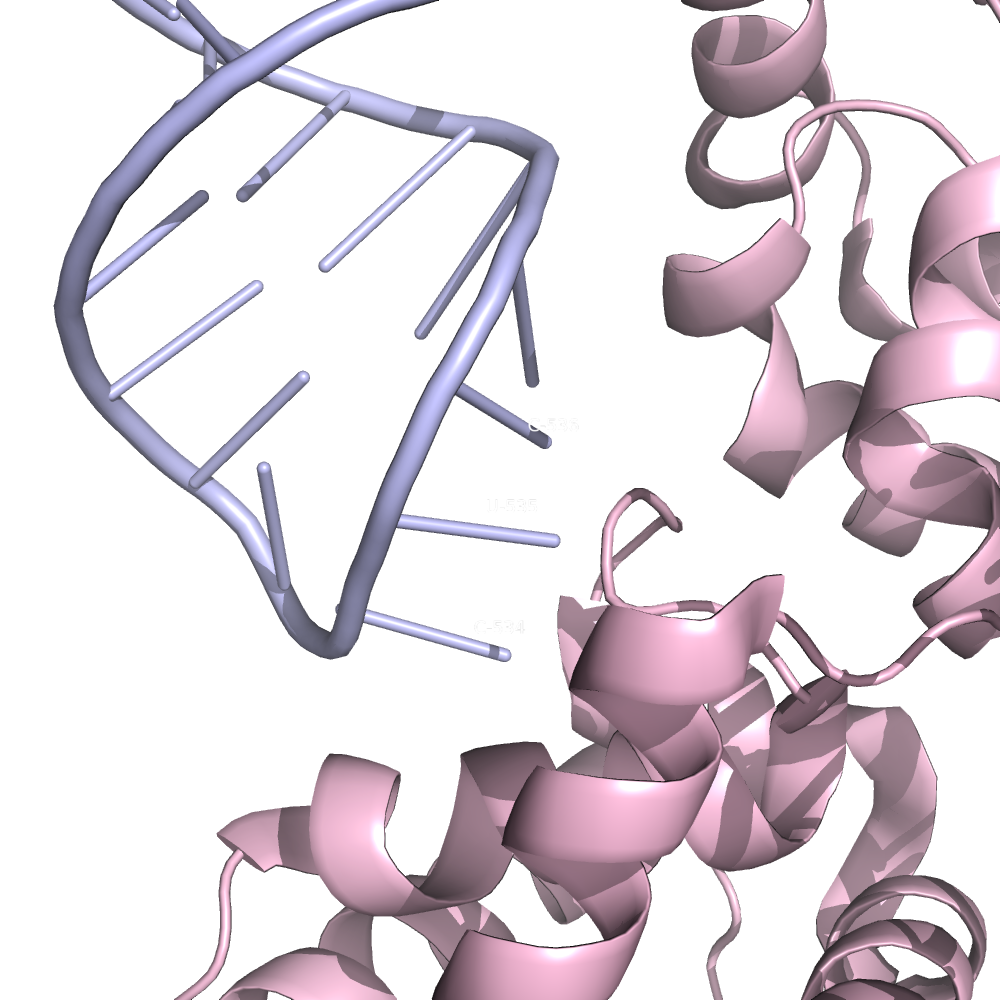
Как остаток с наибольшим числом связей был выбран глутамин 432 - исходя из схемы, полученной в nucplot.
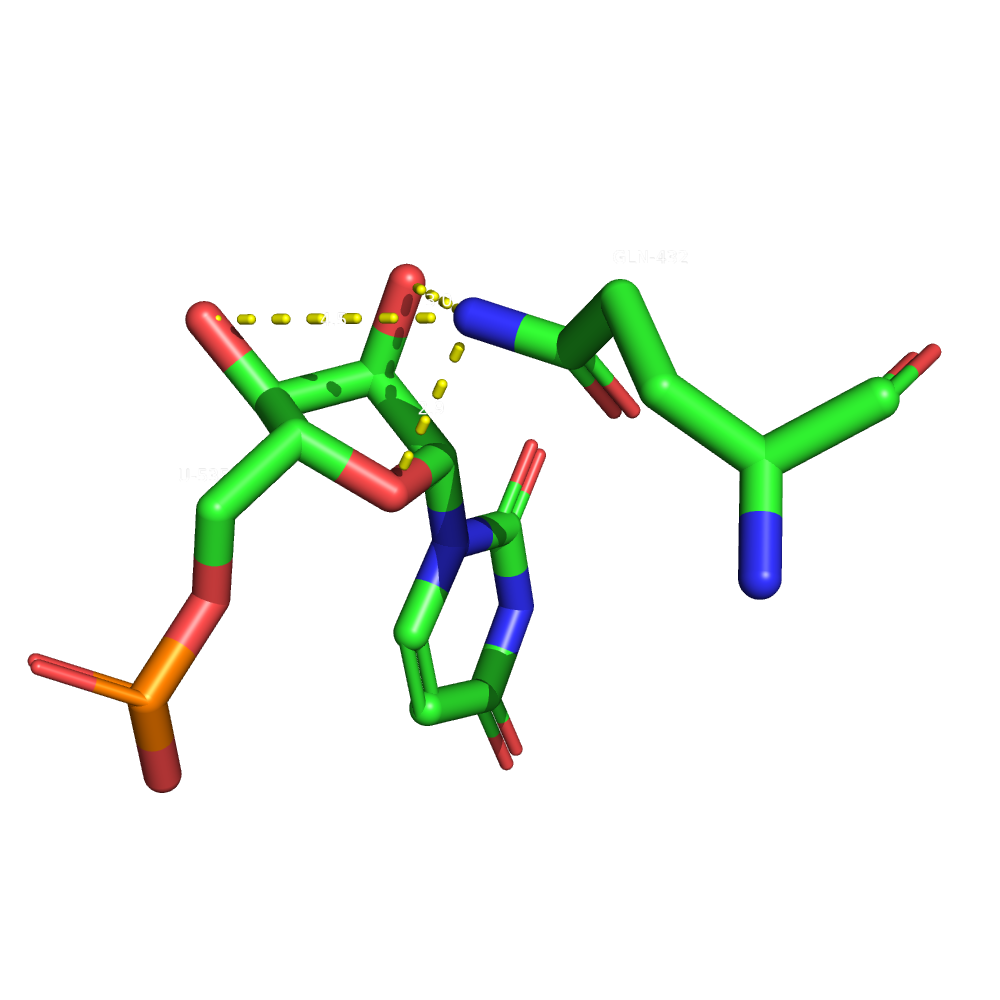
Остаток глутамина связывается с рибозным остовом и вряд ли влияет на распознавание тРНК.
Как наиболее важный для распознавания был выбран треонин 444 - связывается с азотистым основанием.