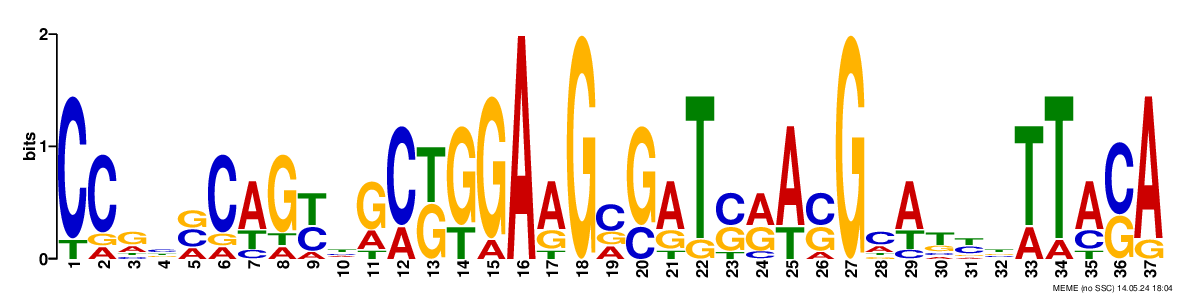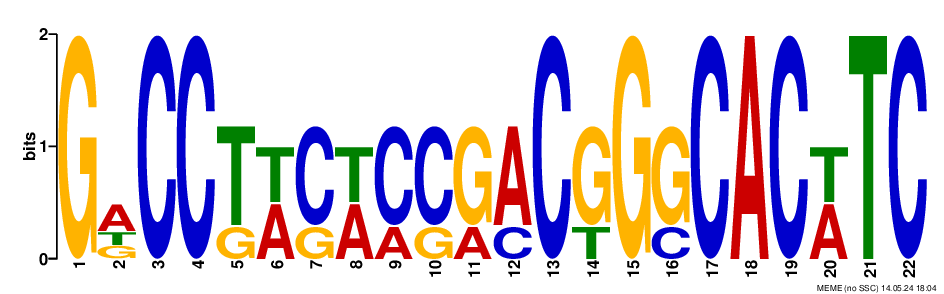Поиск de novo сигналов в ДНК
В этом практикуме я буду искать сигналы посадки сигма-субъединицы РНК-полимеразы в геноме Shigella flexeneri.
Были скачаны геном бактерии и его аннотация. Они были загружены в сервис Operon-mapper - был получен файл с оперонами. С помощью скрипта Георгия Муравьева были получены 3 выборки: обучающая(гены домашнего хозяйства), тестирования(все промоторы) и негативного контроля(случайные участки генома).
Далее была запущена программа meme:
meme housekeeping.fasta -dna -nmotifs 3 -minw 6
HTML-отчет программы MEME и ее текстовый отчет
У первого мотива наименьший E-value, поэтому дальше я буду использовать его.
Далее для поиска мотива в файлах с выборкой и негативным контролем была использована программа fimo. Мотив был взят из текстовой выдачи meme.
fimo --norc -motif GCCAMGCAABVAMCBYAAAAHCACCABCATVMACTAAATCASYDWGTTSG -thresh 0.001 ./meme_out_promotors/meme.txt ./promotors.fasta
fimo --norc -motif GCCAMGCAABVAMCBYAAAAHCACCABCATVMACTAAATCASYDWGTTSG -thresh 0.001 ./meme_out_negative/meme.txt ./negative.fasta
Мотив нашелся в 348 промоторах положительного контроля и 20 последовательностях негативного контроля.