| Главная страница | Обучение | Обо мне | Ссылки |
Укоренение филогенетических деревьев | |||
|
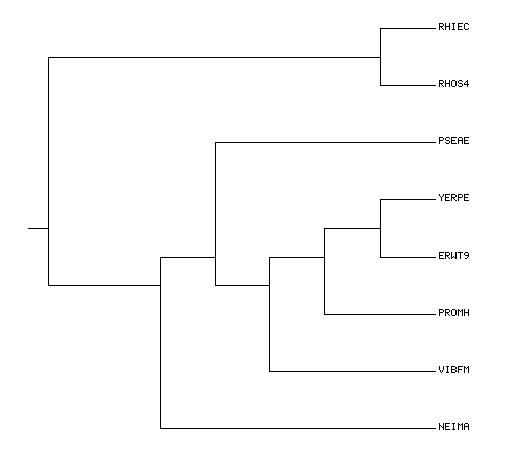
Для удобства в последующей работе на рис. 1 приведено филогенетическое дерево бактерий, построенное в первом практикуме и считаемое правильным. Мнемоника и таксономия организмов приведены на той же странице первого практикума.
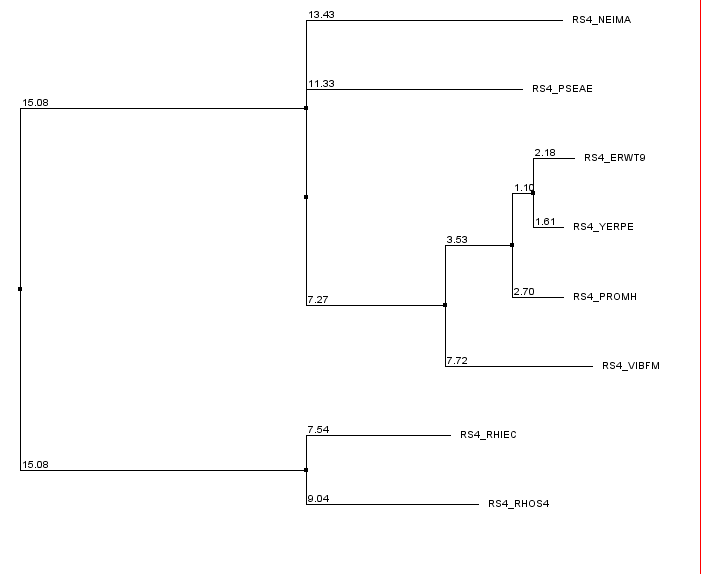
Затем было предложено реконструировать филогенетическое дерево на основе какого-либо консервативного белка; был выбран рибосомальный белок RS4. Выравнивание последовательностей этого белка из 8 разных бактерий (взяты из БД Uniprot) было построено программой Muscle, на основе этого выравнивания с помощью алгоритма Neighbour Joining using % identity было построено дерево. Оно приведено на рис. 2.
Приведенное выше дерево не является укорененным, поэтому далее было проделано укоренение в среднюю точку: исходное дерево было сохранено в newick-формате (файл)и открыто программой retree пакета Phylip. В этой программе оно и было укоренено. Результат приведен на рис. 3.
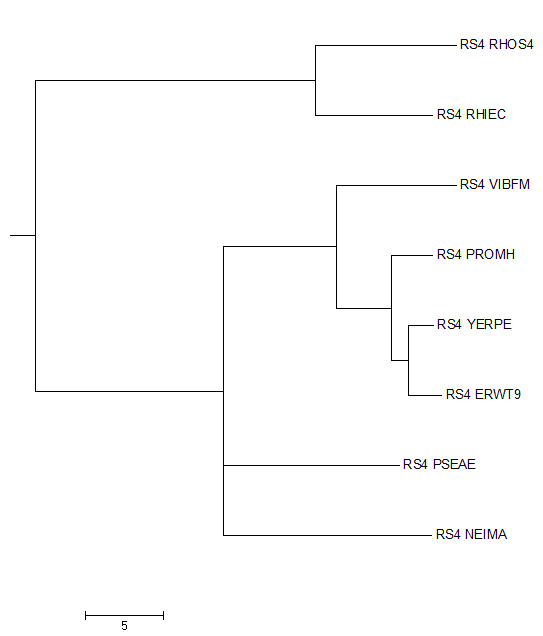
Укоренения в среднюю точку означает, что за корень берется середина наибольшего расстояния между всеми листьями дерева. Таким образом, метод является довольно грубой оценкой филогении организмов, однако в данном случае корень попал в "правильное" место, то есть разделил ветви {RHIEC, RHOS4} (бактерии, относящиеся к альфа-протеобактериям) и {NEIMA, PROMH, YERPE, ERWT9, VIBFM, PSEAE} (в эту ветвь вошли бета- и гамма-протеобактерии). Также можно заметить, что методом Neighbour Joining было построенно неразрешенное (небинарное) дерево. Аналогичное построение по выравниванию в программе MEGA дало дерево с отдельной нетривиальной ветвью {NEIMA, PSEAE}, подтвердить существование такой ветви возможно построением укорененного дерева другими методами. Например, методы UPGMA и MЕ дали результат, показанный на рис. 4.
На этом дереве также пара {NEIMA, PSEAE} выделена в отдельный кластер. Это не очень понятным образом согласуется с таксономией организмов, так как NEIMA относится к классу Betaproteobacteria, а PSEAE - к классу Gammaproteobacteria. На страницу четвертого семестра | |||
| © Alexandra Boyko, 2014. Faculty of Bioengineering and Bioinformatics, MSU. | |||