| Главная страница | Обучение | Обо мне | Ссылки | ||||||||||||||||||
Реконструкция филогении по нуклеотидным последовательностям. Паралоги. | |||||||||||||||||||||
|
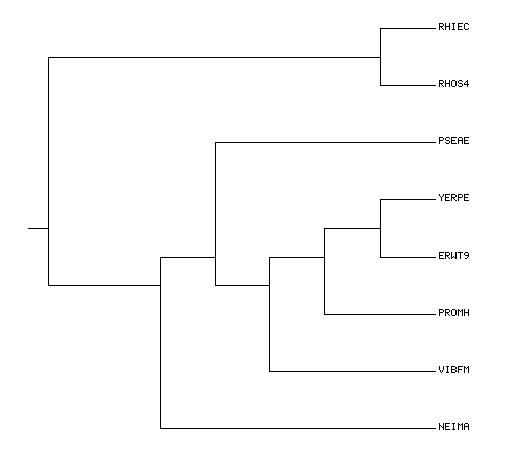
Задание 1. Построение дерева по нуклеотидным последовательностям С сервера NCBI для каждой бактерии были взяты нуклеотидные последовательности 16S РНК (из файлов с расширением .frn). Они были собраны в fasta-файл 16S_all.fasta. Последовательности были выровнены в Jal-view с помощью программы Muscle, выравнивание сохранено в файл 16S_align.fasta. Затем в MEGA с помощью метода Maximum Likelihood было построено филогенетическое дерево (приведено на рис. 1, слева). Для сравнения, на рис. 1 справа показано "правильное" дерево, отвечающее таксономии данных организмов.
Сходство деревьев оценивается по их топологии, которая, в свою очередь, определяется набором разбиений деревьев на нетривиальные ветви. Поэтому, чтобы сравнить эти два дерева, нужно понять, по-разному ли их можно разбить на пары нетривиальных ветвей. В Таблице 1 приведены эти разбиения и отмечено, для какого дерева оно подходит.
Из Таблицы 1 хорошо видно, что наборы разбиений для обоих деревьев совпадают. Это означает, что они одинаковы. Задание 2. Построение и анализ дерева, содержащего паралоги Для поиска гомологов белка CLPX_ECOLI была создана база данных из протеомов 8 исследуемых бактерий (на основе файла bact.fasta). Затем среди белков выбранных бактерий были найдены гомологи (с пороговым E-value = 0.001), результат запуска blastp содержится в файле results.txt. Использованные команды:
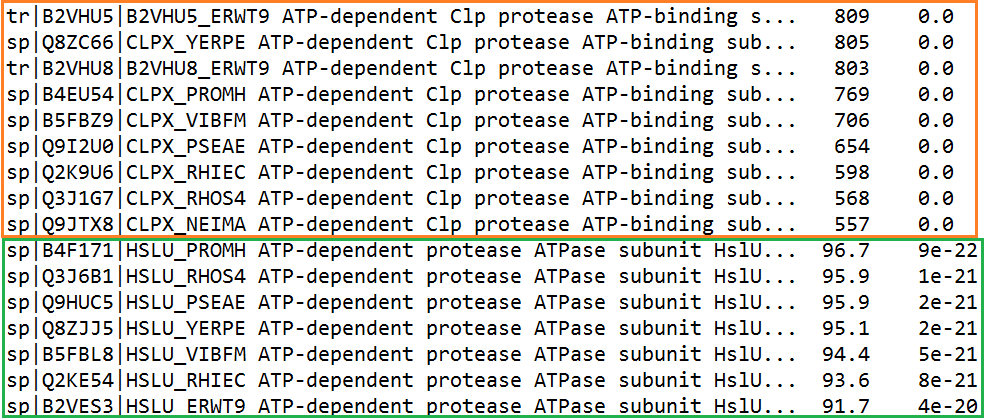
Всего находок, соответствующих установленному E-value, 27, однако действительно гомологами, мне кажется, следуюет считать лишь первые 16: с E-value до порядка -20, так как дальше идет резкий переход к E-value порядка -5/-4, а так же названия находок меняются. При этом внутри найденных последовательностей легко можно выделить 2 группы: собственно белки CLPX (с более хорошими показателями) и белки АТФазной субъединицы протеазы. Гомологи приведены на рис. 2.
Далее были взяты полные последовательности 16 гомологов, они были выровнены программой Muscle, по выравниванию в программе MEGA было построено дерево
Рис. 3. Выравнивание гомологов белка CLPX_ECOLI, построенное с помощью Muscle. Рис. 4. Дерево белков ClpX и HslU, построенное на основе их выравнивания с помощью алгоритма Neighbor-Joining.
Желтая линия обозначает дупликацию гена в организме ERWT9, в желтых и оранжевых рамках белки-паралоги в организмах PSEAE и PROMH соответственно, в зеленые рамки взяты 2 группы ортологов белков ClpX и HslU, голубые рамки обозначают видообразование. Во всех выбранных бактериях есть гомологи белка CplX_ECOLI, а белки HslU нашлись во всех, кроме бактерии NEIMA (Neisseria meningitidis). На этом дереве показаны различные эволюционные события и отношения некоторых белков друг с другом. Итак, ортологами будем считать белки разных организмов при условии, что разделение их ветвей произошло в результате видообразования, а паралогами будем называть гомологичные белки из одного организма. На дереве сразу видно дупликацию гена в бактерии ERWT9 (желтая линия справа), которая привела к образованию двух одинаковых белков B2VHU5 и B2VHU8. Некоторые случаи видообразования отмечены голубыми рамками. Далее, ортологами друг другу являются все белки ClpX (верхняя зеленая рамка на Рис. 4) и HslU (нижняя зеленая рамка), поскольку каждый белок (внутри группы ортологов) отделен веткой, разделяющей виды, от других. На страницу четвертого семестра | |||||||||||||||||||||
| © Alexandra Boyko, 2014. Faculty of Bioengineering and Bioinformatics, MSU. | |||||||||||||||||||||