Занятие 10: Эволюция доменной архитектуры
AC: PF05139
ID: Erythro_esteras
Функция: Семейство включает в себя ферменты эритромицинэстеразы, которые,
расщепляя сложноэфирную связь в молекуле антибиотика эритромицина, обеспечивают
устойчивость клетки к эритромицину.
Доменные архитектуры: всего - 8, представляю 2 из них:
E9CW7_COCPS (436 последовательностей) 
D3D9M8_9ACTO (44 последовательности) 
Другой домен этого семейства - Pribosyltran (PF00156) - входит в состав различных
ферментов с фосфорибозилтрансферазной активностью. Содержится в 26992 последовательностях
у 3187 видов, входит в состав 64 различных доменных архитектур.
Распределение по таксонам: 534 последовательности у 375 видов.
Домен имеет широкую таксономическую нишу и представлен в следующих Царствах (в скобках - количество последовательностей):
Archaea (4)
Bacteria (476)
Eukaryota (52)
и еще 2 неклассифицированные последовательности.
Выравнивание последовательностей из домена
Подготовка выборки:
python swisspfam_to_xls.py -z swisspfam.gz -p PF05139 -o py1.xls
python uniprot_to_taxonomy.py -i flat_text.txt -o taxonomy.xls
Также добавила последовательность Q81BF7_BACCR (2QGM) с известной пространственной структурой
Таблица Excel с описанием последовательностей
Выравнивание выборки последовательностей из двух архитектур
Построение филогенетического дерева выборки последовательностей
Выравнивание с отредактированными именами последовательностей
Расшифровка:
1 - доменная архитектура E9CW7_COCPS (однодоменная)
2 - доменная архитектура D3D9M8_9ACTO (двудоменная)
Pb - Протеобактерии
Ab - Актинобактерии
Cy - Цианобактерии
Ch - Chlamydiae (из царства Бактерий)
F - Фирмикуты (из царства Бактерий)
B - Bacteroidetes (из царства Бактерий)
A - Археи
E - Эукариоты
Филогенетическое дерево, построенное методом UPGMA:
Видно, что клады на дереве расположены хаотично, четкого разделения по доменным аархитектурам, либо по таксономии не произошло.
На мой взгляд, это дерево построено неправильно и ориентироваться на него нельзя.
Скобочная формула
Филогенетическое дерево, построенное методом Neighbor-Joining:
Здесь, очевидно, произошло разделение по доменным архитектурам (исключение составляет одна последовательность,
выделенная толстой серой линией, по-видимому это небольшая погрешность). По данному дереву я могу предположить,
что доменные архитектуры E9CW7_COCPS и D3D9M8_9ACTO развивались независимо друг от друга. Также видно достаточно четкое разделение
по таксонам: видно разделение на Актино- и Протеобактерий с двудоменной архитектурой; у Бактерий независимо эволюционировала
однодоменная архитектура, причем в какой-то момент времени в ней выделились эукариотческая и архейная архитектуры.
Скобочная формула
Профили
Получены профили для первой
и второй групп последовательностей
График зависимости числа ошибок первого (False Negatives) и второго (False Positives) рода
от порогового значения score для первого профиля:
Данный профиль не позволяет отличить первую группу последовательностей
График зависимости числа ошибок первого (False Negatives) и второго (False Positives) рода
от порогового значения score для второго профиля:
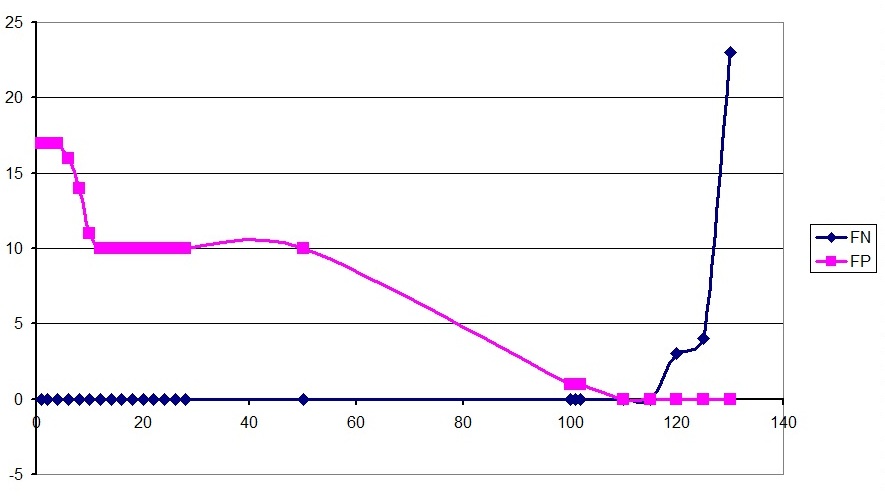
Второй профиль позволяет отличить вторую группу последовательностей: при пороговом значении,
равном 110, число верных находок (True Positives) равно 23, верно пропущенных последовательностей
(True Negativies) - 17, число ошибок первого и второго рода равны 0.
Жаль, конечно, что первый профиль получился плохо, зато второй - отличный, этого и следовало
ожидать, потому что в выравнивании второй группы последовательностей, в отличие от первой,
очень много высококонсервативных позиций.
Также я провела поиск по банку SwissProt, используя второй профиль. Нашлась лишь одна
последовательность с доменной архитектурой D3D9M8_9ACTO, что соответствует истине.
Столь малое количество находок можно объяснить тем, что уже после проведения
поиска я узнала, что все последовательности из моего выравнивания (кроме одной
найденной) были взяты не из SwissProt, а из TrEMBL.
E-mail: yan1303@yandex.ru
Официальный сайт ФББ
Ваши предложения :)))
Проекты
Главная страница