Практикум 9:
Поиск сигнала посадки σ-субъединицы РНК-полимеразы Clostridium butyricum.
Логически продолжная прошлый практикум сигналы будем искать в геноме бактерии Clostridium butyricum. Был использован референсный геном (см. ссылку в практикуме 8, семестр 4), скачаны FASTA-файлы хромосомы и информация о анотации в GFF формате. Эти файлы мы подадим на вход программе Operon-mapper. Выдачу данной программы мы подадим на скрипту, написанному Георгием Муравьёвым.
С помощью данного скрипта мы получили готовую обучающую (промоторы оперонов генов домашнего хозяйства, за промотры принимались участки в 100 нуклеотидов перед старт-кодоном), тестируемую (промоторы всех оперонов) и негативную, состоящую из случайно вырезанных участков генома длиной в 100 нуклеотидов, выборки.
Поиск мотивов с помощью MEME.
Будем искать сигналы с помощью программы MEME. На вход подаём команду: meme housekeeping.fasta -dna -nmotifs 3 -minw 6 -maxw 50 Где: -dna специфицирует поиск в ДНК. -nmotifs==3 устанавливает число мотивов для поиска равное трём. -minw, maxw максимальная и минимальная длины находок в нуклеотидах.
Выдача программы состояла из HTML-файла, txt-файла, а также LOGO, для трех находок.
Находка №1. e-value=2.3e-097
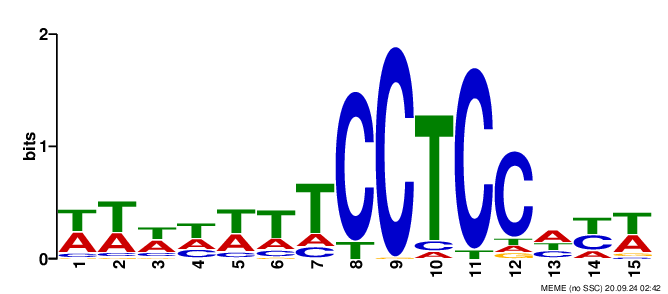
Находка №2. e-value=1.0e-028

Находка №3. e-value=1.3e-003
Поиск сигнала в тестируемой выборке с помощью FIMO.
Возьмём находу с самым небольшим e-value, а именно №1 (WWWHWTTCCTCCHYW)
Возьмём порог p-value 10^-3 тогда наш код будет выглядеть так: fimo --norc -motif WWWHWTTCCTCCHYW -thresh 0.001 ./meme_out/meme.txt ./promotors.fasta fimo --norc -motif WWWHWTTCCTCCHYW -thresh 0.001 ./meme_out/meme.txt ./negative.fasta (--norc означает что поиск будет проводится только по одной цепи 'no reverse complement')
Поиск по негативной выборке обнаружил 81 последовательность, поиск по тестируемой выборке обнаружил 2245 последовательностей похожих на искомый мотив.