Задание 1. Построение модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA.
С помощью программы fiber пакета 3DNA были построены A- , B- и Z-формы дуплекса ДНК.
Для этого в А- и В- форме пять раз были повторены основания gatc, а в Z- форме 10 раз gc.
Задание 2. Упражнение 1.
Для выполнения данного задания был выбран 31 аденин в молекуле тРНК (1u0b.pdb). С помощью MarvinSketch было получено изображение данного основания.
Рис.2. Изображение аденина.
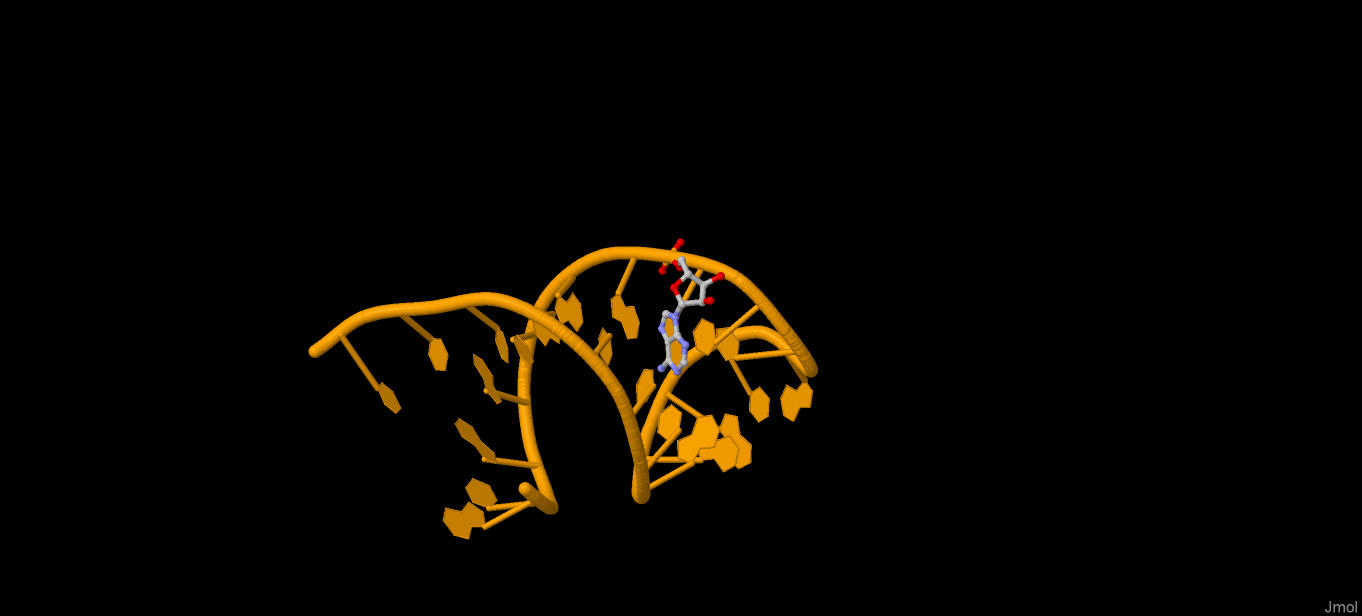
Рис.2. Изображение аденина.
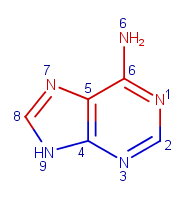 |
На рисунке 1 красным изображены атомы, обращенные в сторону большой бороздки, а синим - в сторону малой.
- большая бороздка: C5, C6, C8, N7, N6, N1
- малая бороздка: C2, C4, N3, N9
|
Задание 2. Упражнение 2.
Таблица 1. Сравнение A- B- и Z-формы ДНК.
| | A-форма | B-форма | Z-форма |
| Тип спирали (правая или левая) | правая | правая | левая |
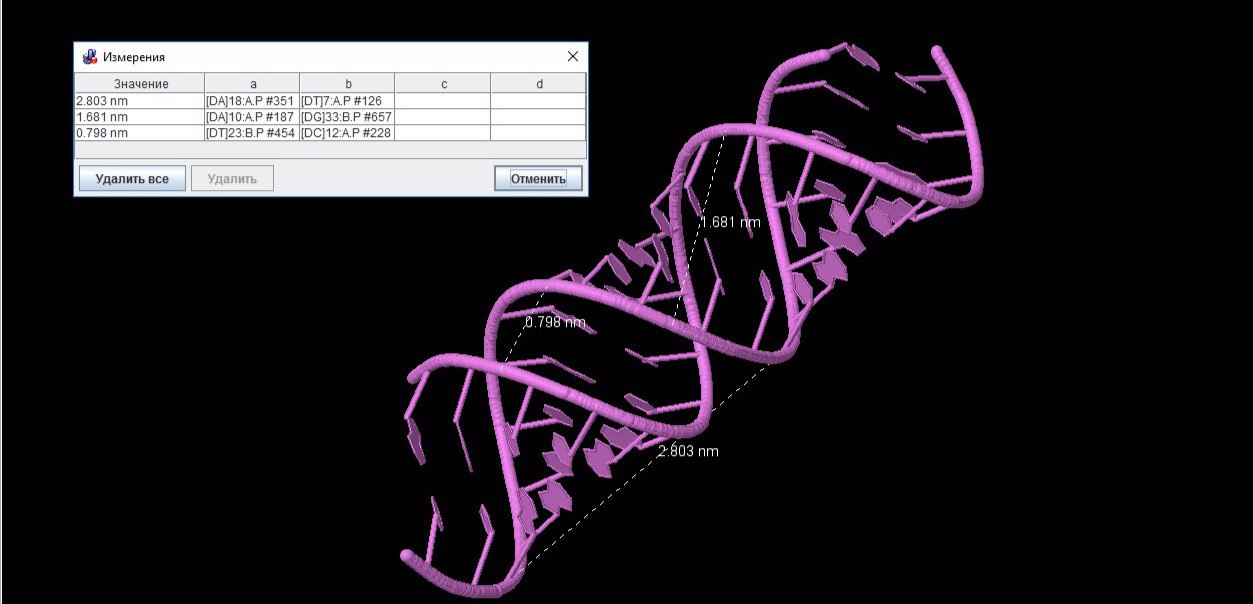
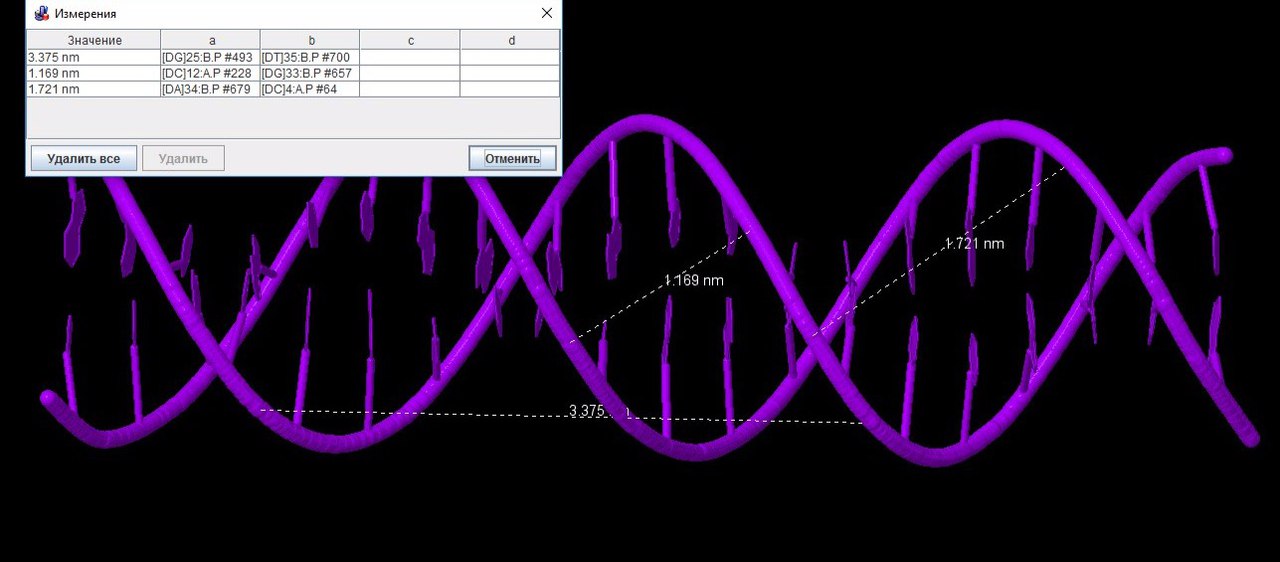
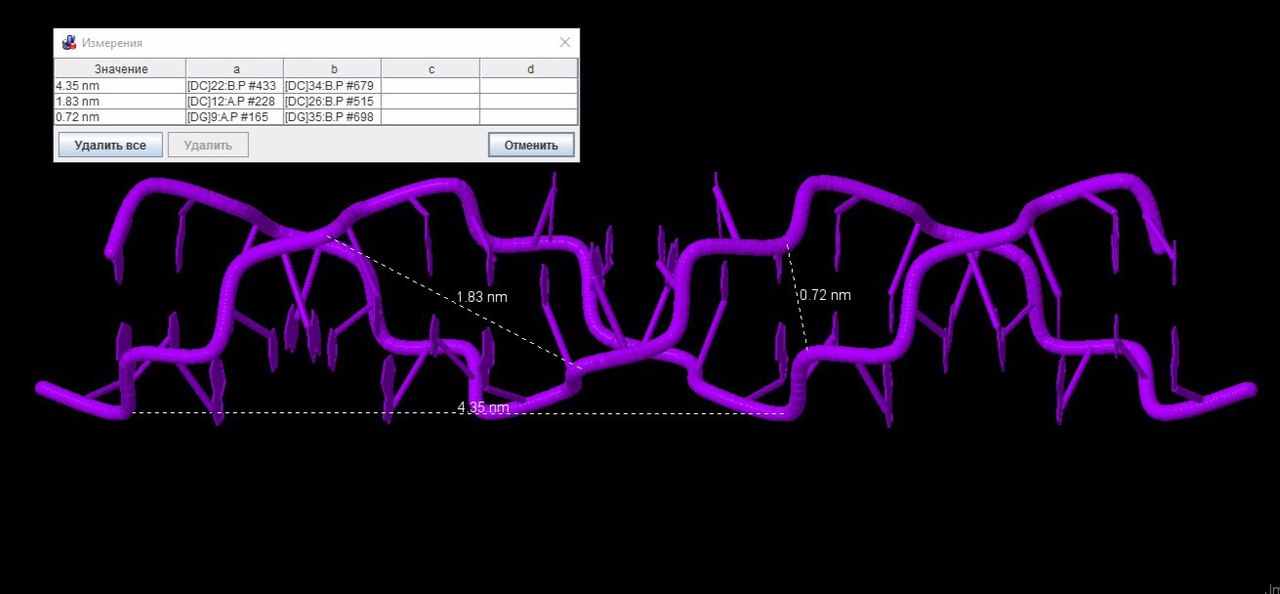
| Шаг спирали (A) | 28.03 ([DA]18:A.P-[DT]7:A.P) | 33.75 ([DG]25:B.P-[DT]35:B.P) | 43.50 ([DC]22:B.P-[DC]34:B.P) |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (A) | 16.81 ([DA]10:A.P-[DG]33:B.P) | 17.21([DC]12:A.P-[DG]33:B.P) | 18.30 ([DC]12:A.P-[DC]26:B.P) |
| Ширина малой бороздки (A) | 7.98 ([DT]23:B.P-[DC]13:A.P) | 11.69 ([DA]34:B.P-[DC]4:A.P) | 7.20([DG]9:A.P-[DG]35:B.P) |
Таблица 2. Изображения A- B- и Z-формы ДНК.
Задание 3. Упражнение 1.
Для выполнения следующих заданий использовала пакет 3DNA.
Так как он работает только со старым форматом PDB, использовала программу remediator для перевода в старый формат.
С помощью команды find_pair -t XXXX.pdb stdout | analyze был записан файл XXX.out ( "XXX" = 1uob_old), из которого брала данные для нахождения торсионных углов и водородных связей.
Таблица 3. Торсионные углы для тРНК и ДНК.
| | alpha | beta | gamma | delta | epsilon | zeta | chi |
| тРНК |
| 1 цепь тРНК | -47,64 | 42,56 | 64,98 | 84,28 | -137,0 | -71,41 | -137,67 |
| 2 цепь тРНК | -36,60 | 68,81 | 46,97 | 82,41 | -126,71 | -63,58 | -159,67 |
| ДНК |
| 1 цепь ДНК | -45,61 | 14,31 | 22,33 | 137,82 | -90,35 | -77,35 | -110,96 |
| 2 цепь ДНК | -29,93 | 59,24 | 31,61 | 137,37 | -67,63 | -82,35 | -109,69 |
| Данные из презентации |
| A-ДНК | 62 | 173 | 52 | 88/3 | 178 | -50 | -160 |
| B-ДНК | 63 | 171 | 54 | 123/131 | 155 | -90 | -117 |
Расчеты среднего значения торсионных углов.
Можно заметить, что значения, полученные для тРНК(1u0b.pdb) и ДНК(1bdt.pdb) значительно отличаются от тех, которые были приведены для A- и B-форм ДНК. Однако,
если сравнивать их, то тРНК больше похожа на А-форму(gamma, delta, zeta, chi), a ДНК на B-форму (delta, zeta, chi).
Наиболее сильно "деформированные" нуклеотиды (с наибольшим отклонением от среднего значения) являются:
- Для тРНК -
- 15 аденин - alpha : 156,50(среднее -47,64) beta : -176,40 (42,56) gamma : 170,70 (64,98)
- 26 аденин - alpha : 138,9 (-36,6) beta -111,7 (68,81) gamma : 153 (46,97) delta: 105,3 (82,41) epsilon: -87,7 (-126,71)
- Для ДНК -
- 18 тимин - beta : -59,50 (14,31) gamma : -176,80 (22,33) epsilon: -146,10(-90,35) chi: -146,10 (-110,96)
- 18 аденин - alpha : 168,80 (-29,93) beta -172,00 (59,24) gamma : 172,50 (31,61) epsilon: -155,00 (-67,63)
Задание 3. Упражнение 2.
тРНК состоит из 4 стеблей:
1 стебель
|
1 (0.009) ....>A:...1_:[..G]G-----C[..C]:..72_:A<.... (0.007)
2 (0.006) ....>A:...2_:[..G]G-----C[..C]:..71_:A<.... (0.010)
3 (0.004) ....>A:...3_:[..C]C-----G[..G]:..70_:A<.... (0.009)
4 (0.012) ....>A:...4_:[..G]G-----C[..C]:..69_:A<.... (0.003)
5 (0.004) ....>A:...5_:[..C]C-----G[..G]:..68_:A<.... (0.011)
6 (0.016) ....>A:...6_:[..G]G-----C[..C]:..67_:A<.... (0.006)
7 (0.008) ....>A:...7_:[..U]U-----A[..A]:..66_:A<.... (0.006) |
2 стебель
|
8 (0.008) ....>A:..49_:[..U]U-----A[..A]:..65_:A<.... (0.006)
9 (0.004) ....>A:..50_:[..C]C-----G[..G]:..64_:A<.... (0.005)
10 (0.006) ....>A:..51_:[..C]C-----G[..G]:..63_:A<.... (0.012)
11 (0.008) ....>A:..52_:[..G]G-----C[..C]:..62_:A<.... (0.005)
12 (0.006) ....>A:..53_:[..G]G-----C[..C]:..61_:A<.... (0.003)
13 (0.010) ....>A:..54_:[..U]U-**--A[..A]:..58_:A<.... (0.013) |
3 стебель
|
15 (0.008) ....>A:..36_:[..A]A-**--U[..U]:..33_:A<.... (0.006)
16 (0.010) ....>A:..38_:[..A]A-**--U[..U]:..32_:A<.... (0.005)
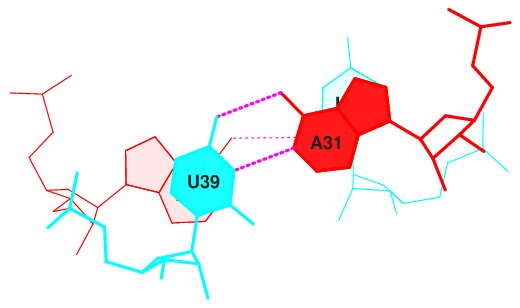
17 (0.006) ....>A:..39_:[..U]U-----A[..A]:..31_:A<.... (0.007)
18 (0.005) ....>A:..40_:[..C]C-----G[..G]:..30_:A<.... (0.012)
19 (0.004) ....>A:..41_:[..C]C-----G[..G]:..29_:A<.... (0.006)
20 (0.008) ....>A:..42_:[..G]G-----C[..C]:..28_:A<.... (0.004)
21 (0.003) ....>A:..43_:[..U]U-*---G[..G]:..27_:A<.... (0.006)
22 (0.006) ....>A:..44_:[..C]C-**--A[..A]:..26_:A<.... (0.004) |
4 стебель
|
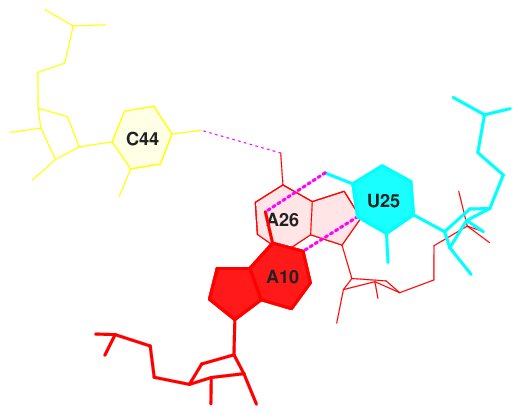
23 (0.008) ....>A:..10_:[..A]A-----U[..U]:..25_:A<.... (0.010)
24 (0.004) ....>A:..11_:[..C]C-----G[..G]:..24_:A<.... (0.007)
25 (0.006) ....>A:..12_:[..A]A-----U[..U]:..23_:A<.... (0.007) |
Таблица 4. Неканонические взаимодействия в молекуле тРНК:
| взаимодействия | водородную связь образуют атомы | водородную связь образуют атомы | водородную связь образуют атомы |
| канонические взаимодействия |
| U-----A | N3 - N1 | O4 - N6 | |
| G-----C | O6 - N4 | N1 - N3 | N2 - O2 |
| неканонические взаимодействия |
| U-**--A | O2 - N6 | O4 * N7 | |
| U-**+-G | O2 - N2 | | |
| A-**--U | OP2- N3 | N7 - O2 | |
| A-**--U | N6 - O2 | | |
| U-*---G | O2 - N1 | N3 - O6 | |
| C-**--A | N4 * N6 | | |
| A-**--A | N6 - N7 | N1 - N6 | |
| A-**--U | N7 - N3 | N6 - O2 | |
| G-**+-G | O6 - N2 | | |
| C-**+-C | O2 - N4 | N3 * N3 | N4 - O2 |
| G-*---C | N2 - N3 | | |
| U-**+-C | O2'- OP1 | O2 - N4 | |
Дополнительные водородные связи стабилизирующие молекулу:
|
26 (0.007) ....>A:..13_:[..A]A-**--A[..A]:...9_:A<.... (0.008)
27 (0.012) ....>A:..14_:[..A]A-**--U[..U]:...8_:A<.... (0.005)
28 (0.006) ....>A:..15_:[..G]G-**+-G[..G]:..48_:A<.... (0.008) |
Задание 3. Упражнение 3.
С помощью команд ex_str -n stacking.pdb stepn.pdb и stack2img -cdolt stepn.pdb
stepn.ps получили изображения азотистых оснований с максимальным(16) и минимальным(22) перекрываниями.
| step | i1-i2 | i1-j2 | j1-i2 | j1-j2 | sum |
| 22 CA/UA | 0.00( 0.00) | 0.00( 0.00) | 0.00( 0.00) | 0.00( 0.00) | 0.00( 0.00) |
| 16 AU/AU | 5.35( 3.23) | 0.00( 0.00) | 0.00( 0.00) | 6.79( 3.50) | 12.14( 6.73) |
|