 |
 |
|
|
Сравнение матриц аминокислотных замен Таблица N1. Результаты поиска гипотетических гомологов изучаемого белка (SCO1_BACSU).
Искомый белок удалось найти и в "nr", и в Swiss-Prot. К сожалению, в PDB удалось найти только очень похожий белок (Идентичность равна 99%). В PDB число явных гомологов меньше, чем в Swiss-Prot, а в Swiss-Prot число явных гомологов меньше, чем в nr. Объясняется это просто. PDB является базой данных для 3D структур белков. В nr хранятся предполагаемые белки, которых в разы больше подтвержденных. Номер последней находки Swiss prot: 59.PDB: 39 .nr: 4218. Лимит был задан значением E-value. Таблица N2.Лучшая находка в наиболее далеких таксонах (Здесь - Eukaryota).
BLAST двух последовательностей. Были составлены две карты локального сходства изучаемого белка и его гомолога Q9ZCW7.1. Был использован разный порог E-value (один равен 10, другой - 0.01) 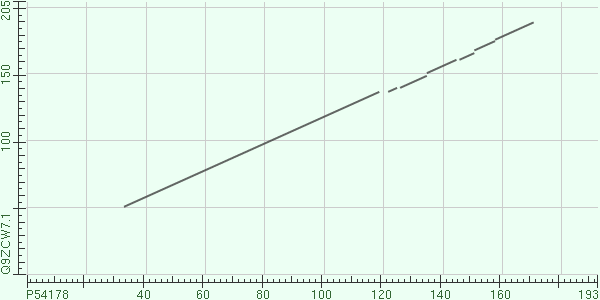
Карта локального сходства с порогом E-value, равным 10. Query 34 PFTFQNQDGKNVSLESLKGEVWLADFIFTNCETICPPMTAHMTDLQKKLKAENIDVRIIS 93
PF +Q+G+ + + L+G + L F FT+C ICP +T++ + L ID+ +
Sbjct 52 PFELIDQNGEIFNSDKLRGHLSLIYFGFTSCPDICPTSLNKITNIVEILHQNKIDIIPVF 111
Query 94 FSVDPENDKPKQLKKFAANYPLSFDNWDFLTGYSQSEIEEFA--LKSFKAIVKKPEGEDQ 151
+VDP+ D P+ LK++ N+ F + LTG ++ +I++ K F A V + +DQ
Sbjct 112 ITVDPKRDTPEVLKEYIKNFHPKFIS---LTG-NEHQIKDVTDKFKVFYARVNS-DNDDQ 166
Query 152 --VIHQSSF-YLVGPDGKVLKDY 171
+I SSF YL+ +G+ +K +
Sbjct 167 NYMIDHSSFTYLIDKNGRYMKHF 189
Парное выравние двух последовательностей с порогом E-value, равным 10. Скачать выравнивание можно по ссылке: SCO1homolog10.fasta 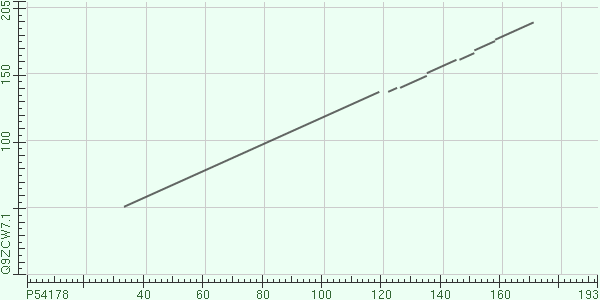
Карта локального сходства с порогом E-value, равным 0.01.
Рисунок 1. Парное выравнивание двух последовательностей с порогом E-value, равным 0.01. Скачать выравнивание можно по ссылке: SCO1homolog001.fasta |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 |
 |
|