|
Составление репрезентативной выборки гомологов изучаемого белка (SCO1_BACSU) с помощью BLAST
Для множественного выравнивания было выбрано 32 гомолога изучаемого белка. Всего гомологов было 3136.
Параметры поиска указаны в таблице N1.
|
Поиск
|
Алгоритм
BLAST
|
Название
базы данных
|
Ограничения
по таксонам
|
Порог e-value
|
Максимальное
количество хитов
|
|
По
прокариотам
|
BLASTP
|
Refseq
|
no Firmicutes
no Eukaryota
|
10
|
3136
|
|
По
эукариотам
|
BLASTP
|
Refseq
|
Eukaryota
|
10
|
335
|
Таблица N1. Указаны параметры, использованные для поиска.
Во второй таблице указаны организмы, из которых были взяты белки.
|
Домен
|
Филум/царство
|
Название организма
|
Количество белков
|
|
Archaea
|
Euryarchaeotes
|
Halogranum_salarium
|
123
|
|
Cenarchaeales
|
Pyrobaculum_sp._1860
|
12
|
|
Nitrososphaerales
|
Candidatus_Nitrososphaera_gargensis_Ga9.2
|
1
|
|
Bacteria
|
Proteobacteria
|
Pseudomonas_protegens_CHA0
|
2098
|
|
Acidovorax_radicis
|
|
CFB_group_bacteria
|
Flavobacteriaceae_bacterium_HQM9
|
398
|
|
Cyclobacteriaceae_bacterium_AK24
|
|
Spirochetes
|
Leptospira_interrogans
|
158
|
|
Actinobacteria
|
Streptomyces_acidiscabies
|
119
|
|
GNS bacteria
|
Nitrolancetus_hollandicus
|
31
|
|
Planctomycetes
|
Rhodopirellula_baltica
|
30
|
|
Aquificales
|
Hydrogenivirga_sp._128-5-R1-1
|
29
|
|
Verrucomicrobia
|
Verrucomicrobium_spinosum
|
25
|
|
Cyanobacteria
|
Arthrospira_platensis
|
20
|
|
Deinococcales
|
Deinococcus_peraridilitoris_DSM_19664
|
18
|
|
Thermales
|
Thermus_sp._RL
|
13
|
|
Acidobacteriales
|
Granulicella_tundricola_MP5ACTX9
|
11
|
|
Solibacterales
|
Candidatus_Solibacter_usitatus_Ellin6076
|
10
|
|
Ignavibacteriales
|
Melioribacter_roseus_P3M-2
|
9
|
|
Candidatus Koribacter
|
Candidatus_Koribacter_versatilis_Ellin345
|
6
|
|
Green sulfur bacteria
|
Chloroherpeton_thalassium_ATCC_35110
|
5
|
|
Synergistales
|
Anaerobaculum_hydrogeniformans
|
3
|
|
Candidatus Chloracidobacterium
|
Candidatus_Chloracidobacterium_thermophilum_B
|
3
|
|
Thermobaculum
|
Thermobaculum_terrenum_ATCC_BAA-798
|
3
|
|
Caldithrix
|
Caldithrix_abyssi
|
3
|
|
Gemmatimonadales
|
Gemmatimonas_aurantiaca_T-27
|
2
|
|
Holophagales
|
Holophaga_foetida
|
1
|
|
Phycisphaerales
|
Phycisphaera_mikurensis_NBRC_102666
|
1
|
|
Deferribacterales
|
Denitrovibrio_acetiphilus_DSM_12809
|
1
|
|
Nitrospinales
|
Nitrospina_gracilis
|
1
|
|
Nitrospirales
|
Thermodesulfovibrio_yellowstonii_DSM_11347
|
1
|
|
Candidatus Methylomirabilis
|
Candidatus_Methylomirabilis_oxyfera
|
1
|
|
Firmicutes
|
Bacillus_subtilis_st._168
|
|
|
Eukaryotes
|
Animals
|
Homo_sapiens
|
140
|
|
Fungi
|
Laccaria_bicolor_S238N-H82
|
83
|
|
Green plants
|
Solanum_lycopersicum
|
61
|
|
Kinetoplastids
|
Leishmania_donovani
|
23
|
|
Apicomplexans
|
Toxoplasma_gondii_ME49
|
11
|
|
Cellular slime molds
|
Dictyostelium_fasciculatum
|
5
|
|
Ciliates
|
Ichthyophthirius_multifiliis
|
4
|
|
Perkinsida
|
Perkinsus_marinus_ATCC_50983
|
2
|
|
Oomycetes
|
Phytophthora_infestans_T30-4
|
1
|
|
Diatoms
|
Thalassiosira_pseudonana_CCMP1335
|
1
|
|
Choanoflagellates
|
Monosiga_brevicollis_MX1
|
1
|
|
Schizopyrenida
|
Naegleria_gruberi_strain_NEG-M
|
1
|
|
Capsaspora
|
Capsaspora_owczarzaki_ATCC_30864
|
1
|
|
Acanthamoeba
|
Acanthamoeba_castellanii_str._Neff
|
1
|
Таблица N2. Встречаемость изучаемого белка в различных таксонах.

Рисунок N1.Краткое филогеническое дерево гомологов изучаемого белка среди прокариот
При изучении эукариотических белков было замечено, что белок кодируется в ядре, но является митохондриальным.
Множественное выравнивание гомологов белка SCO1_BACSU
Рисунок N2. Множественно выравнивание гомологов изучаемого белка. Гомологи были взяты и из прокариот, и из эукариот.
| Свойство аминокислоты (или аминокислота) | Цвета |
| Алифатические | От голубого переход в фиолетовый |
| Нейтрально заряженные | Желтые |
| Ароматические | Стремящиеся к коричневому |
| Отрицательно заряженные | Красные |
| Положительно заряженные | Зеленые |
| Пролин | Серый |
Вспомогательная таблица.Указаны соответствия между аминокислотой и цветом на рисунке
На рисунке использован процент консервативности, равный 30.
На строке SECONDARY указаны альфа-спирали и бета-тяжи. На строке LIGAND указаны лиганды (L). На строке BLOCKS указаны возможные блоки (B).
Файл jar с множественным выравниванием белка (невыровненные концы удалены): multialign.jar.
Файл jar со вторым варианта множественного выравнивания белка (невыровненные концы оставлены): multialign_var2.jar
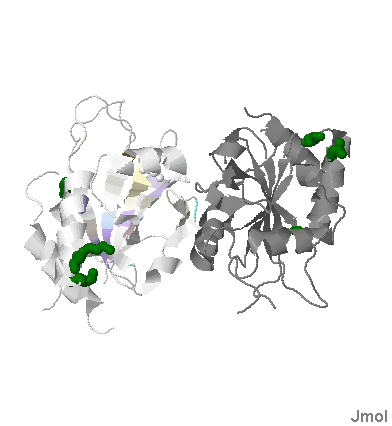
Рисунок N3. Трехмерная структура SCO1_BACSU, с наложением на нее окраски с выравнивания.
Результаты множественного выравнивания гомологов белка SCO1_BACSU
Как можно заметить, участки консервативности в основном соответствуют участкам альфа-спиралей и бета-тяжей. Самые плохие выравнивания находятся на концах последовательностей.
Возможно это из-за того, что на концах у последовательностей нет особо важных частей.
Также на концах особо много "гэп-колонок". Возможно, это можно объяснить тем, что среди выборки присутствуют последовательности примерно в два раза больше SCO1_BACSU.
Почти для всех гомологов изучаемого белка характерна нейтрально заряженная аминокислота под номером 68(45), участвующая в связывании лиганда у исходного белка.
Уже не столь характерны для гомологов аминокислоты цистеин на 64(39) и гистидин на 154(187), тоже связывающие лиганды.
|