 |
 |
|
|
Задание N1 Построение A-, B- и Z- формы ДНК с помощью инструмента 3DNA производилось при помощи программы fiber из пакета 3DNA. Задание N2 Упражнение N1
Рисунок N1. А-форма. Остов ДНК показан красным цветом, все азотистые основания показаны черным цветом, кроме аденина, который покрашен в желтый. Атом N7 в первом по последовательности гуанине покрашен в оранжевый. Упражнение N2 Упражнение N3
Рисунок N2. ДНК цепи из файла 1cf7.pdb. Красным покрашена цепь D, а черным - C. Как можно заметить, в структуре есть разрывы.
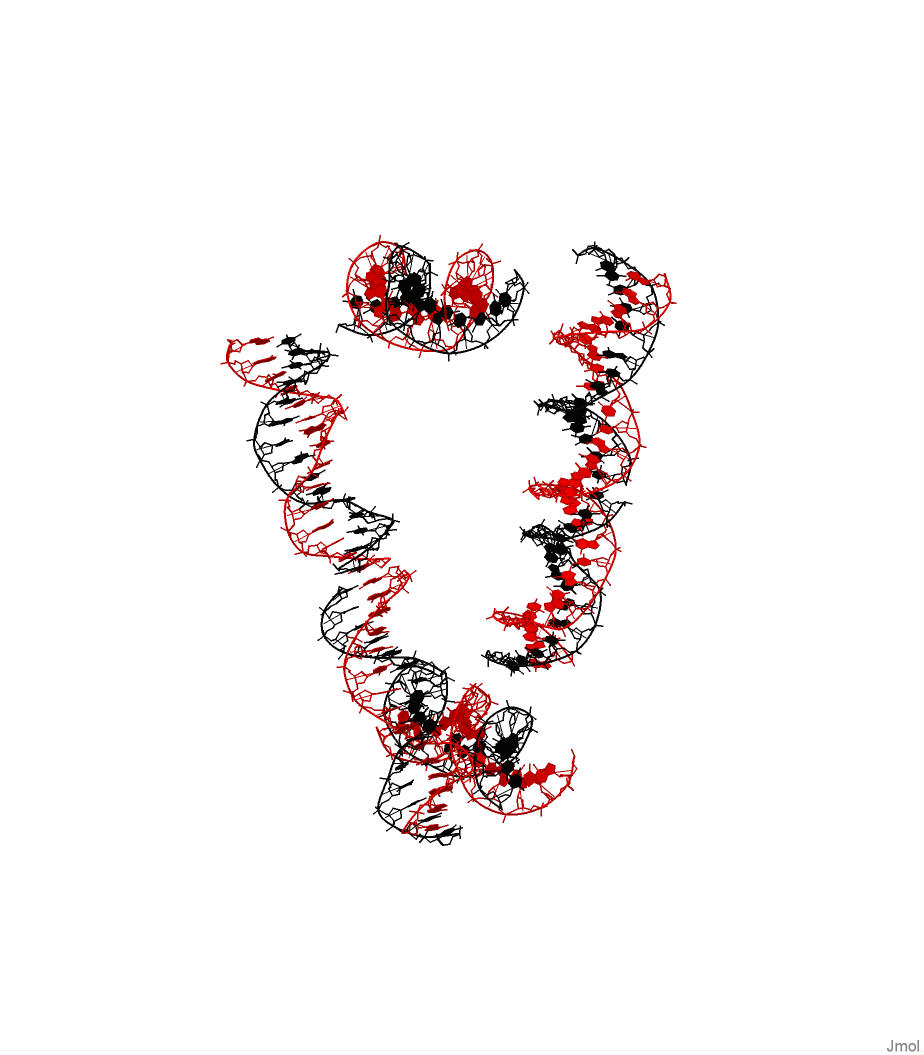
Рисунок N3. РНК цепи из файла 1evv.pdb. Красным покрашена обе цепи, а черным - концы. В структуре нет разрывов. pdb файл с координатами только РНК 1evv. pdb файл с координатами только ДНК 1cf7. Задание N3 Упражнение N1.
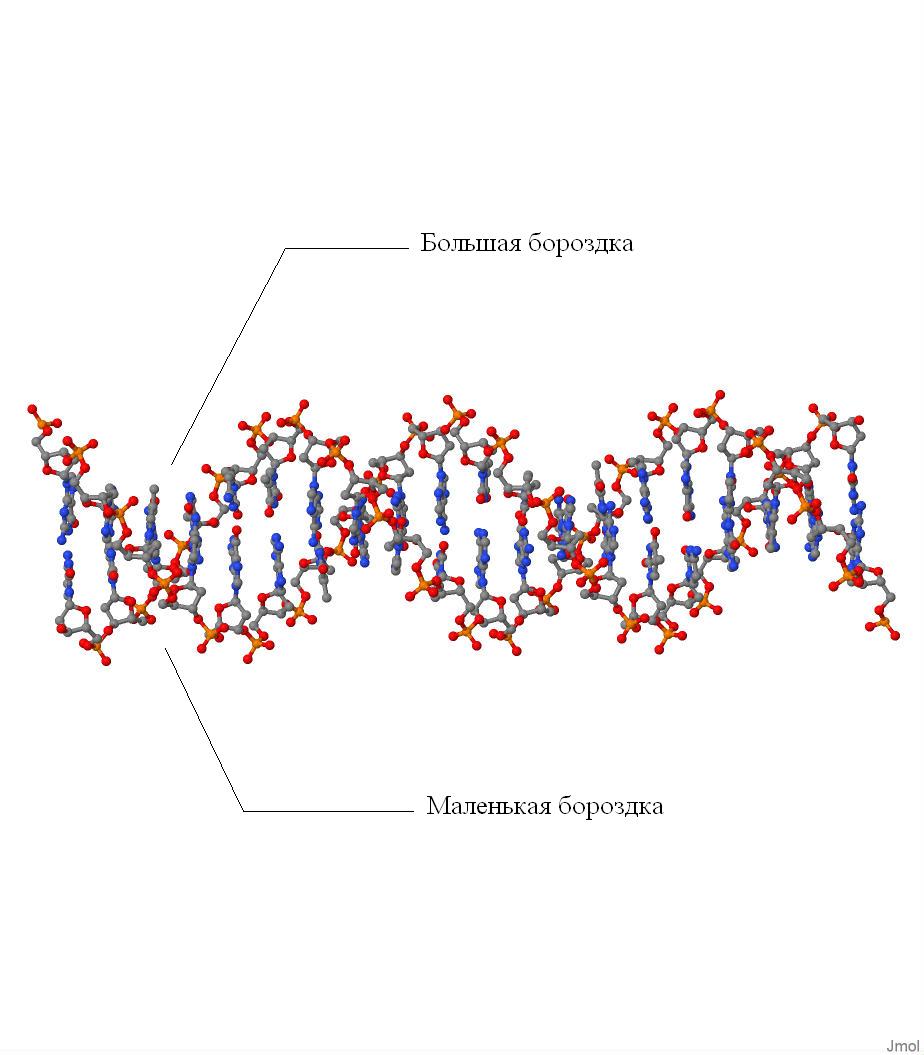
Рисунок N4. Большие и маленькие бороздки на B-форме ДНК.
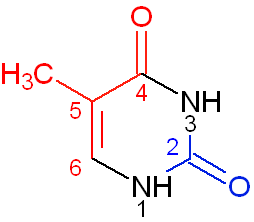
Рисунок N5. Тимин. Синим цветом обозначены атомы, обращенные в сторону малой бороздки, а красным - в сторону большой. В сторону большой бороздки обращены: C6, C5, C5M, C4, O4. В сторону малой бороздки обращены: C2, O2. Остальные атомы: N1, N3. Упражнение N2. Таблица 1. Сравнение основных спиральных параметров разных форм ДНК.
Таблица 2. Сравнение торсионных углов тимина с торсионными углами из презентации.
Задание N4 Таблица 3. Значение торсионных углов в тРНК 1EVV.
Таблица 4. Значения торсионных углов в 1CF7.
Таблица 5. Среднее значение торсионных углов.
Определение структуры водородных связей Акцепторный стебель 5' 1-7 3' 5' 66-72 3' D-стебель 5' 10-13 3' 5' 22-25 3' Антикодоновый стебель 5' 38-44 3' 5' 26-32 3' T-стебель 5' 49-53 3' 5' 61-65 3
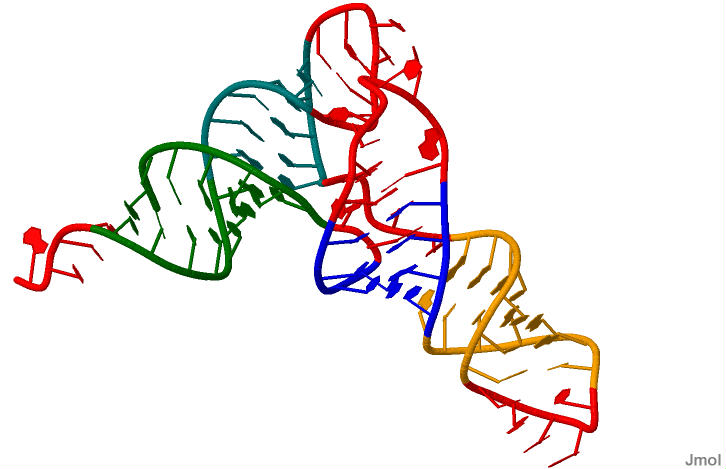
Рисунок N6. Стебли в тРНК 1pdb 1EVV, информация о которых была получена при помощи програмного пакета 3DNA: зеленым выделен акцепторный стебель. Синим - D-стебель. Оранжевым - антикодоновый стебель. Голубым - Т-стебель. Все остальное покрашено красным. * обозначены неканонические пары в файле: 4 (0.009) A:...4_:[..G]G-*---U[..U]:..69_:A (0.007) | 13 (0.011) A:..54_:[5MU]u-**-xa[1MA]:..58_:A (0.048) | 14 (0.018) A:..55_:[PSU]Px**+xG[..G]:..18_:A (0.025) x 16 (0.007) A:..38_:[..A]A-*---c[OMC]:..32_:A (0.008) | 17 (0.021) A:..39_:[PSU]P-*---A[..A]:..31_:A (0.006) | 22 (0.008) A:..44_:[..A]Ax*---g[M2G]:..26_:A (0.010) | Стабилизирующие связи: (0.011) A:..54_:[5MU]u-**-xa[1MA]:..58_:A (0.048) | (0.018) A:..55_:[PSU]Px**+xG[..G]:..18_:A (0.025) x Стекинг-взаимодействия в структуре тРНК 1EVV В файле найдены последовательности с наибольшей и с наименьшей площадьми перекрывания: 13 uP/Ga 5.72( 1.89) 0.00( 0.00) 0.00( 0.00) 8.48( 3.73) 14.20( 5.63) 14 PA/UG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
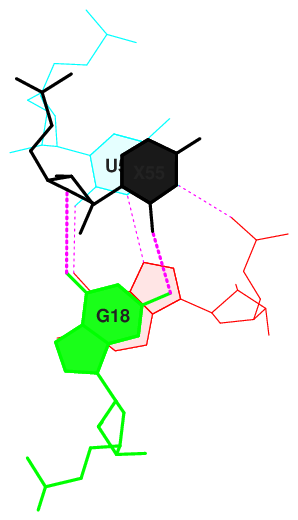
Рисунок N7. Графическое изображение нуклеотидов с наибольшим перекрытием.
Рисунок N8. Графическое изображение нуклеотидов с наименьшим перекрытием.
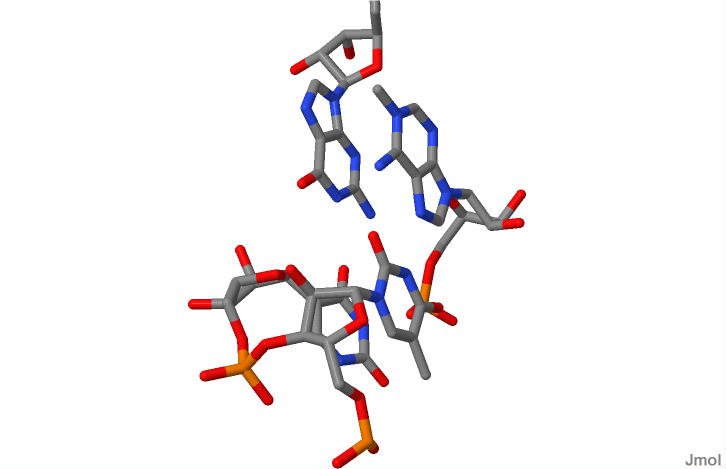
Рисунок N9. Графическое изображение в Jmol нуклеотидов с наибольшим перекрытием.
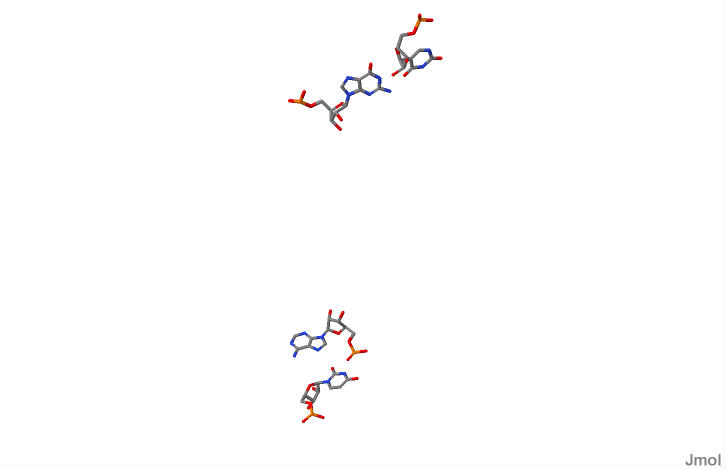
Рисунок N10. Графическое изображение в Jmol нуклеотидов с наименьшим перекрытием. |
 |
 |
|