 |
 |
|
|
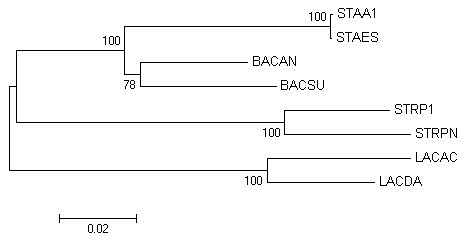
Построение дерева по нуклеотидным последовательностям Было построено дерево тех же бактерий, что и в предыдущих занятиях, но теперь по РНК малой субъединицы рибосомы (16S rRNA). К сожалению, для бактерии Streptococcus pneumoniae не было указано координат последовательности 16S rRNA в записи EMBL. rRNA была найдена при помощи выравнивания генома Streptococcus pneumoniae с последовательностью rRNA Streptococcus pyogenes. Затем было выполненно выравнивание программой Muscle и построено дерево методом Neighbour Joining с бутсрэп-анализом (100 реплик). Результаты показаны ниже. Таблица 1. Набор бактерий из занятия 1.
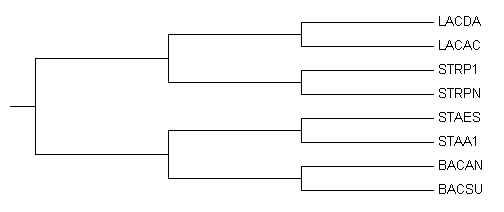
Рисунок 1. Филогенетическое дерево (исходное).
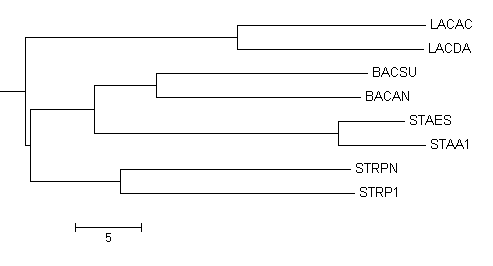
Рисунок 2. Дерево, сделанное методом Neighbour Joining Using % Identity по белкам семейства "пептидил-тРНК гидролазы" (PTH). Укоренено в средней точке
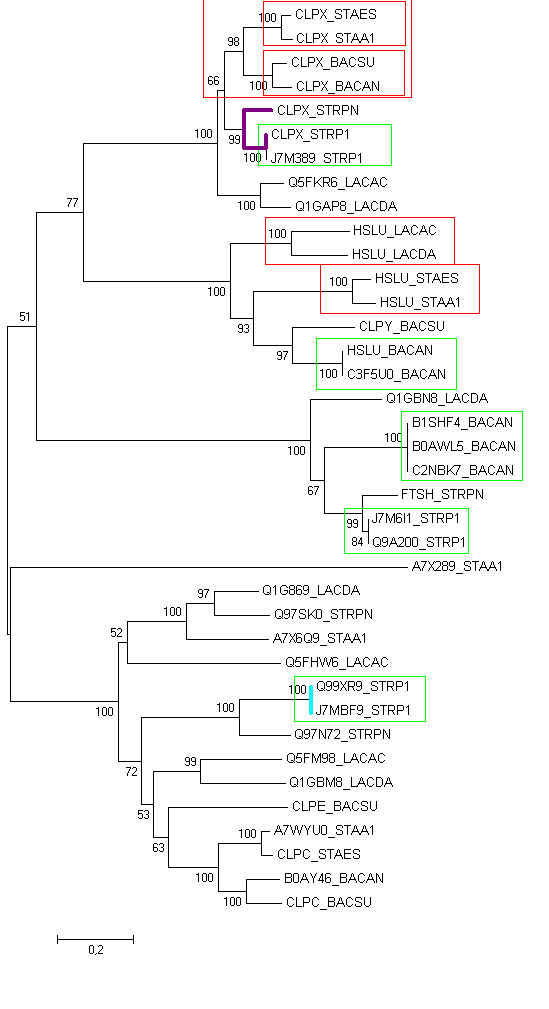
Рисунок 3. Дерево, сделанное методом Neighbour Joining с бутстрэп-анализом (100 реплик) по нуклеотидным последовательностям 16S rRNA. Как можно заметить, полученное дерево не отличается от исходного (различия есть только в иллюстрации ветвей). Построение и анализ дерева, содержащего паралоги Среди изучаемых бактерий были найдены гомологи белка CLPX_BACSU. Гомологи белка брались из файла proteo.fasta. Было выполнено выравнивание программой Muscle, а затем построено дерево методом Neighbour Joining с бутстрэп-анализом (100 реплик).
Рисунок 4. Дерево, построенное на основе гомологов белка CLPX_BACSU. Красным выделены ортологи, а зеленым - одинаковые белки из одного организма (с другим названием). Пример паралогов: CLPX_BACAN и B0AWL5_BACAN, CLPX_STRP1 и J7M6I1_STRP1. Голубым отмечена дупликация гена, а фиолетовым - разделение путей эволюции белков в результате видообразования. |
 |
 |
|