 |
 |
|
|
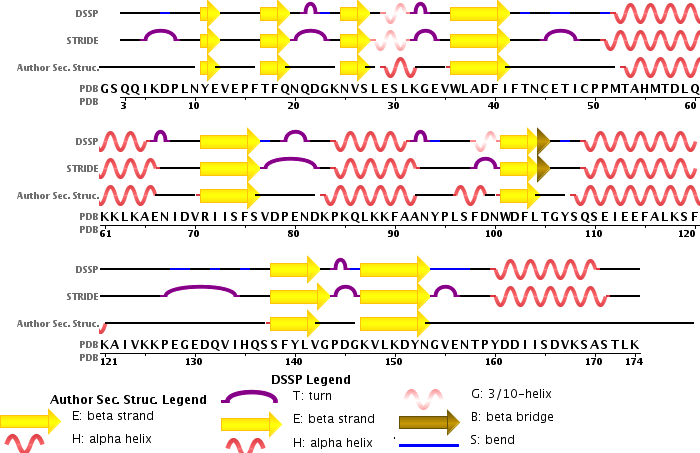
Анализ трехмерных структур Задание d1 DSSP/STRIDE Выбранная структура: 1XZO Вторичная структура была определена с помощью STRIDE и DSSP . На сайте базы данных pdb-структур была найдена удобная картинка с аннотацией вторичной структуры, полученной с помощью DSSP, STRIDE и из PDB-файла (рисунок 1).
Рисунок 1. Аннотация вторичной структуры, взятая с сайта базы данных pdb-структур. Более подробная информация представлена в Excel-таблице. Уже по рисунку 1 видно, что STRIDE показывает более "длинные" участки для вторичной структуры, чем DSSP, а авторы – еще больше, чем STRIDE. Впрочем, участок, соответствующий альфа-спирали с 52-65 остатки (DSSP), получается чуть меньше у авторов (53-66), чем у STRIDE (51-66). SheeP С помощью программы SheeP была получена карта бета-листа структуры 1XZO (рисунок 2).
Рисунок 2. Изображение карты бета-листа для цепи A структуры 1XZO. Красные столбцы обозначают аминокислоты, направленные внутрь ядра, а желтые – аминокислоты, направленные наружу. На основе данной карты было получено изображение бета-листа (рисунок 3).
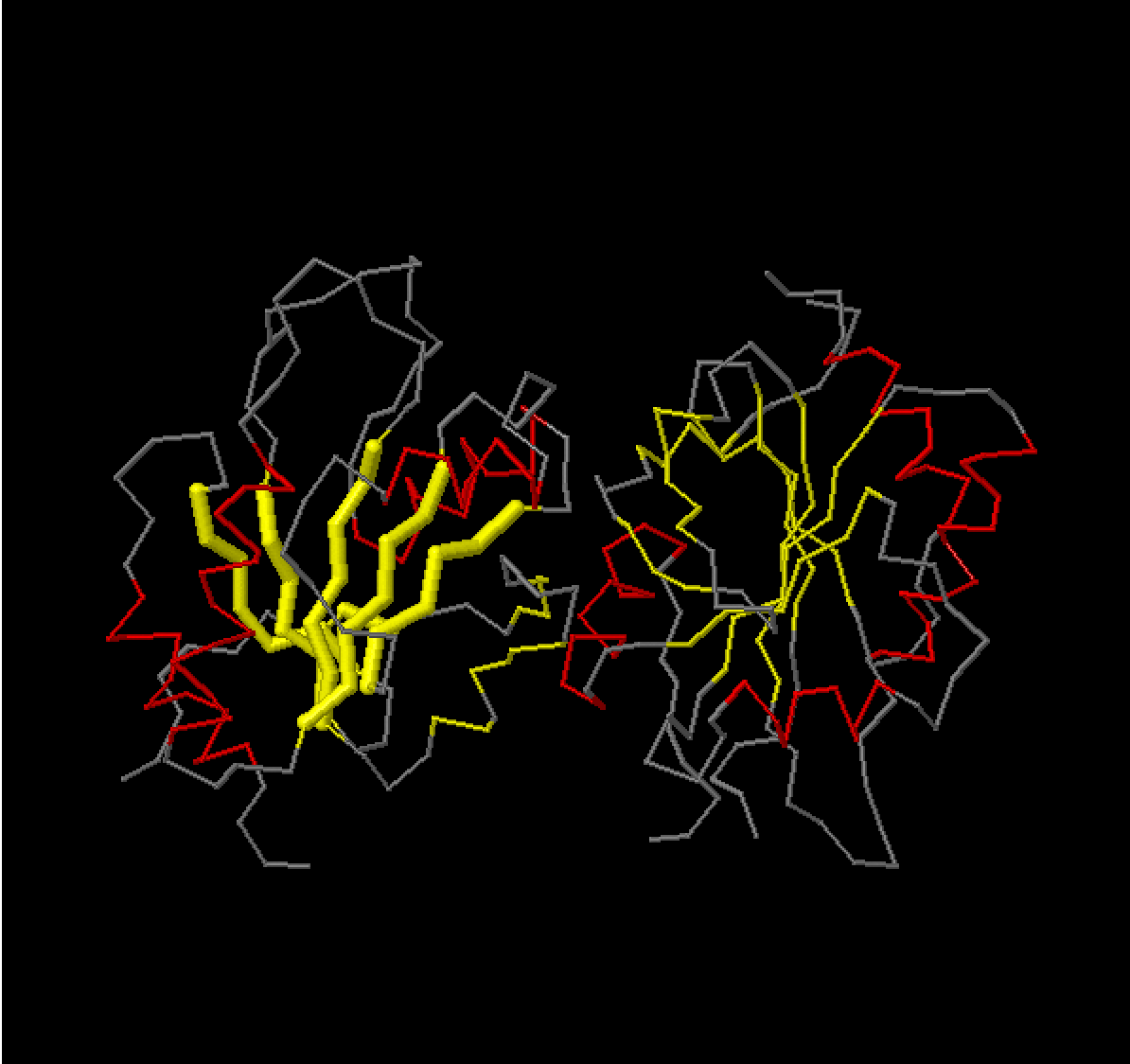
Рисунок 3. Изображение бета-листа цепи A структуры 1XZO. Для второго столбца слева (соответствующего гребню, направленному внутрь ядра) был сделан рисунок 4.
Рисунок 4. Изображение гребня, полученное на основе карты бета-листа (рисунок 2) Была получена карта водородных связей для изучаемой карты бета-листа (рисунок 5).
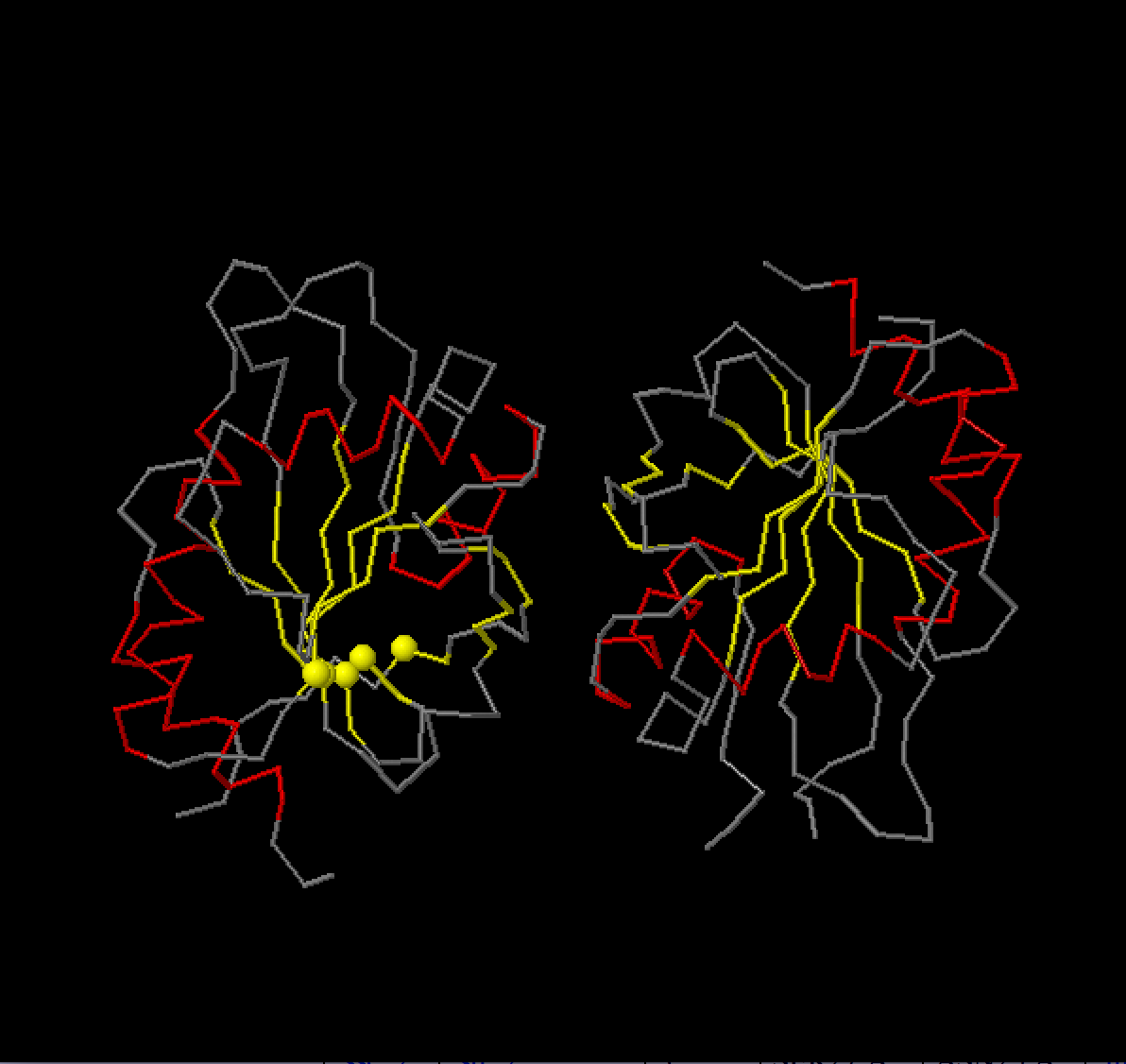
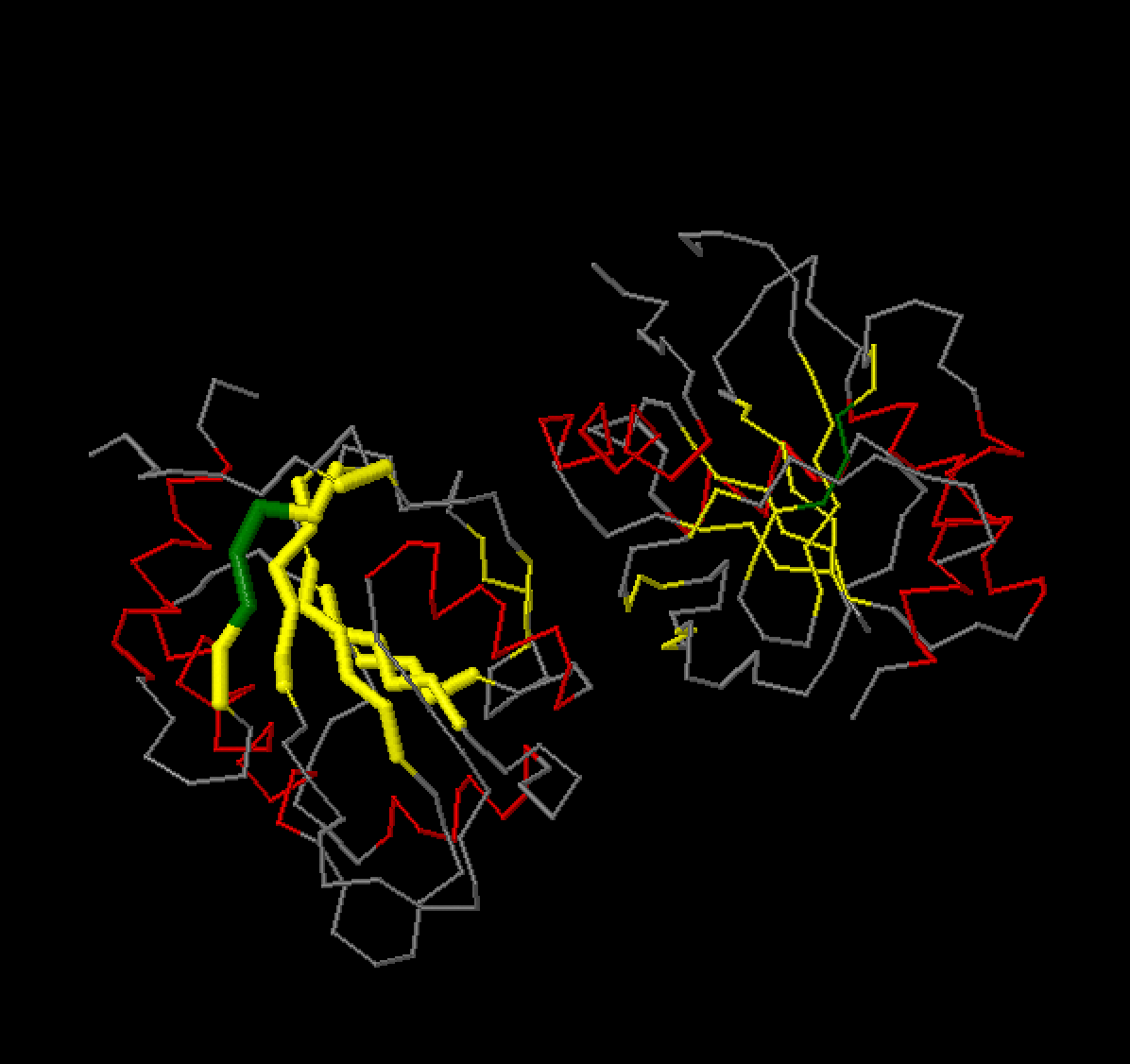
Рисунок 5. Карта водородных связей для бета-листа цепи A структуры 1XZO (рисунок 2). "|, \, /" – водородные связи. "---" – пептидная связь. Как можно заметить, здесь присутствует "выпуклость" (bulge) из Lys150. Было решено его визуализировать (рисунок 6).
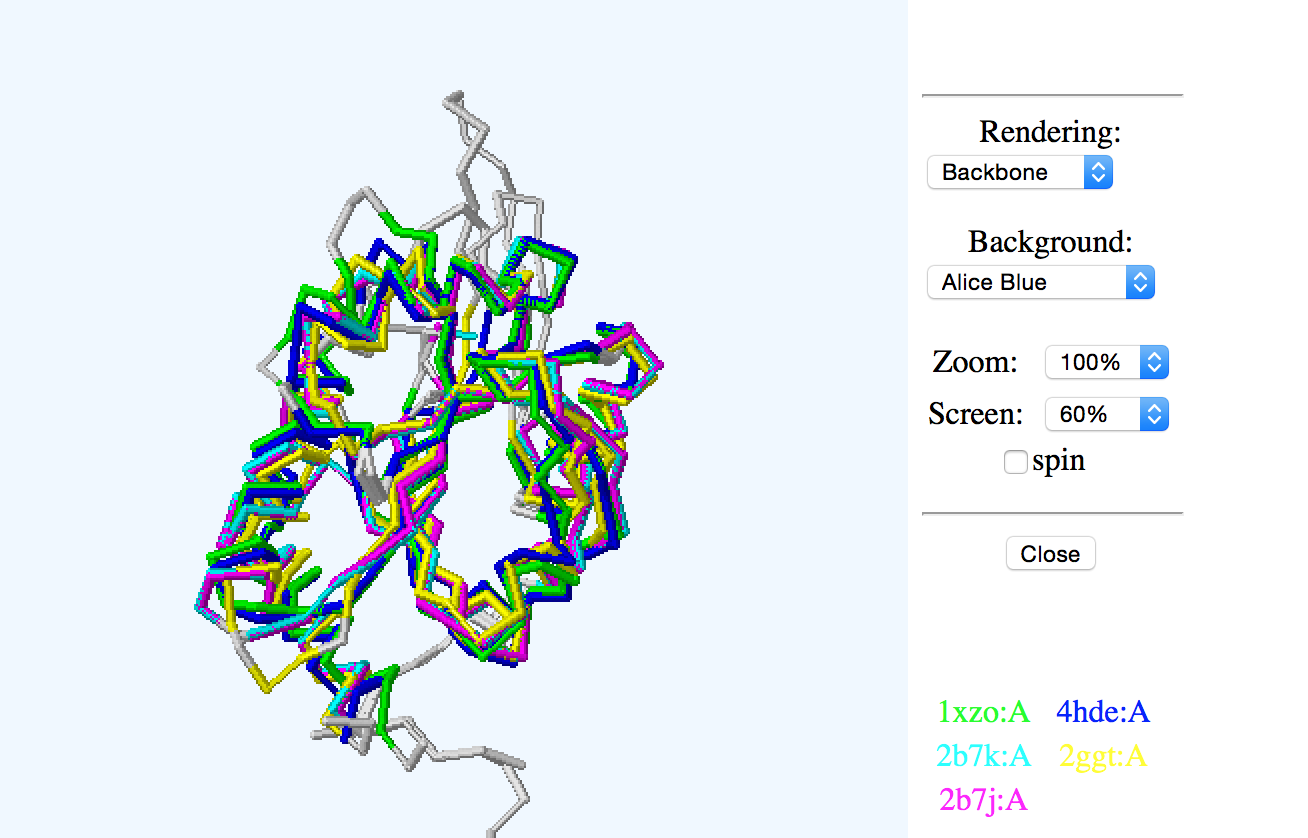
Рисунок 6. Визуализация "выпуклости" (bulge) на в бета-листе. Выпуклость отмечена зеленым. Задание d2 Совмещение структур 1XZO и его гомологов Для работы были выбраны следующие структуры: 4HDE 2B7K 2GGT 2B7J Было получено выравнивание последовательностей по совмещению структур (рисунок 8) и с помощью MUSCLE (рисунок 9).
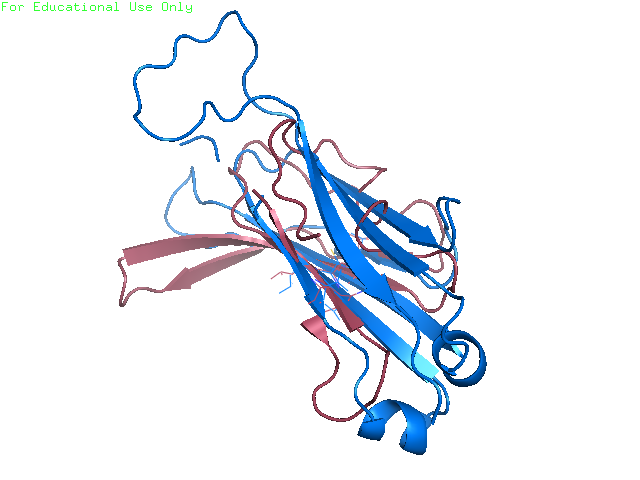
Рисунок 7. Совмещение структур.
Рисунок 8. Выравнивание последовательностей по совмещению структур.
Рисунок 9. Выравнивание последовательностей с помощью MUSCLE. Заметно, что выравнивание по совмещению структур довольно сильно похоже на выравнивание, сделанное с помощью MUSCLE. Однако, во второй строчке выравнивания по совмещению структур можно увидеть столбец L/I, отсутствующий в выравнивании, полученном MUSCLE. На рисунке 10 показаны участки, соответствующие этому столбцу. Как можно заметить, эти позиции весьма сильно отклоняются друг от друга.
Рисунок 10. Совмещение структур, в районе "странного" выравнивания. Поиск структурных гомологов для домена Для работы был выбран рецептор-связывающий домен из структуры 1cii (Colicin IA, 284-384). При поиске с порогом совпадения структур в 70% была найдена структура Colicin I с рецептор-связывающим доменом Colicin IA, что наверное, говорит о том, что домен найден. При поиске с порогом совпадения структур в 60% было найдено 22 структуры, а при пороге в 50% – 3102. Сама структура 1cii находится только при пороге в 30%. Поиск структурных гомологов для домена Для работы были выбрана структура 1OGA, из которой были взяты регион D:118-202 из альфа-цепи и E:119-245 из бета-цепи. Были построены карты бета-листов для региона из альфа-цепи (рисунок 11) и из бета-цепи (рисунок 12).
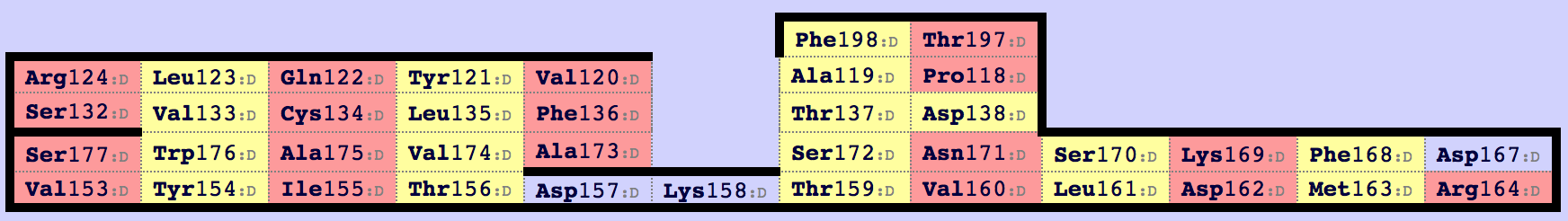
Рисунок 11. Карта бета-листа альфа-цепи.
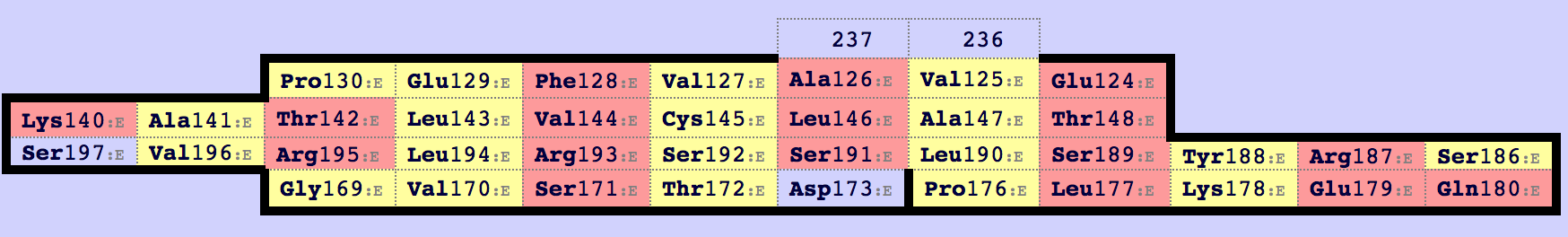
Рисунок 12. Карты бета-листов бета-цепи. Как можно заметить, для бета-цепи существует две карты бета-листов. Судя по количеству тяжей, только первая карта соответствует бета-листу из цепочки альфа. Будем работать с ней. В карте бета-листа из альфа-спирали существует только один цистеин, Cys134, как, впрочем и в первой карте для бета-цепи (Cys145). Совместим структуры в PyMol, используя следующие команды: pair_fit \ alpha and resi 134 and (name CA), beta and resi 145 and (name CA), \ alpha and resi 122 and (name CA), beta and resi 127 and (name CA), \ alpha and resi 175 and (name CA), beta and resi 192 and (name CA), \ alpha and resi 155 and (name CA), beta and resi 172 and (name CA), \ alpha and resi 133 and (name CA), beta and resi 144 and (name CA), \ alpha and resi 135 and (name CA), beta and resi 146 and (name CA) По ссылке можно скачать совмещение, полученное в PyMol. На рисунке 13 можно рассмотреть совмещение выбранных структур.
Рисунок 13. Совмещение альфа- и бета-структур из 1OGA по бета-листам. Синим отмечена структура бета-цепи, а красным – альфа-цепи. Из рисунка хорошо видно, что ход полипептидной цепи совпадает у обеих бета-листов, что говорит о сходстве топологий. Задание d3 Гидрофобные кластеры в 1XZO С помощью Clud были найдены гидрофобные кластеры в 1XZO при порогах расстояния 4 Å, 4.5 Å, 5 Å, 5.5 Å и при пороге размера кластера 10 (рисунки 14-17).
Рисунок 14. Изображение гидрофобных кластеров при пороге расстояния 4 Å.
Рисунок 15. Изображение гидрофобных кластеров при пороге расстояния 4.5 Å.
Рисунок 16. Изображение гидрофобных кластеров при пороге расстояния 5 Å.
Рисунок 17. Изображение гидрофобных кластеров при пороге расстояния 5.5 Å. В целом, 1XZO имеет в себе только один домен, домен тиоредоксина (Thioredoxin domain). Однако, если присмотреться к структуре 1XZO, то можно заметить, что у него присутствует бета-лист, делящий белок пополам. Таким образом, в 1XZO можно визуально увидеть два больших гидрофобных домена (субдомена). К примеру, на рисунке 16. Впрочем, на второй цепи можно заметить еще один гидрофобный кластер. Гидрофобные кластеры на интерфейсе Для работы был выбран белок, образующий не совсем димер и не совсем интерфейс двух цепочек: фибриноген (PDB: 1M1J). С помощью Clud были найдены гидрофобные кластеры на интерфейсе нескольких цепей (рисунок 18) при пороге расстояния в 4.3 Å и при пороге кластеров 3.
Рисунок 18. Гидрофобные кластеры на интерфейсах цепей в фибриногене. Как можно заметить, даже между тремя цепочками возможно образование гидрофобных кластеров. Задание d4 Для работы был взят комплекс пуринового репрессора PurR (PDB: 2PUB) в комплексе с ДНК.
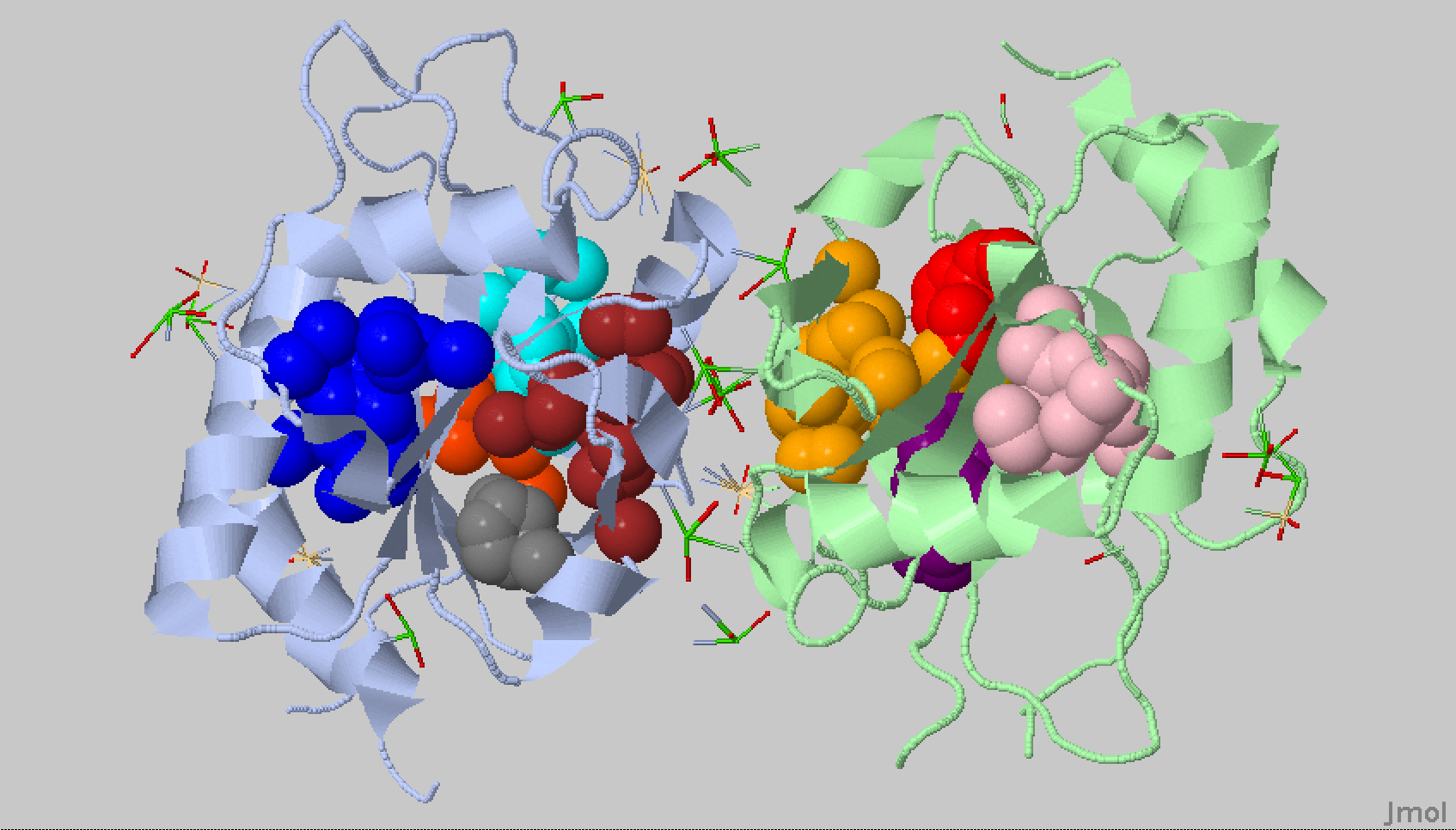
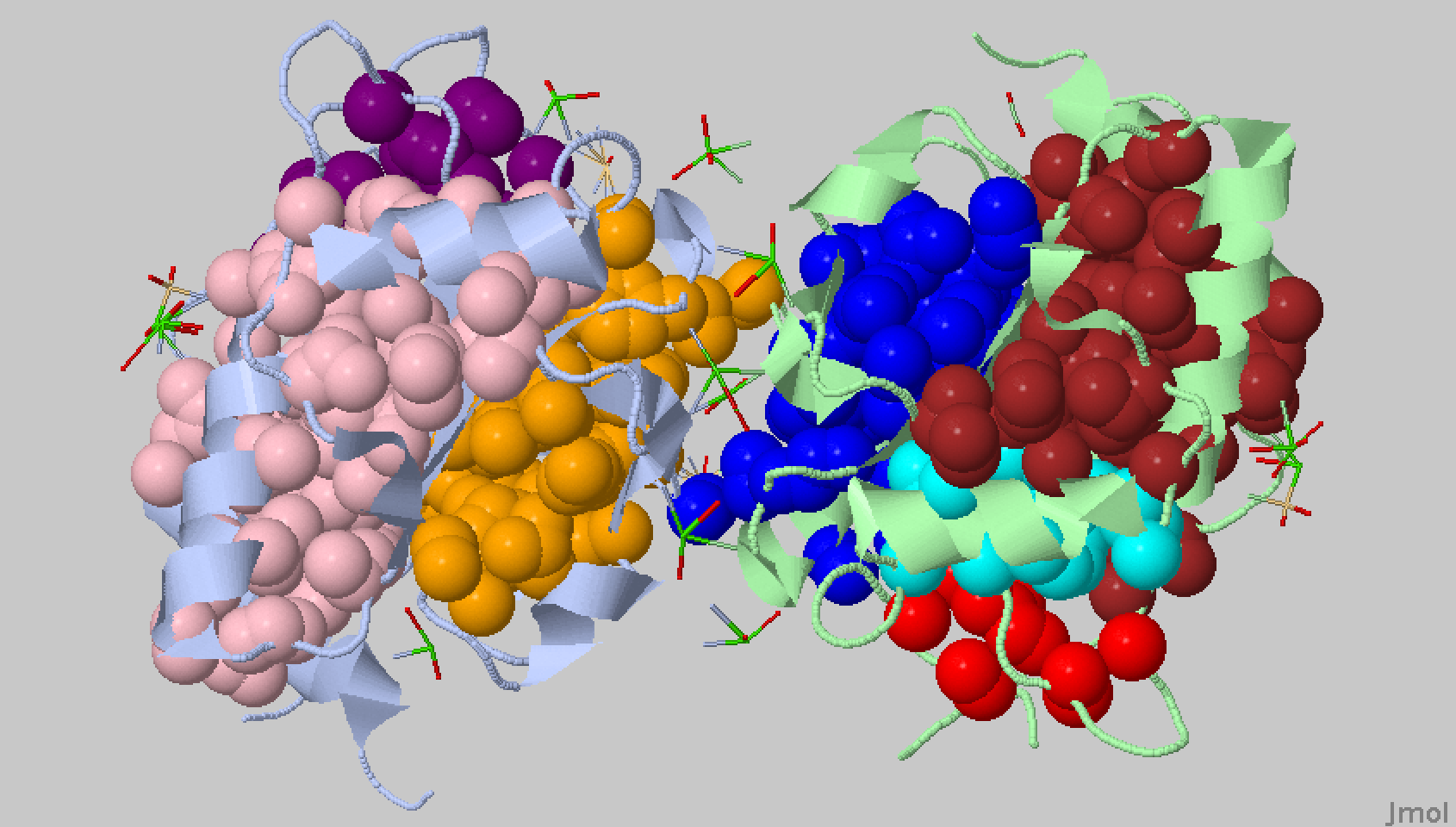
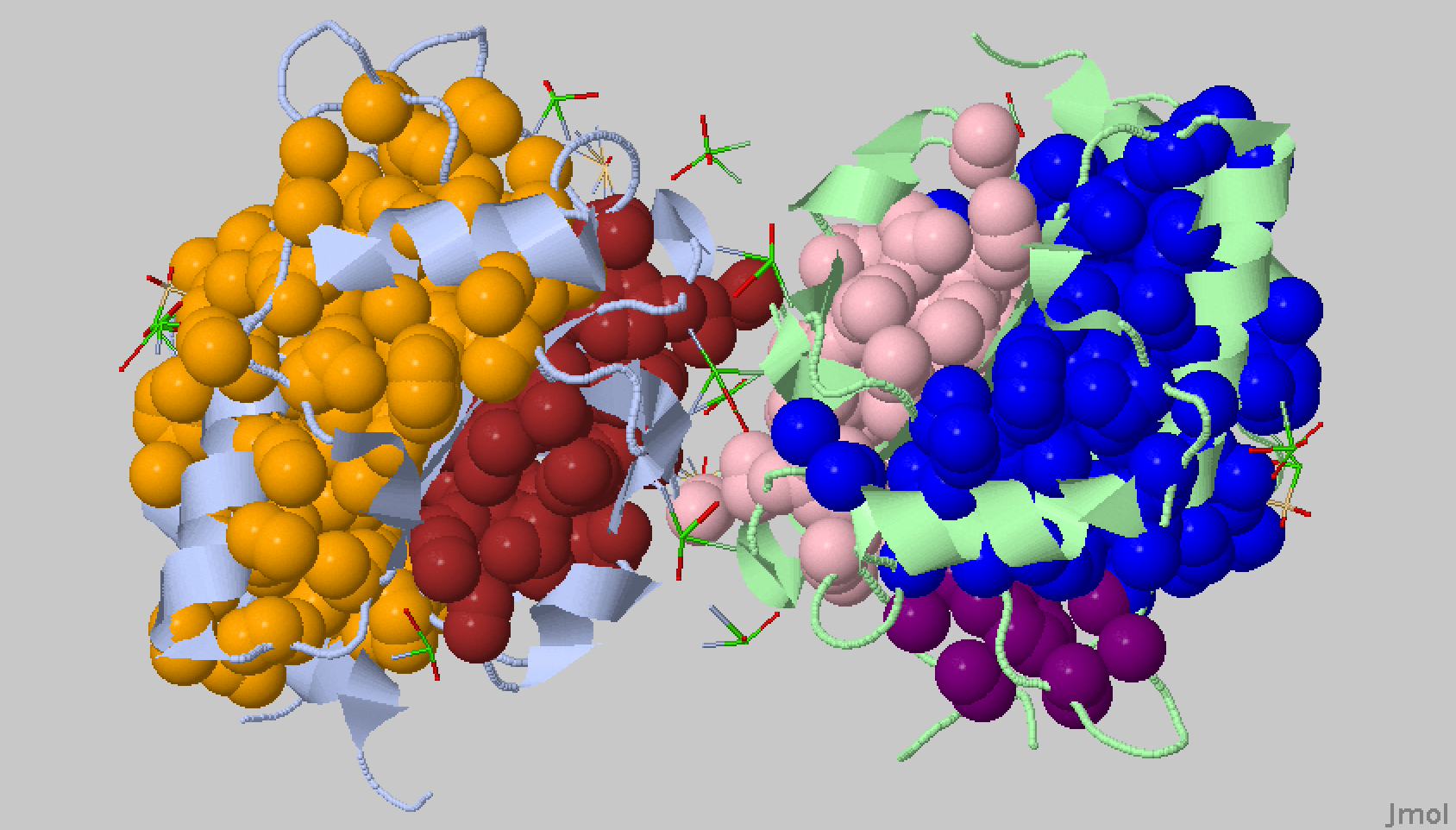
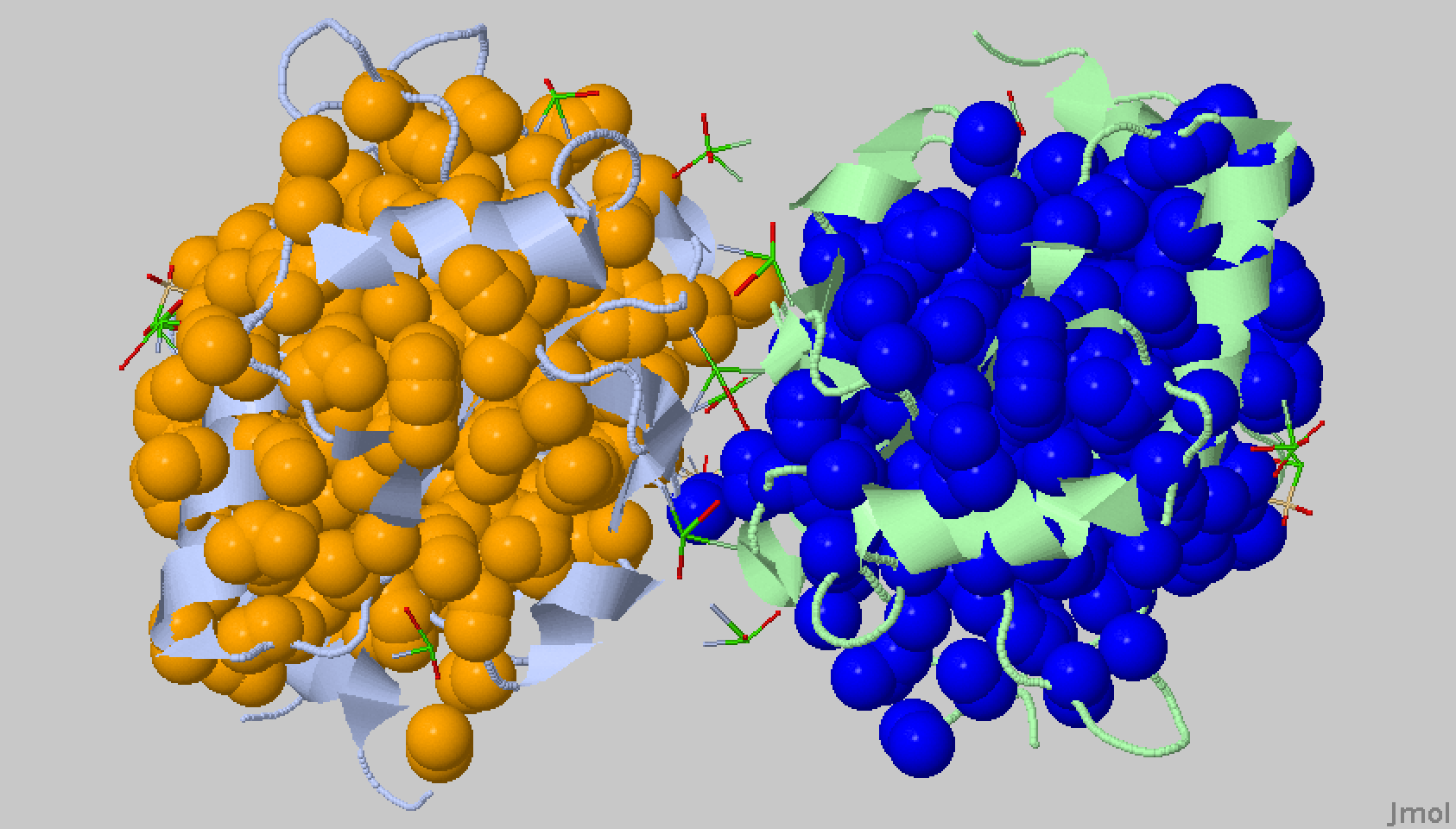
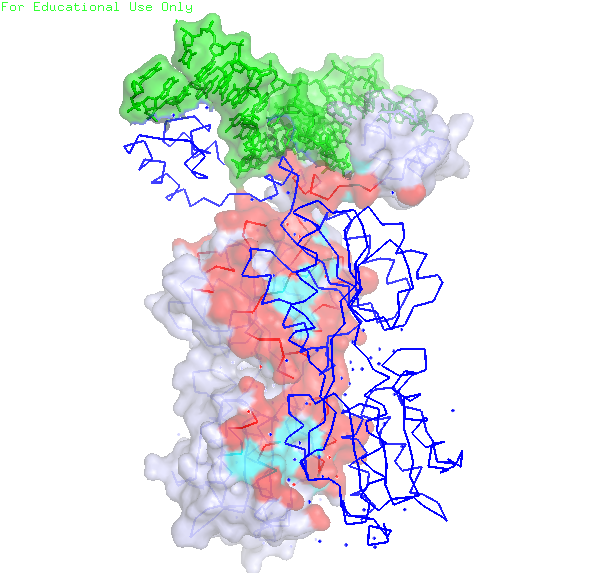
Рисунок 19. Поверхность контакта мономера белка с симметричным мономером. Поверхность контакта выделена красным.
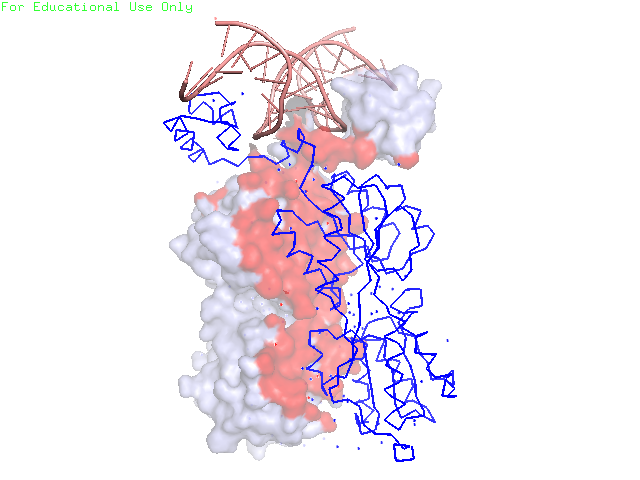
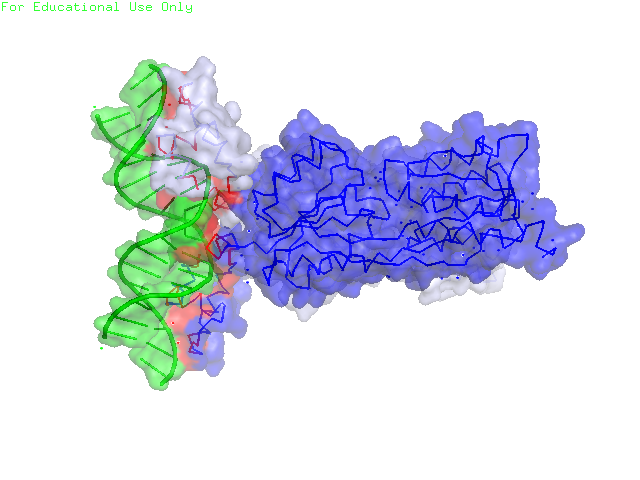
Рисунок 21. Поверхность контакта ДНК с димером белков. Поверхность контакта выделена красным. Поверхность ДНК – зеленым, поверхности белков – синим и сизым.
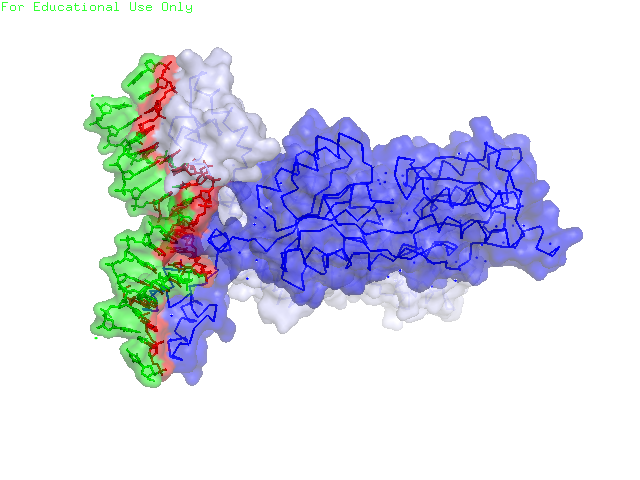
Рисунок 22. Поверхность контакта димера белков с двойной спиралью ДНК. Поверхность контакта выделена красным. Поверхность ДНК – зеленым, поверхности белков – синим и сизым. С помощью сервиса CluD были определены гидрофобные кластеры объемом не менее 10 атомов при пороге в 4.5 Å на интерфейсе мономеров белка в том же комплексе. В файле указаны все найденные гидрофобные кластеры (не только в интерфейсе). Остатки из гидрофобных кластеров визуализировали на поверхности контакта мономера белка с симметричным мономером (рисунок 23).
Рисунок 23. Поверхность контакта мономера белка с симметричным мономером. Поверхность контакта выделена красным. Остатки, участвующие еще и в образовании гидрофобных кластеров, выделены голубым. Задание d5 Для работы была выбрана структура пируваткиназы (PDB: 2g50). Таблица 1. Различные классификации доменов в PK.
SCOP и ECOD нашли одинаковые границы доменов пируваткиназы. CATH выбрал домены иначе, но, в принципе, похожим образом. Различия лишь в определении первого домена (который, видимо, соответствует третьему домену в SCOP и ECOD). Pfam нашел только два домена, с совсем другими границами. Задание d6 Поиск последовательностей всех белков, структуры которых определены методом электронной микроскопиии На сайте www.rcsb.org был произведен поиск (Advanced Search->Methods->Experimental Method->ELECTRON MICROSCOPY) всех структур, определенных методом электронной микроскопии. Всего было найдено 933 структуры. Результаты были скачаны в виде последовательностей в формате FASTA: task6_5_fasta.txt. Сравнение списков структурных гомологов, определенных PDBeFold и jFATCAT. Для структуры 1XZO с помощью PDBeFold с параметрами по умолчанию было найдено 358 гомологов. jFATCAT же, напротив, не нашел гомологов. Было предложено использовать цепь 4HDE:A, которая имеет более чем 50% идентичность в последовательности. Для этой цепи был найден 151 гомолог с помощью jFATCAT и было найдено 265 гомолога с помощью PDBeFold. Некоторые гомологи нашлись как с помощью PDBeFold, так и с помощью jFATCAT, к примеру: 3ME7, 4BPY, 4GRF, 3KEB. Некоторые гомологи нашлись только в jFATCAT, но не нашлись в PDBeFold (D2B7, 2K6V, 3IA1, 4EO3). |
 |
 |
|