- вес совпадения: 2
- вес замены: -1
- штраф за делецию: -2
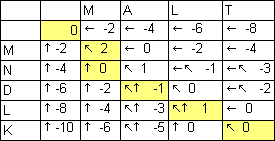
seq1:M_ALT seq2:MNDLK

seq1:M_ALT seq2:MNDLK
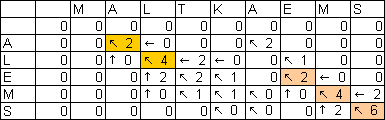
seq1:MALTKAEMS seq2:____ALEMS
seq1:MALTKAEMS seq2:_ALEMS___
Глобальные выравнивания последовательностей из myprot.fasta и thirdprot.fasta с помощью программы needle при разном наборе параметров:
myprot.fasta — аминокислотная последовательность белка IHFA_ECOLI
thirdprot.fasta — искусственно созданная последовательность, склеенная из двух
небольших (10-12 букв каждый) участков аминокислотной последовательности белка
IHFA_ECOLI
| Штраф за открытие делеции - 10 |
IHFA_ECOLI 1 MALTKAEMSEYLFDKLGLSKRDAKELVELFFEEIRRALENGEQVKLSGFG 50
| | : : | | ||
IHFA_ECOLI 1 L-F-D--K-L--G----LS--- 8
IHFA_ECOLI 51 NFDLRDKNQRPGRNPKTGEDIPITARRVVTFRPGQKLKSRVENASPKDE 99
. || .||||||||||||
IHFA_ECOLI 9 K---RD---APGRNPKTGEDIP 24
|
| Штраф за открытие делеции - 1 |
IHFA_ECOLI 1 MALTKAEMSEYLFDKLGLSKRDAKELVELFFEEIRRALENGEQVKLSGFG 50
||||||||||||
IHFA_ECOLI 1 LFDKLGLSKRDA--------------------------- 12
IHFA_ECOLI 51 NFDLRDKNQRPGRNPKTGEDIPITARRVVTFRPGQKLKSRVENASPKDE 99
||||||||||||
IHFA_ECOLI 13 ----------PGRNPKTGEDIP 24
|
При сравнении двух вышеуказанных выравниваний видно, что количество гэпов во
втором выравнивании существенно больше (из-за маленького штрафа за открытие
делеции).
В итоге повышается процент идентичности последовательностей (увеличивается
количество совпавших аминокислотных остатков).
В первом же выравнивании учитывается большая вероятность замен одних аминокислот на другие в соответствующих последовательностях, чем появления в них дополнительных делеций, что более вероятно в природе.