На заглавную страницу
На главную страницу четвертого семестра
Исследование филогенетического дерева MIP из таксонов Lactobacillales, Rhizobiales
Семейство MIP (Major Intrinsic Protein, идентификатор Pfam PF00230) объединяет белки, которые в основном демонстрируют два разных канальных свойства:
- Специфичный транспорт воды (белки - аквафорины);
- Транспорт небольших незаряженных растворенных в воде веществ (например, глицерол транспортируется переносчиком глицерола);
Составление выборки аминокислотных последовательностей:
С помощью SRS в БД UniProt для заданной выборки найдены аминокислотные последовательности.
Выборка состоит из 3-х частей:
- белки семейства MIP (PF00230) из таксонов: Lactobacillales, Rhizobiales
Запрос в SRS:
"([uniprot-DBxref_:PF00230*] & ([uniprot-Taxonomy:Lactobacillales*] | [uniprot-Taxonomy:Rhizobiales*]))"
Найдено 110 белков.
Эта часть выборки была отфильтрована:
- Из разных штаммов одного вида бактерий был выбран один штамм, в котором больше всего белков данного семейства.
- Фрагменты последовательности содержали не менее 200 а.о.
После обработки осталось 63 белка.
- все белки семейства MIP (PF00230) из архей
Запрос в SRS:
"([uniprot-DBxref_:PF00230*] & [uniprot-Taxonomy:archae*])"
Найдено 11 белков.
- как внешняя группа (outgroup) - Q65YQ3_CHICK, Q5W916_BOMMO, AQP3_HUMAN
Запрос в SRS:
"(([uniprot-EntryName:Q65YQ3_CHICK*] | [uniprot-EntryName:Q5W916_BOMMO*]) | [uniprot-EntryName:AQP3_HUMAN*])"
Найдено 3 белка.
В результате было получено 3 файла:
- Последовательности выборки, сохраненные в формате FASTA2Seq, можно посмотреть здесь;
- Последовательности так называемой внешней группы (outgroup) по отношению к данной выборке — последовательности эукариотических белков;
- Список организмов, к которым относятся белки выборки, можно посмотреть здесь.
Построение филогенетического дерева:
- было построено множественное выравнивание белков всех 3 групп с помощью программы emma (параметры были выбраны по умолчанию)
- затем программой eprotdist с параметрами по умолчанию по множественному выравниванию была создана квадратная матрица попарных расстояний между белковыми последовательностями
- при помощи программы eneighbor с параметрами, взятыми по умолчанию, по имеющейся матрице попарных расстояний были созданы скобочная формула дерева и его графическое изображение в виде филограммы (метод Neighbor-Joining). Дерево получилось неукорененное.
- для визуализации изображения была использована программа Gmaster.
Синим цветом выделена внешняя группа, красным — археи.
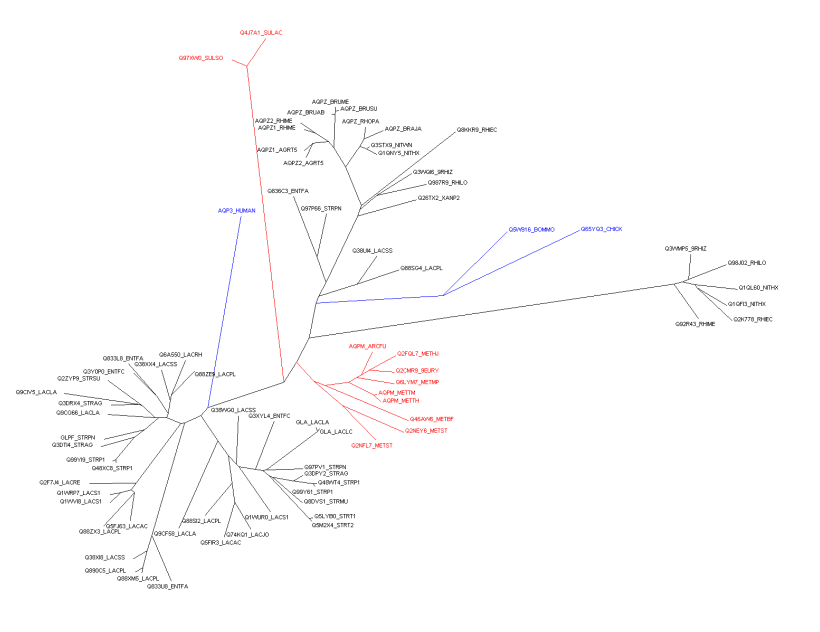
Дерево в полный размер.
Ни внешняя группа, ни белки архей не составляют единых ветвей. Между ними вклиниваются бактериальные белки. Данный пример показывает, что филогенетическое и таксономическое деревья не обязательно совпадают.
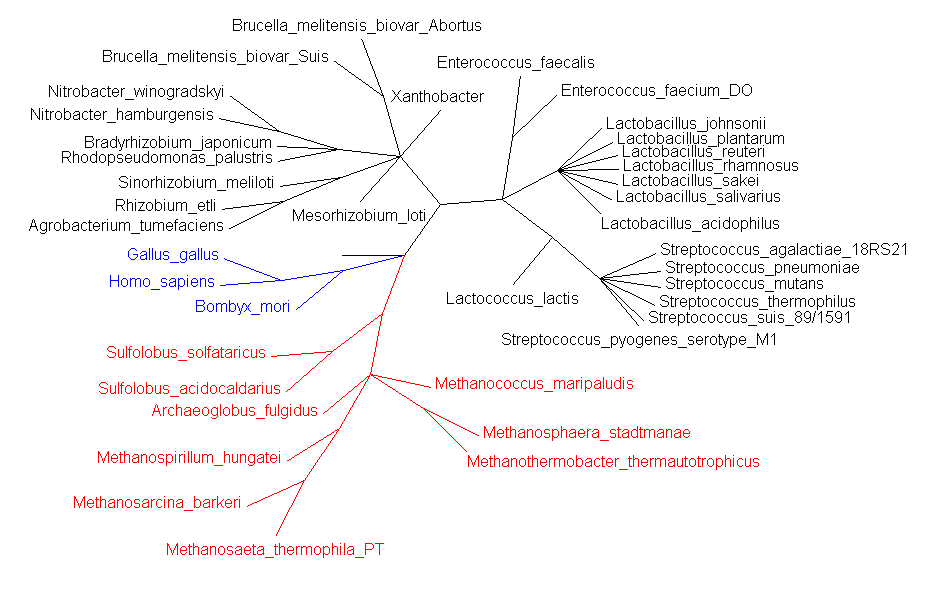
Построение такосономического дерева:
Получено с помощью сайта NCBI.
К сожалению, не все организмы были опознаны, поэтому список организмов был отредактирован.
Синим цветом выделена внешняя группа, красным — археи.
Анализ дерева:
Необходимо выделить паралоги и ортологи.
Ортологи — это гомологи, возникшие в результате видообразования, то есть это белки в разных организмах (чем более далекие, тем лучше), имеющие одну функцию.
Паралоги — это гомологи, возникшие в результате дупликации одного и того же гена в конкретном организме (то есть белки, гены которых появились из одного путем дупликации, но благодаря расхождению и мутациям эти белки имеют разные функции).
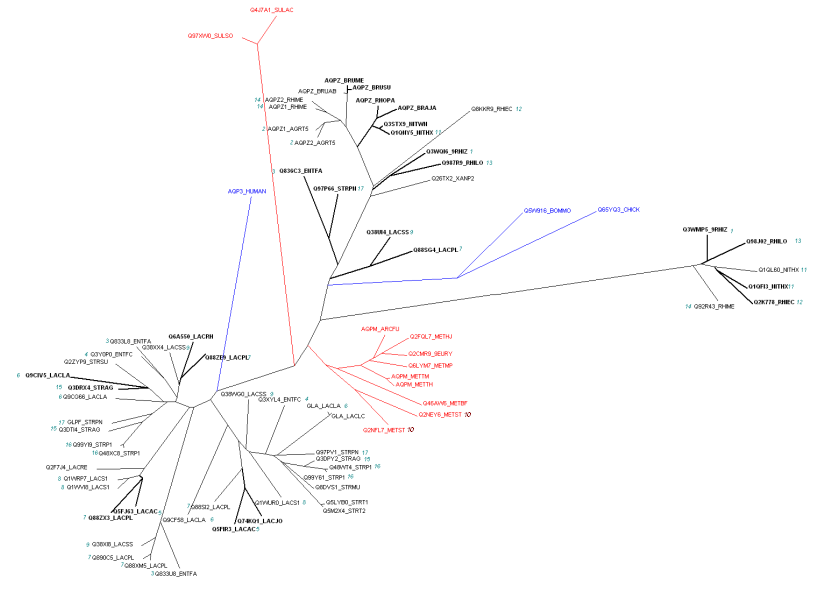
- Для паралогов был составлен список всех белков дерева, отсортированный по организмам.
Белки одного организма, являются между собой паралогами. Oдна группа паралогов обозначена одной цифрой.
Хорошими считались те паралоги, которые находятся как можно дальше друг от друга на дереве, потому что это говорит о сильном расхождении последовательностей (а значит, функций).
Список, в соответствии с которым были найдены паралоги, здесь.
Итак, нашлось 16 групп паралогов. И еще одна (она фигурирует под номером 10) среди архей.
Группа под номером 2 — плохая. Белки находятся на одной развилке.
В группах под номерами: 6, 7, 11, 14, 16 часть белков также отстоят недалеко друг от друга.
- Для ортологов искали "развилки" на дереве (для уверенности рассматривались только пары), после чего проверяли, являются ли организмы близкородственными.
Ортологами считались те белки, которые принадлежат разным видам (случаи ENTFA-ENTFC, LACLA-LACLC, LACRE-LACRH, LACS1-LACSS, METTH-METTM, STRT1-STRT2 не годятся, потому что это подвиды одного вида).
Лучшими ортологами считались белки, принадлежащие максимально далеким организмам. Ортологи и соответствующие им ветки выделены жирным курсивом.
"Видовые списки" можно найти здесь.
Итак, нашлось 12 групп ортологов.
Вид дерева в полный размер можно посмотреть здесь.
В начало
©
Сахарова Ирина, 2006.



