A- и B-формы ДНК. Структура РНК
Задание 1. Строим модели структур А-, В- и Z-форм ДНК.
Для выполнения данного задания я построила А-, В- и Z-формы ДНК с помощью программы fiber пакета 3DNA (пакета программ для анализа и простейшего моделирования структур нуклеиновых кислот). Так как данный пакет программ работает под операционной системой Linux, было необходимо воспользоваться программой Putty.
Было построено 2 дуплекса ДНК, последовательность одной из нитей которых представляет собой 5 раз повторенную последовательность "gatc" (A- и B-формы); Z-форма представлена 5 раз повторенной парой G-C (программа fiber позволяет строить только такую Z-форму ДНК).
Результатом выполнения данного задания являются файлы с полученными структурами форм ДНК в формате .pdb: А-форма, B-форма, Z-форма.
Задание 2. Вспоминаем JMol.
В данном задании требовалось работать со структурами нуклеиновых кислот средствами программы JMol.
Упражнение 1.
Целью выполнения этого упражнения было научиться выделять разные атомы и химические группировки, используя предопределенные множества JMol. Для выполнения упражнения была взята структура А-формы ДНК в формате .pdb: А-форма.
Для наиболее удобной визуализации молекулы и требуемых атомов и группировок я использовала Java-апплет, который последовательно демонстрирует следующие структуры:
- сахарофосфатный остов ДНК (изображен проволочной моделью светло-голубого цвета)
- все нуклеотиды (А - темно-оливкового цвета, G - темно-розового, С - светло-бежевого, Т - золотистого; все изображены картонками)
- все нуклеотиды с аденином (выделены зеленым цветом и шаро-стержневойй моделью)
- атом N7 во всех гуанинах (выделены крупным размером и красным цветом; атом N7 первого по последовательности гуанина выделен синим цветом и подписан)
|
После того, как структура загрузится, надо нажать две кнопки:
1. Запустить скрипт: 2. Продолжить исполнение скрипта: |
Упражнение 2.
В данном упражнении было необходимо получить файлы в формате .pdb структур т-РНК и ДНК-белкового комплекса с сайта PDB. Затем из вариантов для ДНК-белкового комплекса требовалось отобрать подходящий: структуру, содержащую двухцепочечную ДНК.
Были получены файлы со структурами тирозил-тРНК дикого типа из организма Thermus thermophillus и ДНК-белкового комплекса инициаторного белка связывания в Trychomonas vaginalis, связанного с ферредоксиновым инициаторным сайтом ДНК.
Упражнение 3.
В этом упражнении требовалось проверить полученные структуры ДНК и РНК на наличие разрывов. Для этого нужно было рассмотреть проволочные модели только нуклеиновых кислот из файлов, полученных с сайта PDB.
В молекуле ДНК из ДНК-белкового комплекса разрывов не было обнаружено (Рис. 1).
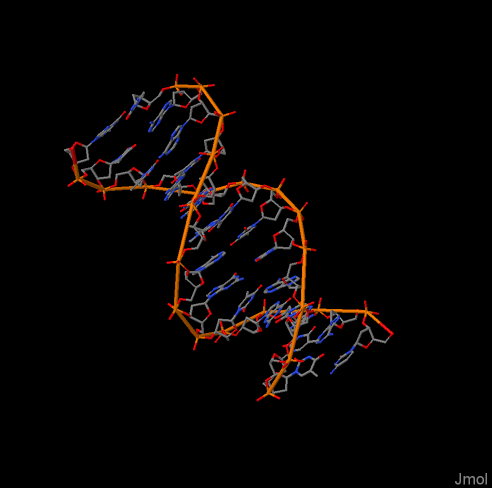
Рис. 1. Изображение ферредоксинового инициаторного сайта ДНК ; разрывов не обнаружено.
В молекуле т-РНК было обнаружено 2 разрыва. Это может быть связано с тем, что рентгеноструктурный анализ не позволил определить координаты части атомов.
На Рис. 2 можно видеть изображение тРНК в проволочной модели, нуклеотиды, между которыми обнаружен разрыв, выделены желтым цветом.
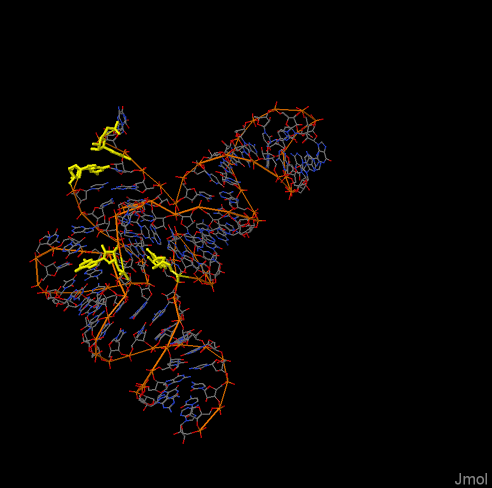
Рис. 2. Изображение тРНК из Thermus thermophillus в виде проволочной модели; разрывы обнаружены между нуклеотидами, обозначенными желтым цветом.
На Рис. 3 для более удобного изображения разрывов молекула тРНК показана ленточной моделью, нуклеотиды, между которыми обнаружен разрыв, выделены желтым и светло-голубым цветами.

Рис. 3. Изображение тРНК из Thermus thermophillus в виде ленточной модели; разрывы обнаружены между нуклеотидами, обозначенными желтым и светло-голубым цветами.
Также были получены файлы формата .pdb с координатами атомов только тРНК и только ДНК.
Задание 3. Сравним структуры разных форм ДНК.
В данном задании требовалось сравнить по различным параметрам структуры трех форм ДНК с использованием средств JMol.
Упражнение 1.
В этом упражнении было необходимо научиться определять большую и малую бороздки в структуре молекулы ДНК. Для этого были взяты структуры А- и В-формы ДНК из файлов в формате .pdb: B-форма, A-форма.
На Рис. 4a-b можно увидеть расположение большой и малой бороздок.
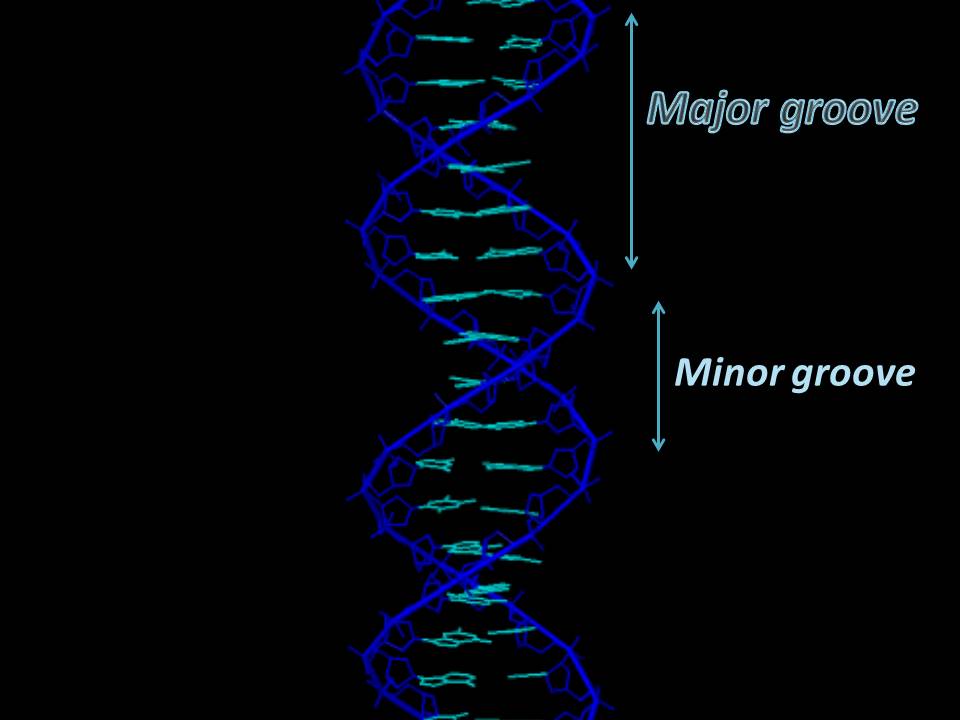
Рис. 4a. Изображение молекулы В-формы ДНК; обозначены большая и малая бороздки.
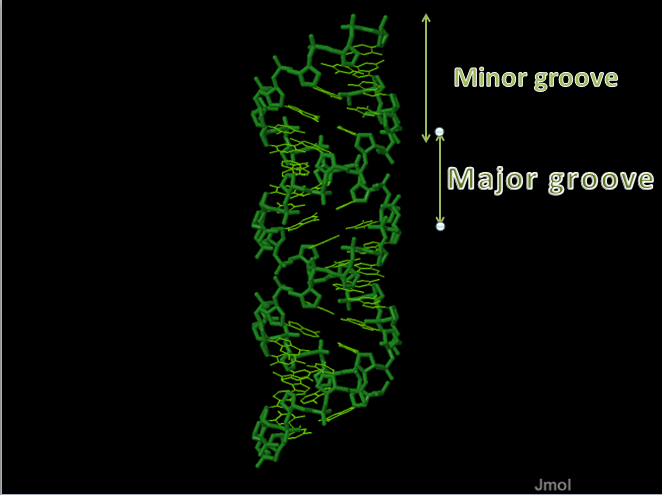
Рис. 4b. Изображение молекулы A-формы ДНК; обозначены большая и малая бороздки.
Несмотря на то, что большая бороздка в А-форме кажется более узкой, чем малая, она глубже.
Далее было необходимо выбрать доставшийся нуклеотид в любом месте молекулы (в моем случае тимин) и определить, какие из атомов основания обращены в сторону большой бороздки, а какие - в сторону малой.
Я выбрала остаток тимидилового нуклеотида под номером 31 и, отобразив в программе JMol, определила, куда обращены атомы основания.
На изображениях, полученных с помощью программы ChemSketch (Рис. 5-6), можно увидеть изображения тимина, где красным цветом обозначены атомы, обращенные в сторону большой бороздки, а синим - в сторону малой в В- и А-формах соответственно.
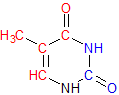
Рис. 5. Изображение 31-го тимина в В-форме ДНК с помощью программы ChemSketch. Красным цветом обозначены атомы, обращенные в сторону большой бороздки, а синим - в сторону малой.

Рис. 6. Изображение 31-го тимина в A-форме ДНК с помощью программы ChemSketch. Красным цветом обозначены атомы, обращенные в сторону большой бороздки, а синим - в сторону малой.
Дополнительно с визуализацией атомов в большой и малой бороздках всех трех форм ДНК можно ознакомиться на Java-апплете (скрипт Grooves).
- В сторону большой бороздки обращены атомы t13.o4, t13.c5m, t13.c6, t13.c4, t13.c5.
- В сторону малой бороздки обращены атомы t13.o2, t13.c2, t13.n3.
- Атом N1 расположен на границе большой и малой бороздок и связан с сахарофосфатным остовом, поэтому его нельзя четко отнести к одной из бороздок.
- В А-форме ДНК в малую и большую бороздки обращены те же атомы (но рувидеть это сложнее, так как А-форма сжата сильнее, поэтому некоторые атомы находятся глубже и хуже видно).
- Z-форму проанализировать не удалось, так как программа fiber построила ее исключительно из остатков G и C, поэтому в ней нет тимина, который я рассматриваю.
В этом упражнении требовалось сравнить основные спиральные параметры разных форм ДНК. Для этого с помощью программы JMol анализировались структуры трех форм ДНК, полученные в Задании 1.
Результатом выполнения задания является Таблица 1.
Таблица 1. Основные спиральные параметры A-, B-, Z-форм ДНК
|
A-форма |
B-форма |
Z-форма |
Тип спирали (правая или левая) |
Правая |
Правая |
Левая |
Шаг спирали (Å) |
28.03 |
33.75 |
43.50 |
Число оснований на виток |
11 |
10 |
12 |
Ширина большой бороздки |
7.98 ([G]25:B.P - [A]10:A.P) |
17.21 ([A]26:B.P - [C]12:A.P) |
18.03 ([C]14:B.P - [C]4:A.P) |
Ширина малой бороздки |
16.83 ([G]25:B.P - [A]18:A.P) |
11.69 ([A]26:B.P - [T]19:A.P) |
7.2 ([G]5:A.P - [G]19:B.P) |
Как видно из Таблицы 1, в А-форме ширина большой бороздки действительно меньше, чем ширина малой, но большая бороздка глубже. Также расположение бороздок в А-форме согласуется с тем, куда обращены атомы 31-го тимина (они должны быть обращены, в целом, в те же бороздки, что и в В-форме, помимо небольших расхождений).
Упражнение 3.
В данном задании предлагалось сравнить торсионные углы в структурах А- и В-форм. Для этого с помощью команды Settings->Torsion JMol нужно было измерить торсионные углы выбранного в упр. 1 нуклеотида (Т[31]). Затем полученные значения требовалось сравнить со значениями, приведенными в презентации.
На Рис. 7 можно посмотреть на примере нуклеотида с основанием А, где какие торсионные углы расположены.
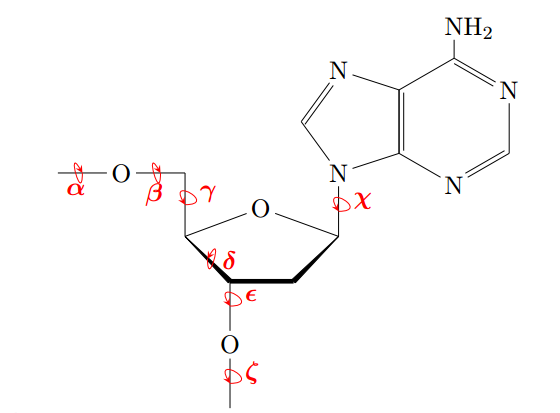
Рис. 7. Изображение торсионных углов на примере нуклеотида с основанием А.
Результатом выполнения задания является Таблица 2.
Таблица 2. Сравнение торсионных углов в А- и В-формах ДНК с данными, приведенными в презентации.
Торсионный угол |
α |
β |
γ |
δ |
ε |
ζ |
χ |
| A-форма из презентации | 62 | 173 | 52 | 88/3 | 178 | -50 | -160 |
| A-форма | 64,1 | 174,8 | 41,7 | 79,1 | -147,8 | -75,1 | -157,2 |
| B-форма из презентации | 63 | 171 | 54 | 123/131 | 155 | -90 | -117 |
| B-форма | 85,9 | 136,3 | 31,2 | 143,3 | -140,8 | -160,5 | -98,0 |
Наибольшее различие между А- и В-формами наблюдается в мере углов δ, ζ, χ, что связано с различной пространственной конформацией сахара в А- и В-формах (в В-форме: C2’-endo, в А-форме: C2’-exo).
Наибольшее расхождение с данными презентации наблюдается по углу ε : значения близки по модулю, но противоположны по знаку.
Задание 4.
В данном задании было нужно определить параметры структур нуклеиновых кислот с помощью программ пакета 3DNA. Так как пакет 3DNA пока работает только со старым форматом PDB, то пришлось воспользоваться программой remediator, которая переводит файлы в старый формат. C помощью программ find_pair и analyze было получено много файлов с информацией. 1 b 2 упражнения выполнены по материалам файлов с расширением .out, 3 упражнение выполнено с помощью данных о стекинг-взаимодействиях из файла stacking.pdb.
Упражнение 1.
В данном упражнении нужно было определить торсионные углы в ферредоксиновом инициаторном сайте ДНК и в тирозил-тРНК. Данные были взяты из файлов тРНК
Со значениями торсионных углов для всех нуклеотидов нуклеиновых кислот, кроме первых и последних (очевидно, что у них не все торсионные углы можно рассмотреть), можно ознакомиться в файле Excel. Средние значения углов представлены в Таблице 3.
Таблица 3. Средние значения торсионных углов в ферредоксиновом инициаторном сайте ДНК и тирозил-тРНК.
Торсионный угол |
α |
β |
γ |
δ |
ε |
ζ |
χ |
| тРНК | -39,2 | 6,3 | 60,5 | 90,9 | -152,7 | -70,8 | -139,9 |
| ДНК | -57,9 | 51,8 | 54,9 | 135,5 | -163 | -92,4 | -108 |
Также были определены "наиболее деформированные" остатки, из всех остальных, то есть нуклеотиды, значения углов которых наиболее отличаются.
Для тРНК: по значениям углов α и γ наиболее выделяются G29 и G23 (цепь 1), G6, G11, C20, G30, U34 (цепь 2) (в основном это нуклеотиды, на которых происходит "смена спирали"
Для ДНК: по значениям углов α, β и γ наиболее выделяется С10 (цепь 1).
Если сравнить данные торсионных углов для тРНК с данными из Таблицы 2, то нетрудно заметить, что тРНК больше всего похожа на А-форму.
Упражнение 2.
В этом упражнении было необходимо научиться определять структуру водородных связей.
Для начала я определила номера нуклеотидов, образующих стебли во вторичной структуре тирозил-тРНК из Thermus thermophilus (Таблица 4).
Таблица 4. Номера нуклеотидов тирозил-тРНК, образующих стебли во вторичной стуктуре.
| Номер стебля | Номер нуклеотида (цепь 1) |
Нуклеотид (цепь 1) |
Номер нуклеотида (цепь 2) |
Нуклеотид (цепь 2) |
| Stem 1 | 1-7 | G, G, G, C, A, G, G | 72-66 | C, C, C, G, U, C, C |
| Stem 2 | 49-52 | G, C, U, G | 65-62 | C, G, A, C |
| Stem * | 47E-G | G, C, C, G | 47B, 46, 45 | C, G, G, C |
| Stem 3 | 23-25 | G, G, G | 12-10 | C, C, C |
| Stem 4 | 27-31 | A, C, G, G, U | 43-39 | U, G, C, C, A |
Было обнаружено 11 неканонических пар: 2 пары с неканоническими основаниями (псевдоуридин - гуанин, 5-метилурацил - 1-метиладенин), 3 пары G-C, 1 пара A-C. Остальные пары между типичными комплементарными основаниями, однако водородные связи в них расположены по-другому.
Также были обнаружены дополнительные водородные связи, удерживающие третичную структуру РНК (это предположение, так как в этом месте расположен разрыв, то неизестно, что здесь было): между G15-C48 и G19-C56; также довольно странные связи Stem * могут служить для стабилизации структуры.
Что еще интересно в этой структуре, так это то, что у нее последние нуклеотиды ACC, которые должны быть свободными для соединения с аминокислотой, являются спаренными и образуют стебель. Это необходимо для того, чтобы не произошла реакция присоединения аминокислоты, катализируемая тРНК-синтетазой, что, в свою очередь, необходимо для получения кристалла для расшифровки структуры при помощи рентгеноструктурного анализа.
Упражнение 3.
В данном задании было необходимо научиться находить возможные стекинг-взаимодействия. Для этого в файле Out я нашла данные о величине площади "перекрывания" 2-х последовательных пар азотистых оснований. Для пары с наибольшими значениями (step12) я получила стандартное изображение стекинг-взаимодействия с помощью программ ex_str и stack2img пакета 3DNA.
Результат выполнения задания можно видеть на Рис. 8. Изображение конвертировано в формат .png с помощью сервиса Ghost.
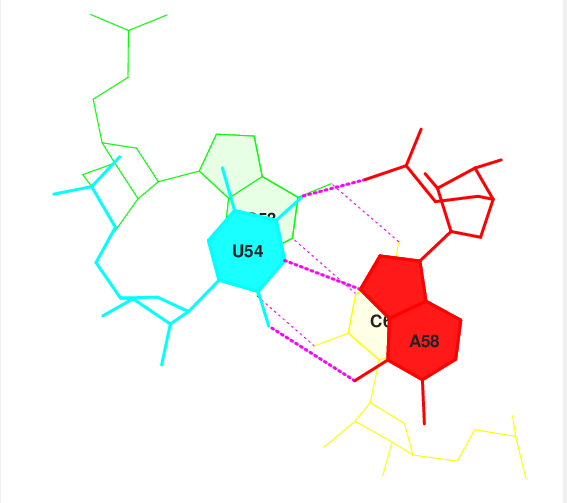
Рис. 8. Изображение стекинг-взаимодействия для набора step12.