Комплексы ДНК-белок
Задание 1. Предсказание вторичной структуры тРНК.
В данном задании я училась предсказывать вторичную структуру моей тирозил-тРНК дикого типа из организма Thermus thermophilus разными способами, а затем сравнить с данными, полученными в прошлом практикуме с помощью программы find_pair.
Для этого мне потребовалась нуклеотидная последовательность тРНК, которую я скачала с сайта сайта PDB (файл в формате fasta). Что интересно, в данном файле все неканонические основания заменены на наиболее сходные по свойствам.
тРНК обладают определенным набором элементов с довольно консервативной структурой (см. Рис. 1), поэтому было бы интересно обнаружить эти элементы в своей структуре.
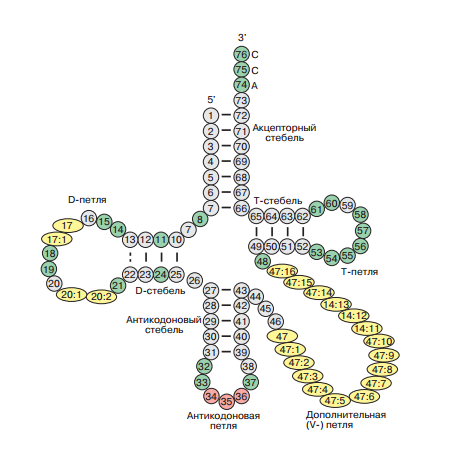
Рис. 1. Универсальная структура тРНК.
Упражнение 1.
В данном упражнении требовалось предсказать вторичную структуру тРНК путем поиска инвертированных повторов.
Для этого я использовала программу einverted из пакета EMBOSS, которая позволяет найти инвертированные участки в нуклеотидных последовательностях. Я попыталась найти возможные комплементарные участки в последовательности тРНК. Для того, чтобы программа сработала, мне пришлось заменить в fasta-файле все U на Т, так как исходно программа работала с ДНК.
При первом запуске с параметрами, заданными по умолчанию, не было получено никаких данных. При запуске с параметром Minimume score threshold, равным 40 и 25, также не было получено результата. При запуске со значением 10 этого параметра был получен 1 акцепторный стебель и тот неверный.
На данный момент лучшим признан 6 запуск программы, который по-прежнему даже не определяет правильно акцепторный стебель. Параметры: Gap penalty [12]: 0, Minimum score threshold [50]: 0, Match score [3]: 3, Mismatch score [-4]: -3. Благодаря наличию гэпов, присутствуют некоторые совпадения с find_pair.
С результатом выполнения задания можно ознакомиться на Рис. 2 и в Таблице 1.
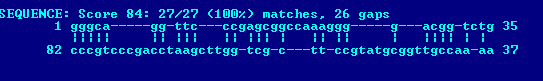
Рис. 2. Результат работы программы einverted: поиск инвертированных повторов в последовательности тирозил-тРНК.
Упражнение 2.
В данном упражнении было необходимо предсказать вторичную структуру, используя алгоритм Зукера (подсчитывает минимальную свободную энергию). Для выполнения этой задачи я воспользовалась web-сервисом. Программа вывела одинаковые результаты после подсчета минимальной свободной энергии и термодинамического предсказания. Результат работы программы катастрофически не совпадает с реальными позициями в структуре (но параметры изменить нельзя, поэтому нет необходимости в последующих запусках). Так, снова не совпадает даже акцепторный стебель, вследствие чего далее не совпадает вся нумерация, кроме D-стебля. Что интересно, данный результат похож на результат работы программы einverted. Также в моей структуре получается 5 стеблей, что согласуется с данными find_pair, но не является типичным для тРНК и усиливает проблемы с несовпадением нумерации. В целом количество и длина стеблей соответствуют эталону, но все нарушает исходно неверная нумерация, начиная с акцепторного стебля.
С результатом можно ознакомиться на Рис. 3 и в Таблице 1.
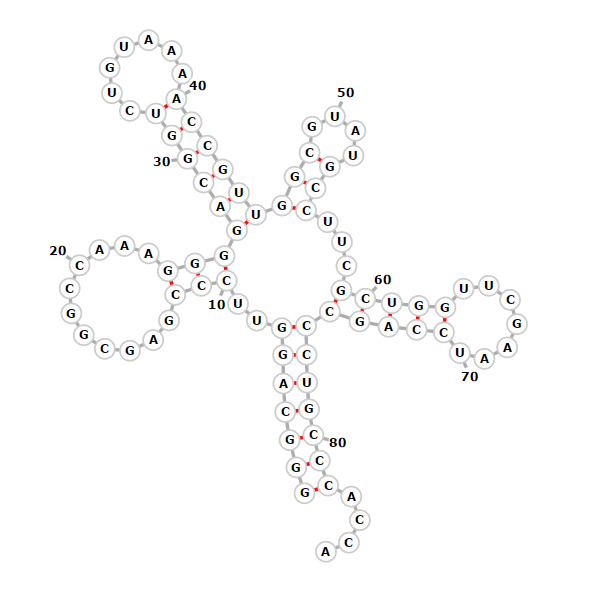
Рис. 3. Результат работы программы RNAfold: предсказание вторичной структуры тирозил-тРНК с помощью алгоритма Зукера.
Участок структуры |
Позиции в структуре (по результатам find_pair) |
Результаты предсказания |
Результаты предсказания по алгоритму Зукера |
Акцепторный стебель |
5'-1-7-3' 5'-66-72-3' Всего 7 пар |
0 пар из 7 реальных | 0 из 7 (не совпадает нумерация) |
D-стебель |
5'-10-12-3' 5'-23-25-3' Всего 3 пары |
не совпадает нумерация | все пары |
T-стебель |
5'-49-52-3' 5'-62-65-3' Всего 4 пары |
0 совпадений | 0 пар (не совпадает нумерация) |
Антикодоновый стебель |
5'-27-31-3' 5'-39-43-3' Всего 5 пар |
0 совпадений | 0 пар (не совпадает нумерация) |
Общее число канонических пар нуклеотидов |
22 | 27 | 24 |
Задание 2. Поиск ДНК-белковых контактов в заданной структуре.
В данном задании требовалось найти контакты между ДНК и белком в заданной структуре. Я исследовала комплекс инициаторного белка связывания в Trychomonas vaginalis с ферредоксиновым инициаторным сайтом ДНК (файл в формате .pdb ).
Упражнение 1.
Целью выполнения данного упражнения было вспомнить, как задаются различные группы атомов в JMol. Так как нумерация атомов в старом и новом форматах файла .pdb отличаются, я при выполнении всего задания использую файл в старом формате, полученный при выполнении прошлого практикума с помощью программы remediator.
Для поиска ДНК-белковых контактов мне потребовалось определить различные группы атомов с помощью команды define.
Я написала скрипт Sets(с которым можно ознакомиться, нажав на ссылку Text of the script 1), который позволяет на приведенном ниже Java-апплете последовательно увидеть изображения:
- ДНК-белкового комплекса в ленточной модели, покрашенного в соответствии с вторичной структурой
- только ДНК в стержневой модели, покрашенной cpk (по цветам атомов)
- ДНК в стержневой модели и набора атомов set1 (множество атомов кислорода 2'-дезоксирибозы), показанного шариками красного цвета
- Команда:
define set1 (*:*.O?')
- Команда:
- ДНК в стержневой модели и набора атомов set2 (множество атомов кислорода в остатках фосфорной кислоты), показанного шариками желтого цвета
- Команда:
define set2 (*:*.O?P and dna)
- Команда:
- ДНК в стержневой модели и набора атомов set3 (множество атомов азота в азотистых основаниях), показанного шариками синего цвета
- Команда:
define set3 (*:*.N? and dna)
После того, как структура загрузится, надо нажать две кнопки: 1. Запустить скрипт:
2. Продолжить исполнение скрипта:
Упражнение 2.
В данном упражнении было нужно описать ДНК-белковые контакты в заданной структуре и сравнить количество контактов разной природы. Это было сделано в программе JMol с помощью скрипта Contacts, который последовательно отображает необходимые (полярные или неполярные) атомы белка, находящиеся на требуемом расстоянии от определенного набора атомов, и сам набор атомов. Для удобства использовались отдельно наборы полярных и неполярных атомов, так как они образуют разные типы контактов (подробнее см. ниже).
Для выполнения упражнения определим понятия полярных и неполярных атомов и контактов:
- Полярные атомы: "N", "O"
- Неполярные атомы: "S", "P", "C"
- Полярный контакт - пара полярных атомов белка и ДНК, расстояние между которыми меньше 3.5Å
- Неполярный контакт - пара неполярных атомов на расстоянии меньше 4.5Å
Под контактом подразумевается потенциальная связь требуемой длины. Один атом способен образовывать более одного контакта соответствующего типа.
При выполнении упражнения использовались группы атомов, определенные при выполнении предыдущего упражнения (set1-set2 - группы полярных атомов), а также наборы:
- set4 (множество атомов углерода 2'-дезоксирибозы), показанное шариками белого цвета (неполярная группа)
- Команда:
define set4 (*:*.C?')
- Команда:
- set5 (множество атомов фосфора остатков фосфорной кислоты), показанное шариками белого цвета (неполярная группа)
- Команда:
define set5 (*:*.P and dna)
- Команда:
- set6 (множество полярных атомов азотистых оснований, обращенных в большую бороздку), показанное шариками красного цвета (полярная группа)
- set7 (множество неполярных атомов азотистых оснований, обращенных в большую бороздку), показанное шариками белого цвета (неполярная группа)
- set8 (множество полярных атомов азотистых оснований, обращенных в малую бороздку), показанное шариками красного цвета (полярная группа)
- set9 (множество неполярных атомов азотистых оснований, обращенных в малую бороздку), показанное шариками белого цвета (неполярная группа)
При определении множеств атомов азотистых оснований, обращенных в большую и малую бороздки, я пользовалась данными об остатках, полученными мной и моими однокурсниками при выполнении задания 3 прошлого практикума.
Результатом выполнения задания является Таблица 2, а также можно пронаблюдать интересующие контакты в JMol-апплете.
Таблица 2. Контакты разного типа в комплексе 1PP7_old.pdb.
Контакты атомов белка с
Полярные
Неполярные
Всего
остатками 2'-дезоксирибозы
5 41 46 остатками фосфорной кислоты
9 14 23 остатками азотистых оснований со стороны большой бороздки
3 10 13 остатками азотистых оснований со стороны малой бороздки
4 1 5
При выполнении этого задания были получены довольно интересные данные. Как нетрудно заметить, посмотрев на данные в Таблице 2 или на сами структуры, белок довольно мало взаимодействует с ДНК. Даже визуально они находятся не слишком близко, и только некоторые участки белка подходят ближе к цепи ДНК. В этих нескольких небольших участках и сконцентрированы все атомы, потенциально образующие контакты. Таким образом, можно предположить, что ДНК имеет довольно короткие сайты связывания с конкретным белком.
Во взаимодействии представлено значительно больше неполярных контактов (в 5 раз больше). Также намного больше контактов с сахарофосфатным остовом, это типично для "заякоривания" ДНК-связывающих белков на ДНК. Однако функциональное связывание белка должно происходить с азотистыми основаниями (вообще типично, что белок должен узнавать некий паттерн ДНК). В моем случае можно сказать, что белок связывается с последовательностью в большой бороздке.
Что интересно, сначала я посчитала количество контактов на глаз, а затем решила посчитать более точно и измеряла расстояния между атомами. Во втором случае получилось гораздо меньше контактов.
Упражнение 3.
В данном упражнении требовалось получить схему ДНК-белковых контактов с помощью программы nucplot. Для ДНК-белковой структуры я получила файл nucplot.ps с данными о популярных контактах, затем я конвертировала его в формат .jpg. С результатом визуализации работы программы nucplot можно ознакомиться на Рис 4a-b.
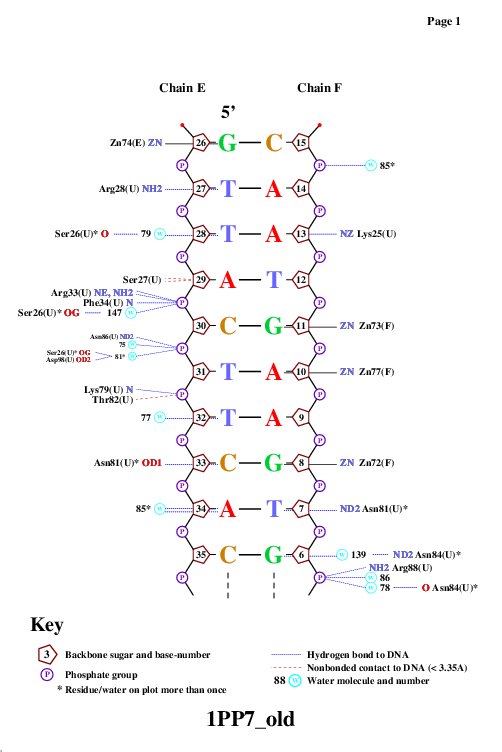
Рис. 4a. Схема ДНК-белковых взаимодействий инициаторного белка связывания в Trychomonas vaginalis с ферредоксиновым инициаторным сайтом; часть 1.
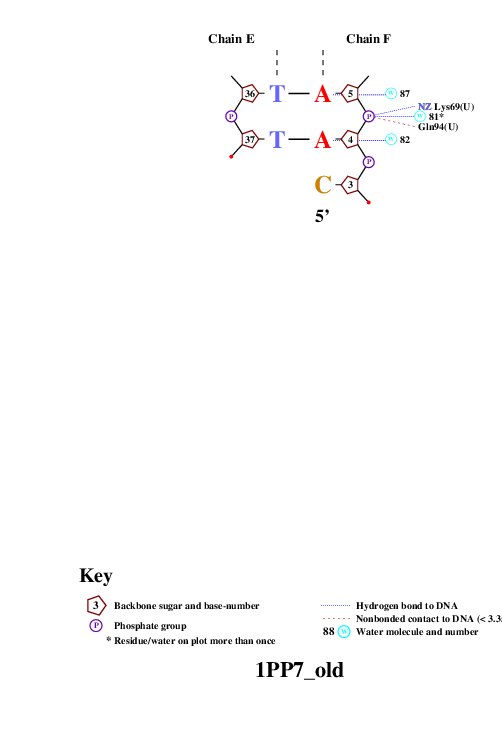
Рис. 4b. Схема ДНК-белковых взаимодействий инициаторного белка связывания в Trychomonas vaginalis с ферредоксиновым инициаторным сайтом; часть 2.
Упражнение 4.
Для выполнения этого упражнения я выбрала на полученной схеме два остатка:
- аминокислотный остаток с наибольшим числом указанных на схеме контактов с ДНК (Рис. 5а). Мне было трудно сделать выбор, так как большинство остатков связаны с ДНК через молекулы воды. Единственный остаток, контактирующий с ДНК без посредства воды более, чем 1 раз, это остаток 81 аспарагина (цепь U). Он связан с 7 тимином и 33 цитозином. Однако согласно схеме, данные контакты даже не являются связями, просто расстояние между атомами менее 3.35Å.
- аминокислотный остаток, наиболее важный для распознавания последовательности ДНК (Рис. b). С выбором данного остатка было еще сложнее. В схеме нет какого-либо остатка,
который был бы связан с ДНК большим количеством связей. К тому же, многие контакты идут через посредничество воды. Предположительно, это может быть вышеупомянутый остаток
81 аспарагина.
Я решила разобраться в этом и прочитала статью людей, которые тоже хотели в этом разобраться. Оказалось, что влияние многих остатков крайне неочевидно, и потребовались эксперименты для проверки гипотез.
Следует сказать, что рассматривается взаимодействие между инициаторным белком IBP39 (Inr binding protein 39kDa), который связывается с инициаторным участком Inr. У большинства эукариот решающий вклад в инициацию транскрипции вносит связывание инициаторного белка с последовательностью ТАТА-box, а связь с Inr и нижележащим элементом (downstream element) носят некий регуляторный характер. Но у данного организма (Trichomonas vaginalis), являющегося одним из наиболее примитивных представителей эукариот, наибольшую значимость имеет именно связывание инициаторного белка с Inr. Вообще, белок IBP имеет N-конец, связывающийся с ДНК (как раз и представлен на структуре), далее некую связку и С-конец, который связывается с РНК-полимеразой II.
В рассматриваемом нами N-конце большую роль при связывании играют, во-первых, связи с фосфатами, которых большое количество, так как они наиболее соответствующим образом располагают белок относительно инициаторного сайта, коим является последовательность ACTTC. Область белка, связывающаяся с этим сайтом, называется α6, и именно ей принадлежит мой 81 аспарагин, так что моя гипотеза относительно оправдалась. Однако с помощью метода флюоресцентной поляризации было показано, что без связывания белка (петля 310) с остатком тимина 27 (цепь Е) и аденина 13 (цепь F), расположенными выше относительно инициаторного сайта (upstream the Inr core region) дальнейшее связывание с инициаторным сайтом не происходит. Поэтому на Рис. 5b я изобразила взаимодействие 28 аргинина и 25 лизина с вышеупомянутыми основаниями.
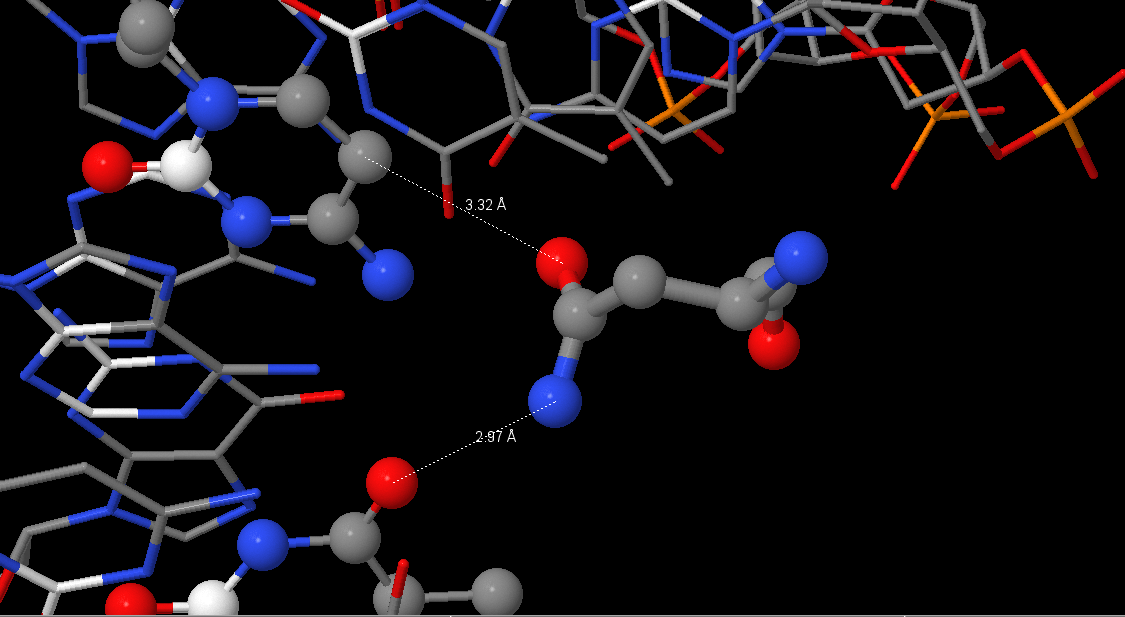
Рис. 5a. Изображение аминокислотного остатка ASN81 (цепь U) и двух его контактов с 7 тимином и 33 цитозином ДНК (не являются водородными связями).
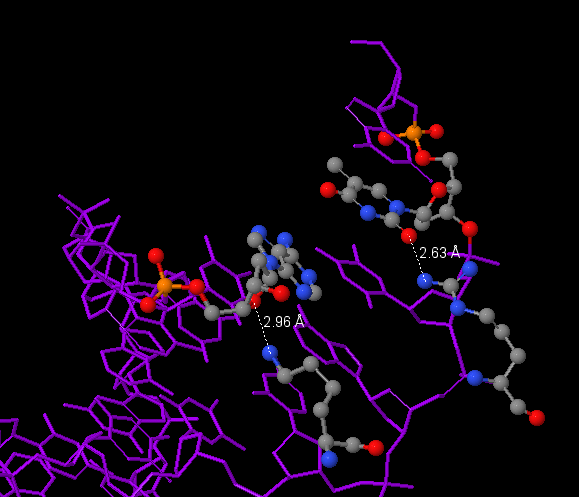
Рис. 5b. Изображение аминокислотных остатков ARG28 и LYS25 (U) с T27 (E) и A13 (F) ДНК соответственно; между лизином и аденином водородная связь, между тимином и аргинином - контакт.
- Команда: