Банки нуклеотидных последовательностей
Задание 1. Качество сборки генома эукариотического организма.
Все любят котиков. А я... нет. Поэтому для выполнения данного задания, в котором нужно охарактеризовать качество сборки генома эукариотического организма, я выбрала того представлителя эукариот, которого больше всего люблю... употреблять в пищу: это курица (Рис. 1).

На странице базы данных Genomes сайта NCBI я прошла по ссылке Browse by organism, после чего получила таблицу со списком организмов. С помощью фильтров я оставила для рассмотрения эукариот, затем животных и наконец птиц, среди которых и был организм Gallus gallus. Курица (а также еще 3 птичьих организма из 65 представленных в таблице) имеет наиболее полную сборку генома среди имеющихся на данный момент у птиц (закрашены 3/4 кружочка в колонке Levels, что означает сборку на хромосомном уровне).
Такое внимание к куриному геному не случайно: помимо того, что курица является важнейшим сельскохозяйственным животным, она еще и представляет собой одну из первичных моделей для эмбриологии и биологии развития (Рис. 2), а также изучения вирусов и рака (информация почерпнута из обзора организма [Organism Overview]). [1]
Число сборок генома.
Более подробную информацию о сборках генома можно получить, пройдя по соответствующей ссылке колонки Assemblies. На данный момент имеется одна сборка генома, принадлежащая самке одомашненной формы банкивской джунглевой курицы (инбредная линия UCD001), распространенной исходно в Юго-Восточной Азии (Рис. 3).

Число проектов и образцов.
Можно найти 3 проекта по секвенированию организма (BioProjects) (PRJNA10808, PRJNA13342, PRJNA10807), и всего 1 образец SAMN02981218 (BioSample). Из исходной таблицы (Рис. 4) ведет ссылка только на проект PRJNA10808 (референсный геном), но среди связанных с ним проектов указаны сборка митохондриального генома (PRJNA10807) и сборка 12Х 454 (с 12-кратным покрытием на 454 платформе - PRJNA13342).
Описание образца.
На Рис. 5 представлено описание образца SAMN02981218 (BioSample) с сайта NCBI (ссылка).

В описании указано следующее:
- Идентификаторы образца: SAMN02981218 (в базе данных NCBI Genomes); SRA: SRS926532 (архив ридов на сайте NCBI); GenBank: gb|AADN00000000.3 (в базе данных GenBank)
- Систематическое положение вида: является типовым видом в роде джунглевых кур (Gallus), принадлежит семейству фазановых (Phasianidae) отряда курообразных (Galliformes). Более подробная номенклатура представлена на Рис. 5 (не привожу перевод ввиду общеупотребительности номенклатуры на латыни)
- Уже ранее упомянутые мной характеристики организма:
Пол - самка
Порода - банкивская джунглевая курица, инбредная линия UCD001
Изолят (субпопуляция) #256 - Cсылки на проекты по секвенированию организма (и на образцы для каждого из проектов)
- Подача результатов (по сути источник и дата) - The Genome Institute, Washington University School of Medicine; 2014-08-11
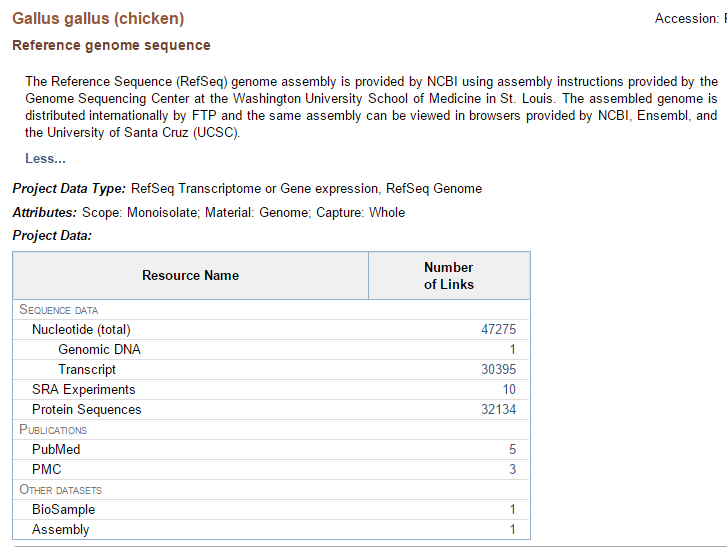
Описание проекта (PRJNA10808)
Данный проект представляет собой сборку референсной последовательности генома Gallus gallus, осуществленную NCBI в соответствии с инструкциями Genome Sequencing Center at the Washington University School of Medicine in St. Louis. Собранный геном был распространен в Сети с помощью сетевого протокола (FTP - File Transfer Protocol), и его можно посмотреть в браузерах, предоставленных NCBI, Ensembl, и University of Santa Cruz (UCSC).
Описание и информация о проекте представлены на Рис. 6.
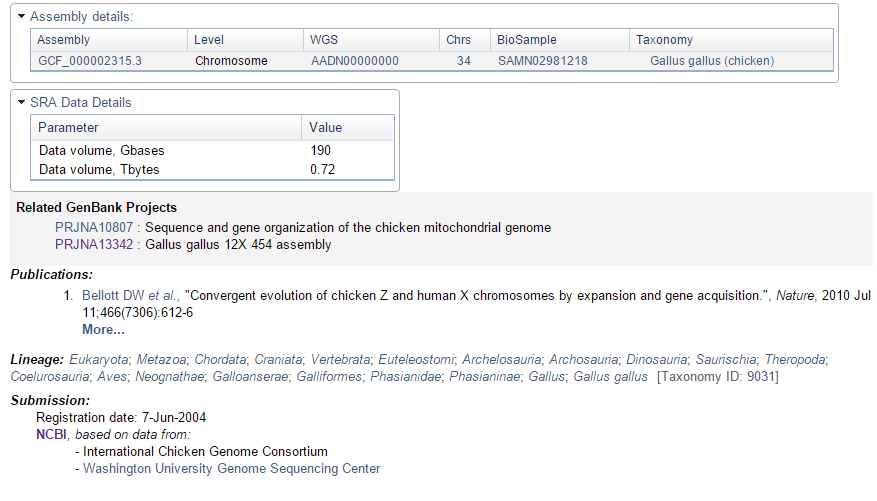
На Рис. 7 содержится дополнительная информация о сборке, проектах SRA (архив ридов), подаче проекта (Submission), а также систематическое положение и ссылки на публикации и другие проекты.
Данные о проекте также отражены в Таблице 1. Разобраться в параметрах помог словарь (BioProject Glossary). [2]
Таблица 1. Описание проекта PRJNA10808.
| Тип данных проекта | Референсный транскриптом, референсный геном |
| Параметры исследования: охват и чистота образца (Scope) | Отдельное животное (Monoisolate) |
| Параметры исследования: материал | Геном |
| Параметры исследования: тип полученной информации | Полный геном (Whole) |
| Данные о последовательности | Количество нуклеотидов, геномных ДНК, транскриптов, белковых последовательностей, групп ридов |
| Количество публикаций | PubMed и PMC |
| Другие группы данных | Образец и сборка |
| Количество публикаций | PubMed и PMC |
| Параметры сборки: сборка, уровень, номер хромосомы, проект полногеномного секвенирования (WGS) | GCF_000002315.3, хромосомный уровень, 34, AADN00000000 |
| Параметры ридов | Объем в гигабазах (Gb) и Тбит |
| Дата регистрации | 7/06/2004 |
| Источники данных (на чем основаны) | Международный консорциум по геному курицы и Washington University Genome Sequencing Center |
Контиги & скэффолды.
Далее требовалось проанализировать информацию, касающуюся контигов и скэффолдов.
- Контиг - полностью секвенированный фрагмент ДНК.
- Скэффолд - набор контигов, которые идут подряд в ДНК (между контигами может быть некоторое количество букв N, или достоверность этих учатсков ниже, чем в контигах).
Если пройти по ссылке WGS Project, то можно получить информацию о скэффолдах (их 4 - строки WGS_SCAFLD) и контигах (пройти далее по ссылкеWGS): их 27102.
Ссылка на таблицу с контигами (файл в формате .txt с сайта): таблица.
Для контигов указаны следующие параметры:
- N50 = 279,750 (означает, что не менее 50% генома покрыто контигами длиной не меньше, чем 279,750 п.н.; чем выше значение, тем лучше качество сборки)
- L50 = 950 (число контигов с длиной, удовлетворяющей параметру N50, то есть покрывающей не менее 50% генома)
- Самый длинный контиг: ctg7180000800512 (длина 1,808,339 нуклеотидов; accession: AADN03004003)
- Самый короткий контиг: ctg7180000832391 (длина 205 нуклеотидов; accession: AADN03004794)
Ссылка на последовательность контига ctg7180000706126 (длина 77,557; accession: AADN03000001): fasta.
Задание 2. Митохондриальные гены мха.
В данном задании требовалось построить таблицу митохондриальных генов мха. В моем случае, мха Racomitrium elongatum (Рис. 8).

Далее в базе данных Nucleotide (NCBI) я нашла митохондриальный геном моего мха. Для этого я использовала запрос (Racomitrium elongatum[ORGN]) AND mitochondrion[FILT] (того же результата можно достичь, если просто ввести запрос Racomitrium elongatum mitochondrion). Я получила 2 результата, которые отличаются датой и тем, что первая - референсная последовательность с NCBI, а вторая из GenBank. В дальнейшем буду работать с первой (она не намного свежее, но насколько я помню, в GenBank хуже определены концы последовательностей; судя по тому, что авторы одни и те же, сначала результаты поступают в GenBank, а потом проверяются и поступают на сайт NCBI).
Пройдя по первому результату, я затем прошла по ссылке Genome в разделе Related information. Там представлена таблица с количеством генов в митохондриальной ДНК (Рис. 9).

Итак:
- Общее число генов: 66
- Число генов белков: 39
- Число генов рибосомальных РНК: 3
- Число генов транспортных РНК: 24
Затем прошла по ссылке Gene в разделе Related information, где представлена таблица с иформацией обо всех генах. Ее нужно было сохранить так, чтобы гены располагались в порядке следования по цепи ДНК. Для этого я воспользовалась опцией Sort by Chromosome.
Ссылка на файл со всеми генами: genes.
Задание 3. Ключи для таблицы особенностей (Feature Table).
Для выполнения данного задания я воспользовалась документом, посвященным таблице особенностей, с сайта INSDC. [3]
Примеры для каждого ключа было удобно искать на сайте NCBI в базе данных Nucleotides по полю [Feature Key] (можно [FKEY]).
Результатом выполнения задания является Таблица 2.
Таблица 2. 10 ключей для таблицы особенностей.
| Ключ | Описание | Пример |
| terminator | Последовательность ДНК, вызывающая терминацию транскрипции РНК полимеразой | 723..746
/gene="sod" |
| sig_peptide | Последовательность, кодирующая сигнальный белок; последовательность, кодирующая N-концевой домен
подлежащего секреции белка; этот домен участвует в прикреплении растущей полипептидной цепи к мембране |
1..54
/gene="TCR1A" |
| V-region | Вариабельный участок легкой и тяжелой цепей иммуноглобулинов,
а также альфа, бета, гамма цепей рецепторов Т-клеток; может быть составлен из V-, D-, J-сегментов и N-участков |
1..277
/gene="VFM1" /product="immunoglobulin heavy chain variable region" |
| polyA_site | Сайт на РНК-транскрипте, к которому будут присоединены остатки аденина в ходе посттранскрипционного полиаденилирования | 863
/gene="crasp" |
| rep_origin | Ориджин репликации; сайт старта дупликации нуклеиновой кислоты с целью получения двух идентичных копий | 6
/direction=LEFT /note="ori" |
| repeat_region | Участок генома, содержащий повторяющиеся элементы | 1..206
/rpt_type=tandem /satellite="microsatellite:Gals032" |
| 5'UTR | 1)Участок 5'-конца зрелого транскрипта (предшествующий инициаторному кодону), который не транслируется в белок
2)Аналогично для генома РНК-вирусов |
1..63
/gene="crasp" |
| mat_peptide | Зрелый пептид или зрелая последовательность, кодирующая его; последовательность, кодирующая зрелый, или финальный, пептид
или белковый продукт, подвергающийся посттрансляционным модификациям; не включает стоп-кодон, в отличие от соответствующего CDS |
2764..8460
/gene="orf1ab" /locus_tag="BCoVgp01" /product="coronavirus nsp1 (PL1-PRO, PL2-PRO, HD)" /note="Contains papain-like proteinase 1 domain (PL1-PRO), X-domain, papain-like proteinase 2 domain (PL2-PRO) and hydrophobic domain (HD)." /protein_id="NP_742169.1" /db_xref="GI:26008083" |
| misc_feature | Участок, который не может быть описан никаким другим ключом; новое или редкое свойство | 21445..21450
/note="transcription regulatory sequence mRNA2" |
| mobile_element | Участок генома, содержащий мобильные (подвижные) генетические элементы | complement(2433133..2433282)
/locus_tag="An08e10250" /note="Title: probable transposon 160bp LTR (Tndm1-LTR) -Aspergillus niger" /mobile_element_type="transposon:Tndm1" |
Часть II
Задание 4. Какому гену принадлежит последовательность?
В данном задании требовалось установить, какому гену принадлежит последовательность, полученная в практикуме 6 (последовательность), и таксономию организма.
Для этого я использовала ресурс BLASTN, который осуществляет поиск последовательностей ДНК по базе nr, получив на входе последовательность ДНК.
Запуск я совершала с сайта BLAST, алгоритм - Somewhat similar sequences (blastn), все параметры оставила по умолчанию.
На Рис. 10 можно видеть часть результата работы программы: отображено 10 достоверных находок (E-value = 0.0, покрытие входной последовательности свыше 90%, процент идентичности свыше 80). Для последующей работы я отобрала 3 лучшие находки из достоверных, у которых процент идентичности превышает 90 (на Рис. 10 выделены черной рамкой).
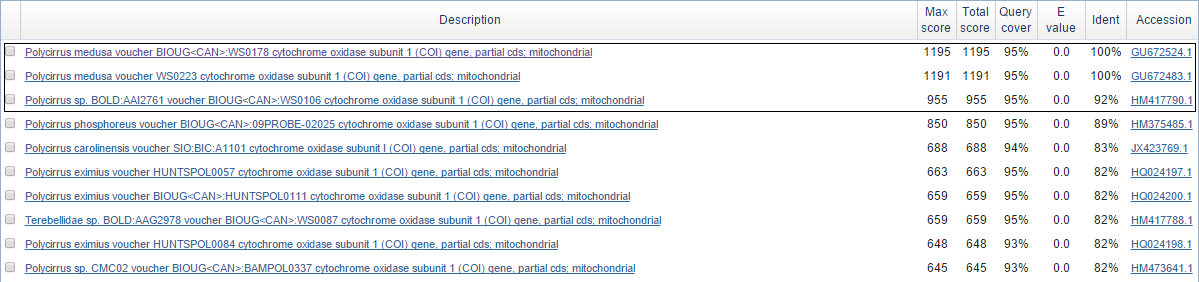
Как видно из Рис. 10, две первые находки принадлежат к роду Polycirrus medusa, а третья - Polycirrus sp. BOLD:AAI2761. Таким образом, все эти находки представляют собой виды рода Polycirrus - представителя многощетинковых кольчатых червей (Рис. 11а). На Рис. 11b можно ознакомиться с таксономическим положением данного рода (легко получить, пройдя от находки на страницу в GenBank, а затем на ссылку с названием вида).

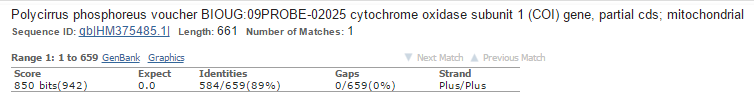
Все три находки являются последовательностями митохондриального гена, кодирующего субъединицу 1 цитохромоксидазы. В качестве подтверждения - Рис. 12 со страницей в GenBank лучшей находки - Polycirrus medusa (в Таблице 3 представлены ссылки на страницы GenBank для каждой из 3 находок).

В Таблице 3 для каждой находки представлены: вид организма, ссылка на страницу GenBank, количество и процент совпадений, выравнивание с исходной последовательностью (так как сходство очень велико, то было достаточно вручную сдвинуть последовательность-находку на 31 нуклеотид исходной последовательности).
Таблица 3. Информация о находках.
| Вид | Ссылка на GenBank | Identities | Выравнивание | Выравнивание в fasta-формате |
| Polycirrus medusa (voucher BIOUG:WS0178) | 1 | 662/662(100%) | Рис. 13а | 1 |
| Polycirrus medusa (voucher WS0223) | 2 | 660/660(100%) | Рис. 13b | 2 |
| Polycirrus sp. BOLD:AAI2761 (voucher BIOUG:WS0106) | 3 | 609/662(92%) | Рис. 13c | 3 |



Также с выравниваниями можно ознакомиться в проекте JalView.
Как можно заметить, первые две находки полностью совпадают с исходной последовательностью (просто отсеквенированная последовательность немного длиннее последовательности гена), а между собой эти находки отличаются только длиной (2 нуклеотида). Третья находка принадлежит неопределенному виду рода Polycirrus, и она имеет уже 92% сходства.
Можно заключить:
- Последовательность, полученная в практикуме 6, - митохондриальный ген субъединицы 1 цитохромоксидазы.
- Ген из организма Polycirrus medusa (так как выравнивание с исходной последовательностью не содержит замен).
- Число замен на 100 п.н. в лучшей находке - 0
- Число замен на 100 п.н. в лучшей находке из гарантированно другого вида (4 находка, так как видовая принадлежность 3 находки не ясна) - 11 (см. Рис. 14)
Использованные ресурсы: