Предсказание генов прокариот
Задание 1. Аннотировать последовательность с помощью ресурса RAST и сравнить с аннотацией генов в записи GenBank.
В данном задании требовалось аннотировать прокариотическую последовательность (хромосому или плазмиду) или вирусный геном с использованием ресурса RAST, а затем сравнить с аннотацией генов в соответствующей записи GenBank.
Для выполнения данного задания я, как нетрудно догадаться, выбрала последовательность хромосомы моей бактерии из 1-го семестра: Aquifex aeolicus VF5. Также у этой бактерии есть плазмида (запись GenBank: AE000667).
Информация о бактерии и ее хромосоме представлена в Таблице 1.
Таблица 1. Информация о бактерии Aquifex aeolicus VF5 и ее хромосоме.
| Название генома | Aquifex aeolicus VF5 chromosome, complete genome |
| Taxonomy ID | 224324 |
| Таксономия | cellular organisms; Bacteria; Aquificae; Aquificae; Aquificales; Aquificaceae; Aquifex; Aquifex aeolicus |
| Тип хромосомы | кольцевая хромосома (полный геном) |
| Длина последовательности (п. н.) | 1551335 |
| GenBank AC (ссылка на запись) | AE000657.1 |
Я добавила последовательность хромосомы моей бактерии в формате fasta (последовательность), заполнила данные, касающиеся таксономии организма и запустила программу с параметрами, указанными по умолчанию.
Выдача программы содержит очень много полезной информации:
- файл с аннотацией генов в формате .gb (ссылка)
- файлы с последовательностями генов (нуклеотиды) и их продуктов (аминокислотные остатки)
- файл Excel с информацией о локализации гена в хромосоме и его продукте (ссылка)
- графическое отображение местоположения каждого из генов в хромосоме нашего организма и некоторых близких (пример: Рис. 1а)
- совершенно прекрасная диаграмма, демонстрирующая долю генов с определенной функцией (функциональные субсистемы генов) (Рис. 1b)
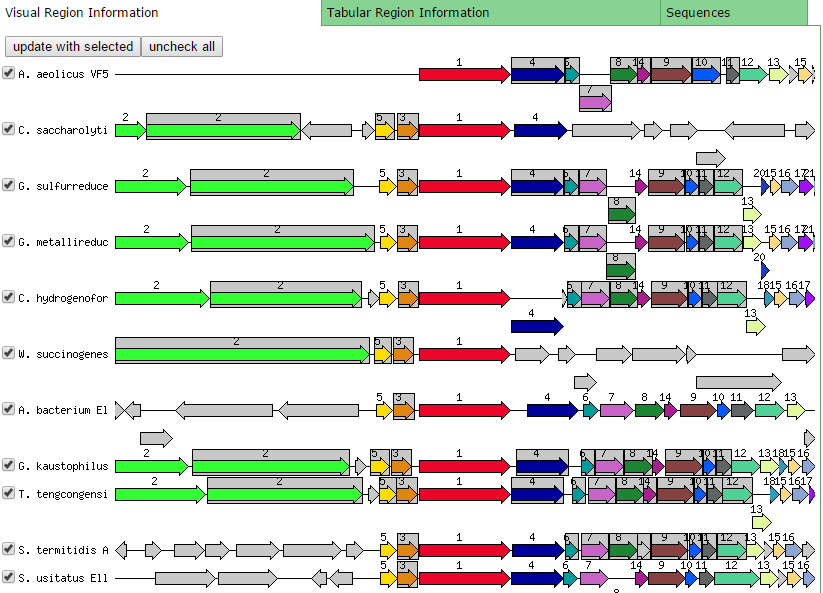
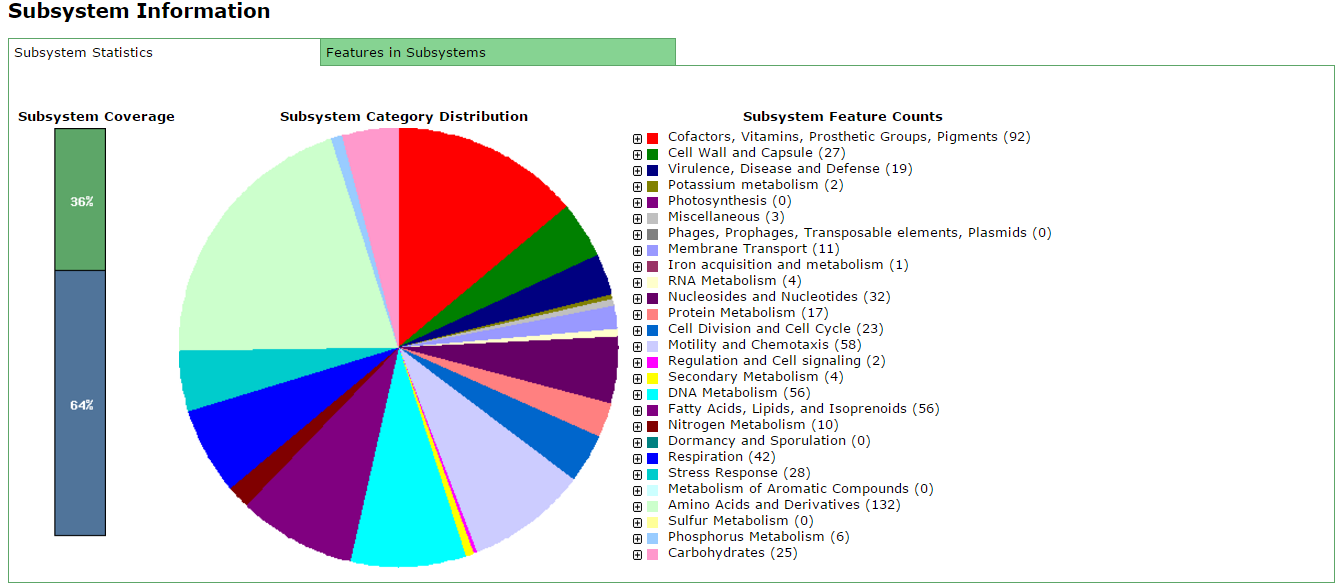
Чтобы сравнить аннотацию генов бактерии из записи GenBank и сделанную ресурсом RAST , я сделала сводную таблицу с информацией о генах. Для этого я использовала таблицу с информацией о генах белков из GenBank, полученную мной в практикуме 9 (таблица), а также обработала таблицу, которую выдал RAST (таблица).
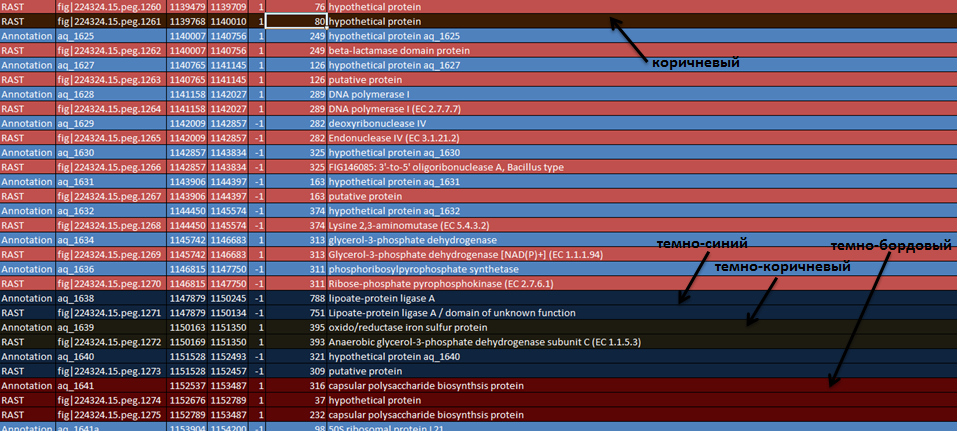
В сводной таблице (таблица) строчками разных цветов выделено следующее (для определенности цвета обозначены на Рис. 2)
- аннотации генов из записи GenBank (голубого цвета)
- аннотации генов с помощью RAST (вишневого/бордового цвета)
- гены, аннотированные GenBank, но не RAST (зеленого цвета)
- гены, аннотированные RAST, но не GenBank (коричневого цвета)
- гены, расположенные на прямой цепи, у которых не совпадают старт-кодоны, а стоп-кодоны совпадают (черного цвета)
- гены, расположенные на обратной цепи, у которых не совпадают старт-кодоны, а стоп-кодоны совпадают (темно-синего цвета)
- гены, у которых не совпадают ни старт-кодоны, ни стоп-кодоны (красного цвета)
- гены, у которых одной записи GenBank (возможно!) соответствуют 2 записи RAST (темно-бордового цвета)
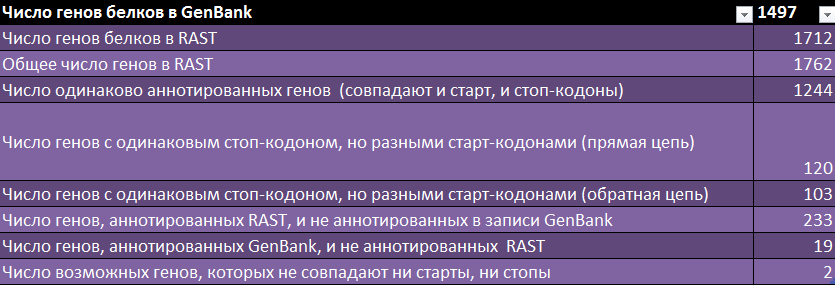
Результаты количественного сравнения вышеупомянутых групп генов приведены в файле Excel и в Таблице 1.
Таблица 1. Результаты количественного сравнения аннотаций.
Далее я выбрала 3 гена, у которых не совпадают аннотации (30S ribosomal protein S19, sugar fermentation stimulation protein, protoporphyrinogen oxidase) и аннотировала их с помощью blastp по базе данных Swiss-Prot, чтобы получить более подтвержденную информацию о белках (в этой БД содержатся последовательности только тех белков, существование которых было подтверждено хотя бы по гомологии).
К сожалению, при проверке разных аннотаций blastp в лучшем случае выдавал одну адекватную последовательность белка, которая была подтверждена по гомологии (то есть, по сути, не подтверждена совсем). Но пришлось работать с тем, что есть, поэтому я выбрала примеры, когда проверка аннотации приводит к разным результатам.
Также я хотела бы упомянуть, что геном моей бактерии (штамм) отсеквенирован (и белки аннотированы) только один раз, геномы других штаммов и видов рода не отсеквенированы, а находки близких родов слишком отличаются, чтобы помочь принять решение в пользу какой-либо из аннотаций.
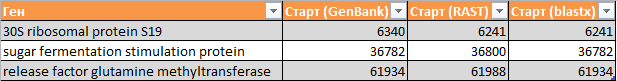
- У гена белка 30S ribosomal protein S19 в разных аннотациях не совпадают гены стартов (Рис. 3а).
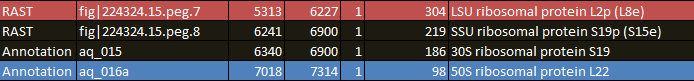
Рис. 3a. Фрагмент сводной таблицы (ген белка 30S ribosomal protein S19).
В выдаче blastp (Рис. 3b) красной рамкой выделена лучшая находка, которая является единственной относящейся к организму Aquifex aeolicus VF5. Выравнивание лучшей находки с входной последовательностью представлено на том же рисунке. Видно, что по длине она совпадает с аннотацией, предложенной RAST , и сами последовательности идентичны.
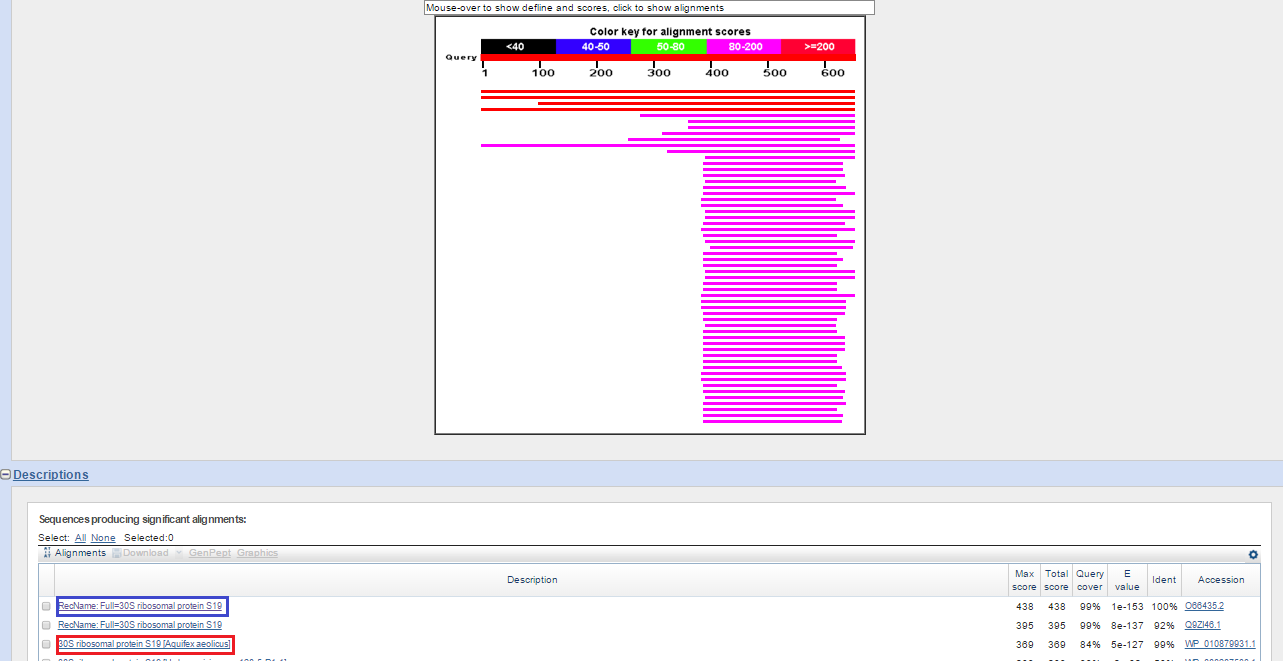
Рис. 3b. Выдача blastp для последовательности гена белка 30S ribosomal protein S19.
На Рис. 3с приведен скриншот фрагмента страницы записи данного белка в Swiss-Prot (ссылка на запись). Красной линией подчеркнута информация о том, что белок был аннотирован по гомологии. Следовательно, можно принять решение о выборе в качестве старта кодона, предложенного RAST.
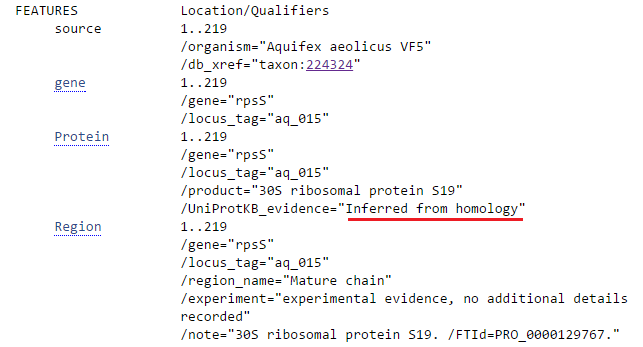
Рис. 3с. Фрагмент страницы записи в Swiss-Prot белка 30S ribosomal protein S19.
- На Рис. 4а представлен фрагмент сводной таблицы, иллюстрирующий, что у гена белка sugar fermentation stimulation protein в разных аннотациях также
не совпадают старты.

Рис. 4a. Фрагмент сводной таблицы (ген белка sugar fermentation stimulation protein).
Снова blastp выдал находку, идентичную входной последовательности на 100%, но на этот раз query совпадает с последовательностью из GenBank: одинаковы длина белка (214) и сами последовательности (выдача blastp и выравнивание на Рис. 4b). На Рис. 4с - подтверждение существования белка по гомологии (фрагмент записи Swiss-Prot, ссылка). Вывод: выбираем старт, предложенный GenBank.
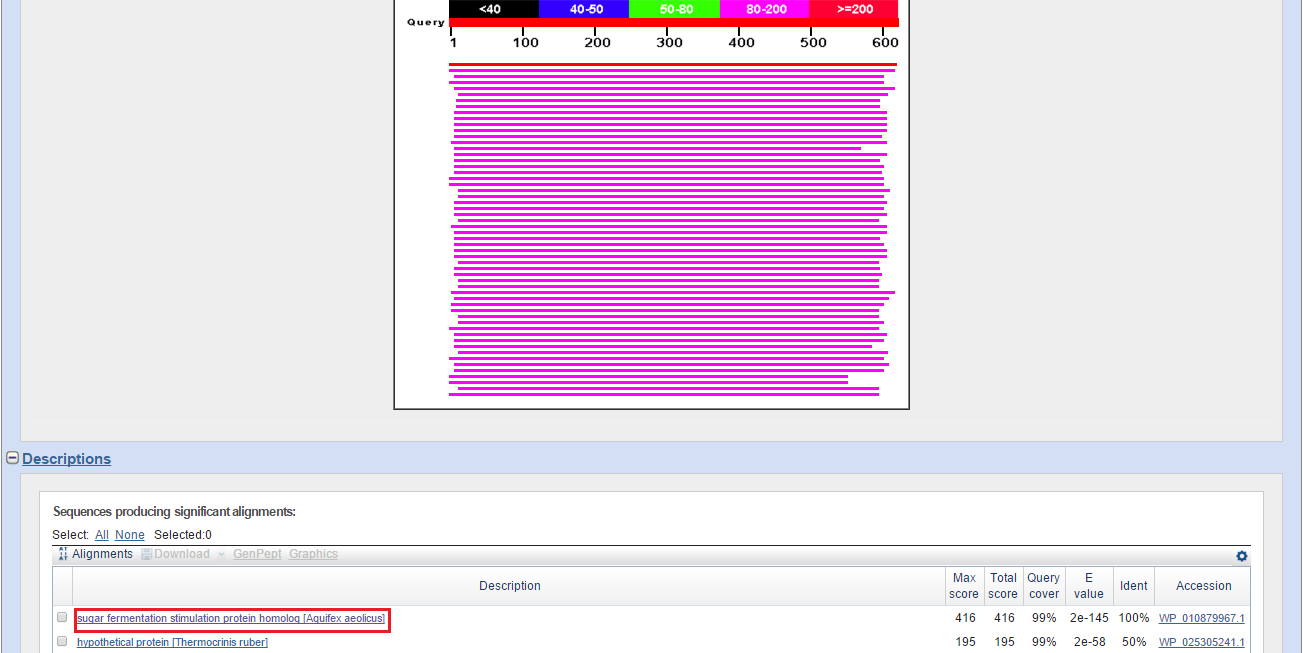
Рис. 4b. Выдача blastp для последовательности гена белка sugar fermentation stimulation protein.
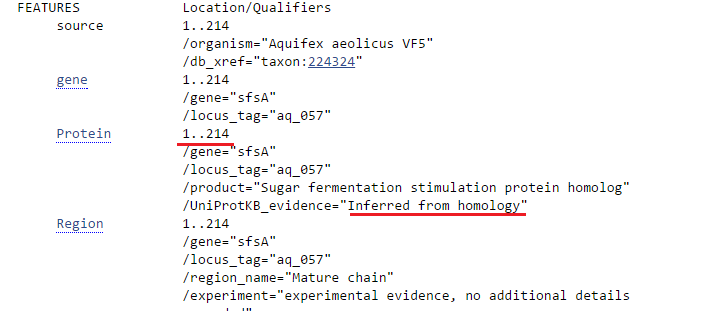
Рис. 4с. Фрагмент страницы записи в Swiss-Prot белка sugar fermentation stimulation protein.
- На десерт представлен интересный случай: у гена protoporphyrinogen oxidase в GenBank с сводной таблице (Рис. 5а) с аннотацией в RAST не совпадают не только старты,
но и названия продуктов.

Рис. 5a. Фрагмент сводной таблицы (ген белка protoporphyrinogen oxidase).
blastp выдал следующие результаты: последовательность query полностью по всей длине совпадает с последовательностью GenBank (Рис. 5b), но при этом название продукта у находки отличается, от предложенного GenBank: release factor glutamine methyltransferase.
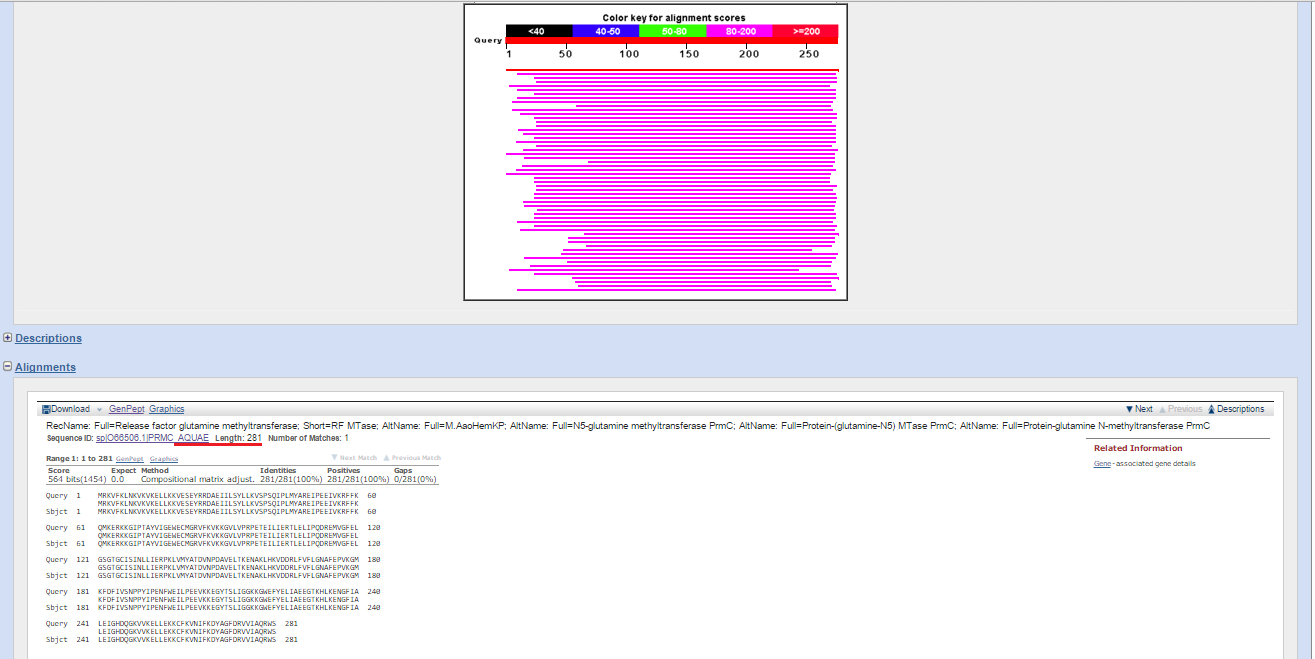
Рис. 5b. Выдача blastp для последовательности гена белка release factor glutamine methyltransferase.
Запись данного белка в Swiss-Prot (Рис. 5с) демонстрирует интереснейшую вещь: предсказанным продуктом одного из участков гена данного белка является белок, аннотированный RAST (по длине тоже почти совпадает). Это может послужить аргументом в пользу того, что название продукта в GenBank ошибочно (хотя проверить функцию можно только экспериментально), и что правильное название белка: release factor glutamine methyltransferase.
Также этот пример наталкивает на такую мысль: в большинстве примеров расхождения аннотаций последовательность, предлагаемая RAST , короче и соответствует функциональному участку или каталитической субъединице, поэтому, возможно, алгоритм работы RAST часто приводит к поиску не гена целиком, а наиболее "значимой" его части (в случае наличия еще и других фрагментов в гене и его продукте). Если такая часть способна существовать автономно in vivo, то это поднимает фундаментальный вопрос о том, что же на самом деле считать геном.
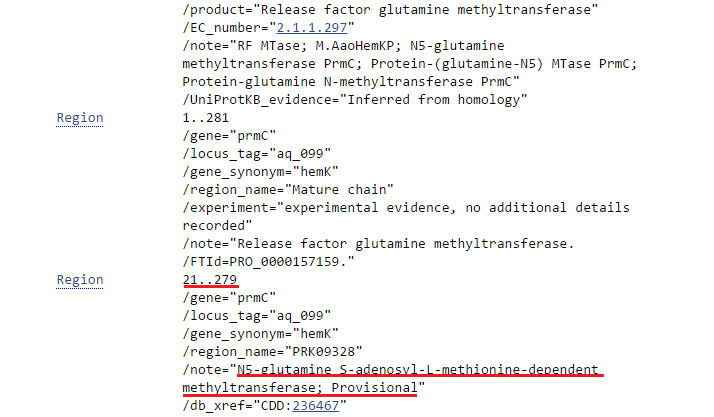
Рис. 5с. Фрагмент страницы записи в Swiss-Prot белка release factor glutamine methyltransferase.
В заключение хотелось бы сказать, что конкретно на моем примере аннотация генов с помощью RAST оказалась полезной, потому что:
- Так как большинство аннотаций генов совпадают, расхождения сигнализируют о спорных или (чаще) плохо изученных генах, на которые стоит обращать внимание при работе.
- Очень часто аннотации белка "hypothetical protein" в GenBank соответствует продукт с конкретной функцией в RAST , где, вероятно, менее строгое отношение к предсказанию функции. Думаю, полезно хотя бы предполагать функцию белка, хотя эта информация и не подтверждена.
- В ряде случаев RAST позволяет уточнить последовательность белка.
- Вышеупомянутые "расщепления" генов в GenBank на несколько записей в RAST не дают мне покоя. Думаю, что из этого тоже можно извлечь дополнительную информацию.