 Сборка de novo
Сборка de novo


За окном по-прежнему не идет снег. Красная икра в бутербродных количествах по-прежнему не по карману. А электрические гирлянды по-прежнему запрещены в общежитиях. Все это побуждает искать альтернативные источники новогоднего настроения. Но, к счастью, последний в этом году практикум дает возможность прикоснуться к настоящему чуду - таинству сборки генома de novo.
Пролог.
Собирать мы будем чтения экзома хромосомы 14 того же пациента, что и в практикуме 13, прошедшие сложнейшую процедуру очистки программой Trimmomatic (файл с очищенными чтениями).
Глава 1. "Мы наш, мы новый мир построим..".
Для начала мы притворились, что нет никаких результатов многолетней работы всемирного научного сообщества, то есть сборки генома человека не существует. Поэтому нужно было заново собрать контиги из чтений своего набора без использования референса.
Для выполнения этой непростой задачи нам был дан пакет velvet, установленный на kodomo (ссылка на руководство для желающих познакомиться поближе).
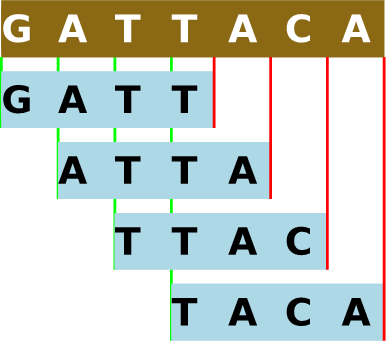
Хитроумная программа работает так: сначала она выделяет в каждом чтении так называемые k-меры - все возможные последовательности указанной длины (в нашем случае длины 31 - это максимально допустимая длина), так что их количество вычисляется, как длина чтения - длина k-мера + 1 (на случай недопонимания - на Рис. 2а показано выделение k-меров).
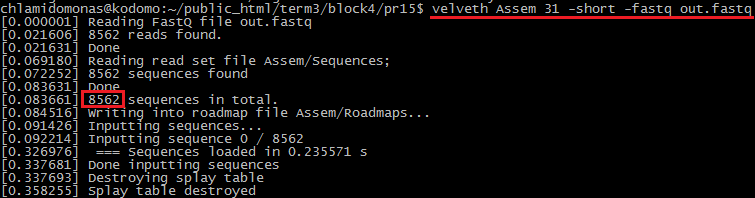
Эта операция приводится в действие командой velveth, для которой я указала директорию Assem, в которую она будет складывать результаты своей работы, длину k-мера (31), опции -short (короткие одноконцевые чтения - вообще-то она стоит по умолчанию) и -fastq (формат входного файла).
Но верить мне на слово не стоит, поэтому на Рис. 2b приведен запуск команды velveth и сообщение программы о проделанной работе.
Следующим этапом является сборка контигов на основе построения ориентированного графа de Bruijn (Брайна/Брюйна/Брейна/Брёйна
 ):
k-меры, имеющие очень консервативные участки перекрывания, соединяются стрелками графа (впоследствии объединяются в контиги). Полученный граф и есть вариант сборки
генома (на Рис. 2с расшифровка написанного).
):
k-меры, имеющие очень консервативные участки перекрывания, соединяются стрелками графа (впоследствии объединяются в контиги). Полученный граф и есть вариант сборки
генома (на Рис. 2с расшифровка написанного).
Строить Граф-Который-Нельзя-Называть и на его основе объединять k-меры в контиги умеет команда velvetg, которой я только указала директорию Assem (на Рис. 2d приведена команда).
Итоговый граф имеет 411 узлов (то есть собрано 411 контигов), что, на мой взгляд, довольно плохо, так как в самой новой сборке генома человека 1456 контигов, а этот результат получен всего лишь с хромосомы 14...
Также для полученной сборки параметр N50 = 232, это означает, что контигами длиной не менее 232 п.н. можно покрыть 50% генома. Чем больше значение N50, тем лучше (опять же обратимся к данным лучшей сборки человеческого генома, для нее N50 = 56413054, "почувствуйте разницу").
Еще один интересный показатель: длина наибольшего контига равна 736.
На сами последовательности контигов можно взглянуть в файле.
Глава 2. "Ах, обмануть меня не трудно, я сам обманываться рад".
После пребывания в недолгом, но приятном зачарованном сне наступило время пробуждения: пришлось вспомнить о том, что Северный Полюс до нас покорили уже не один раз, то есть уже существуют сборки генома человека. И поэтому возникает совершенно простая идея - проверить качество сборки генома программой velvet, выполнив поиск получившихся контигов по базе референсной хромосомы 14.
Благородная идея и осуществляется с помощью нехитрого приспособления: я использовала blastn (алгоритм megablast), который ищет высококонсервативные сходства входных последовательностей с базой данных.
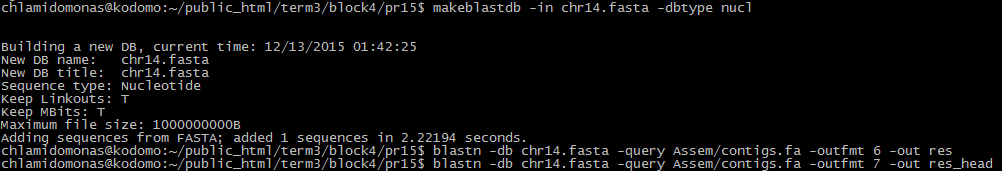
На Рис. 3 приведены команды, которые я использовала: сначала makeblastdb с опцией -dbtype nucl, создающей базу данных из нужной последовательности, а затем blastn (megablast по умолчанию), которому я заказала табличную выдачу (опция -outfmt 7) и указала базу данных (-db) и файл с входными последовательностями (-query).
Результат выдачи blastn я перенесла в таблицу Excel. И вот тогда, только тогда я наконец поняла, что волей провидения именно я в этот раз сорвала джекпот: общее количество мест картирования контигов на референсную хромосому составляет 26374. Остается только выдохнуть и анализировать :)
Итак, пойдем по порядку.
Для начала предлагаю ознакомиться с листом all файла Excel. Для каждого контига я подсчитала, сколько раз он откартировался на хромосому. В общем случае если контиг более одного раза встречается в хромосоме, то он соответствует некоторому повтору. Для удобства зрителей контиги, которые встречаются в хромосоме 1 раз, выделены в таблице вишневым цветом с белым текстом, а все повторы, относящиеся к одному контигу, выделены для каждого контига своим цветом (вот так вот непросто!).
Результаты ошеломляющие! Предлагаю Вашему вниманию топ-5 встретившихся наиболее часто в хромосоме контигов (Таблица 1).
Таблица 1. Контиги, встретившиеся чаще всего в хромосоме 14.
| # места | Номер контига | Длина контига* | Количество картирований на хромосому |
| I место | 103 | 243 | 7419 |
| II место | 360 | 152 | 7128 |
| III место | 107 | 699 | 4930 |
| IV место | 144 | 104 | 3747 |
| V место | 211 | 699 | 2715 |
* - длина, указанная в таблице Excel в названии контига после слова "length" - это количество k-меров, поэтому реальная длина контига +30
Ситуация крайне неожиданная. Во-первых, удивляет число - несмотря на то, что ДНК человека на 90% состоит из повторов, трудно представить, чтобы один повтор присутствовал в хромосоме в таких количествах (но, видимо, придется принять это, как данность). Во-вторых, наши контиги получены из экзома, а повторы, как правило, выбраковываются эволюцией, когда попадают в кодирующую область.
На волне трудового энтузиазма и подогреваясь научным интересом, я решила посмотреть, чему соответствует самый часто откартировавшийся на хромосому контиг 103.
Для этого я сначала запустила blastn (алгоритм megablast) для последовательности этого контига по базе Human genomic + transcript (там собраны последовательности генома человека и транскрипты), чтобы посмотреть, попадалась ли ученым людям эта последовательность. Результат работы программы, не изобилующий находками, можно видеть на Рис. 4а.
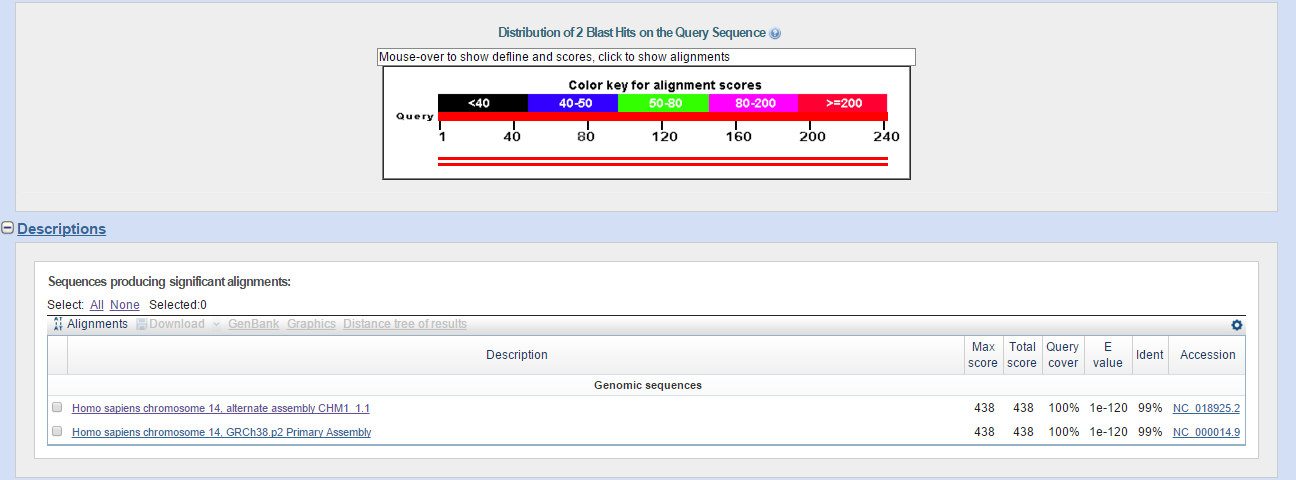
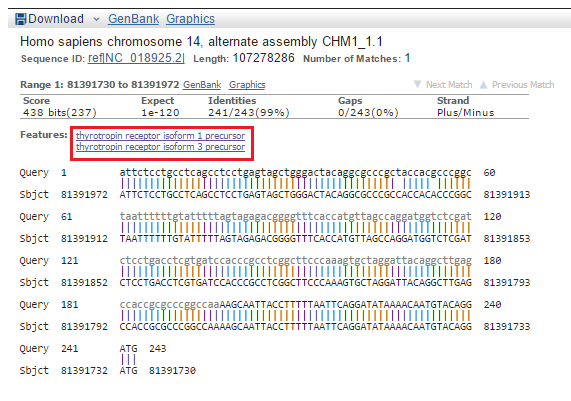
Находки соответствуют двум разным сборкам моей хромосомы 14. Что интересно, находки соответствуют участкам генов предшественников разных изоформ тиротропинового рецептора (красная рамка на Рис. 4b с выравниванием находки).
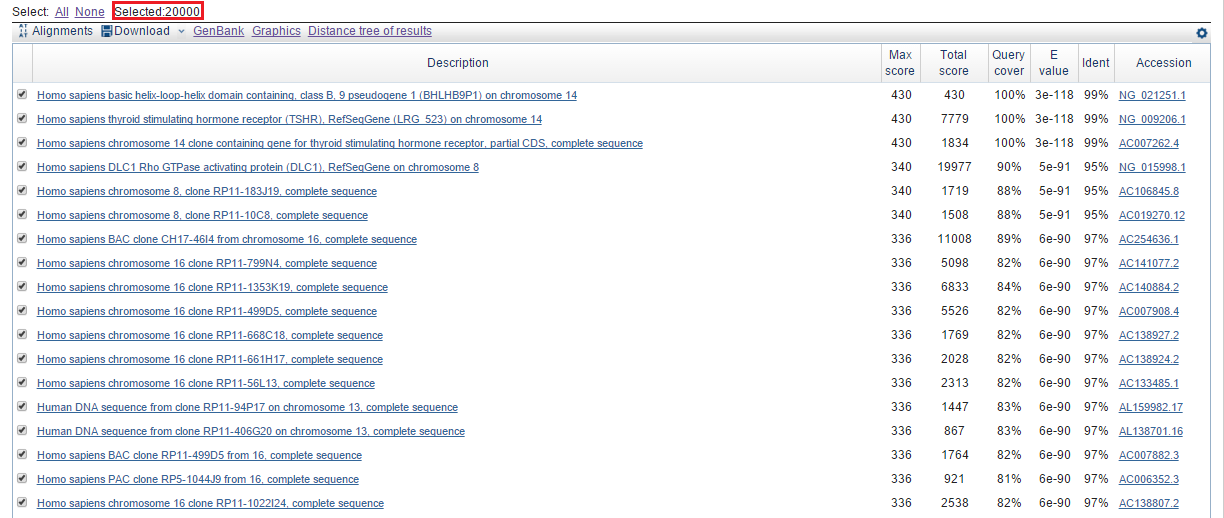
Затем я запустила просто blastn по базе non-redundant, указав организм human и увеличив число выдач до 20000, чтобы посмотреть, что вообще находится.
На Рис. 4с можно видеть результат: в выдаче 20000 находок (то есть, возможно, их гораздо больше), среди которых много находок на каждой хромосоме, и в некоторых этих находках присутствует несколько выравниваний (!) (причем все находки "хорошие" - с высоким Score и очень маленьким E-value).
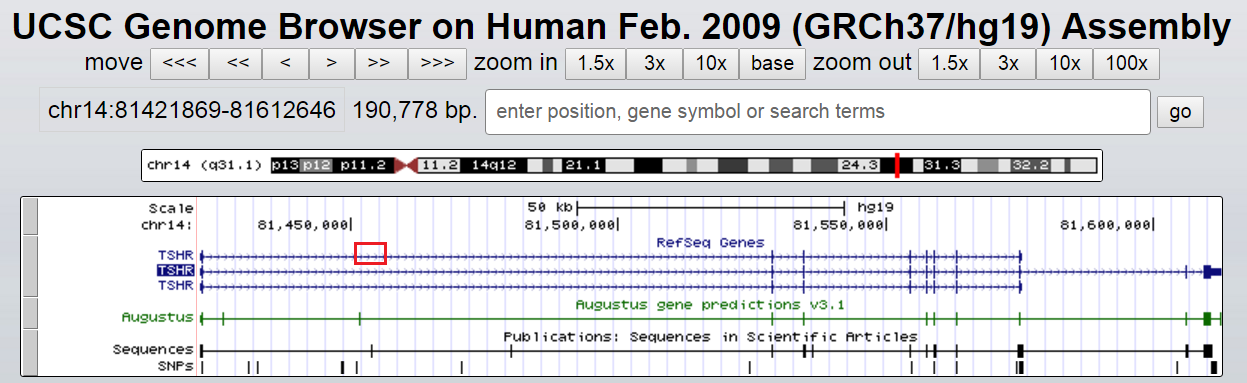
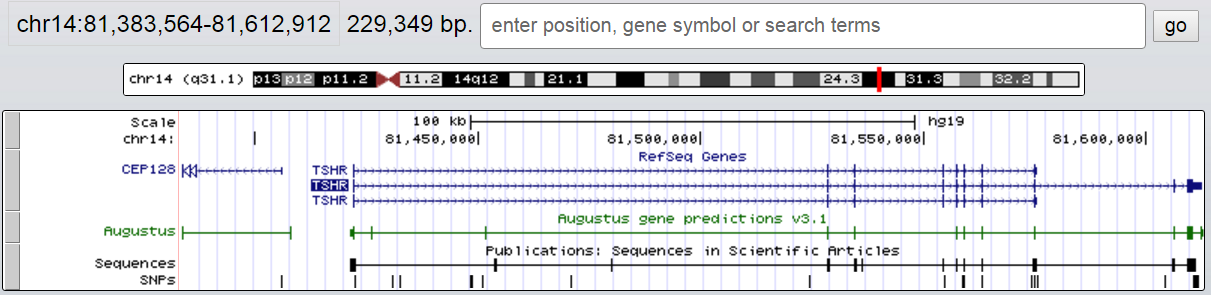
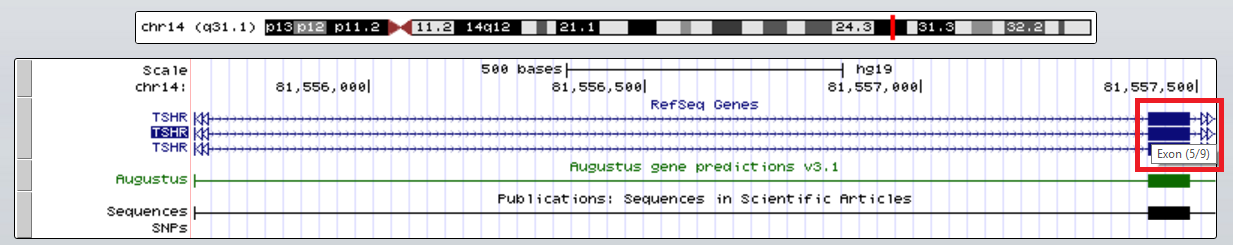
Судебное заключение будет таким: контиг 103 соответствует очень часто встречающемуся повтору (возможно, транспозону), который, однако, не изучался, но встречался благодаря попаданию в ген. Для дополнительной иллюстрации я решила привести изображения двух мест расположения повтора на хромосоме 14 в USCS Genome Browser: на Рис. 5а повтор попадает в интрон того самого гена рецептора тироид-стимулирующего гормона (81451507-81451265), а на Рис. 5b повтор вообще не попадает в ген (32761926-32761732).
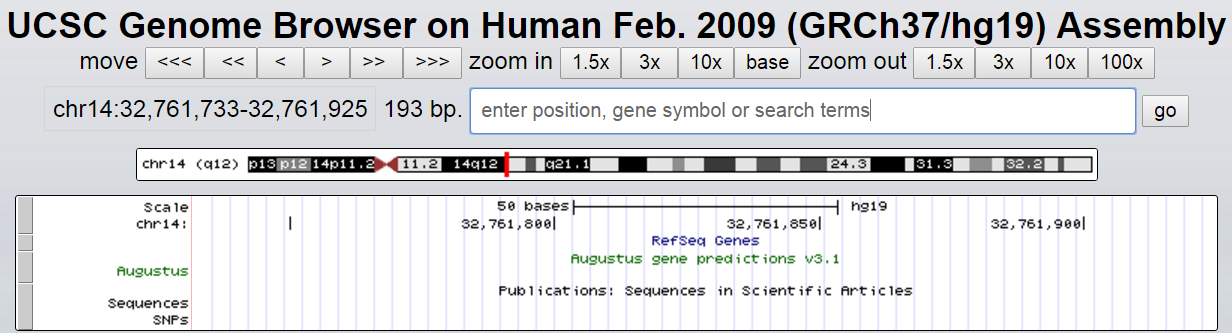
NOTE! Секвенировали мы (ну, положим, не мы) экзом, а все находки, в лучшем случае, из интрона. Это невольно наводит на мысль о том, что экзом выделяется не совсем аккуратно, что в сиквенс попал повтор из интрона, который, к слову, довольно близко расположен от ближайшего экзона. Либо повтор попадает в ген, но это не изучено.
Для тех, кто еще с нами, приключение продолжается. Устройтесь поудобнее в том, в чем сидите, и познакомьтесь с листом no-repeat файла Excel, в который я заботливо перенесла все контиги, которые картировались на референсную хромосому только один раз. Я отсортировала их по порядку следования в хромосоме и подсчитала величину перекрытия соседних генов (отрицательное значение в таблице означает разрыв между контигами). Ненулевые значения (страшно радует, что нулевые вообще есть) вызывают следующие вопросы: если есть перекрытия, то почему программа не объединила перекрывающиеся контиги в один, если разрывы, то хотелось бы понимать, чему они соответствуют (интронам, межгенным промежуткам или каким-то странностям).
Попробуем разобраться на некоторых примерах.
Глава 3. "Разорвало Володьку". (OST День Выборов)
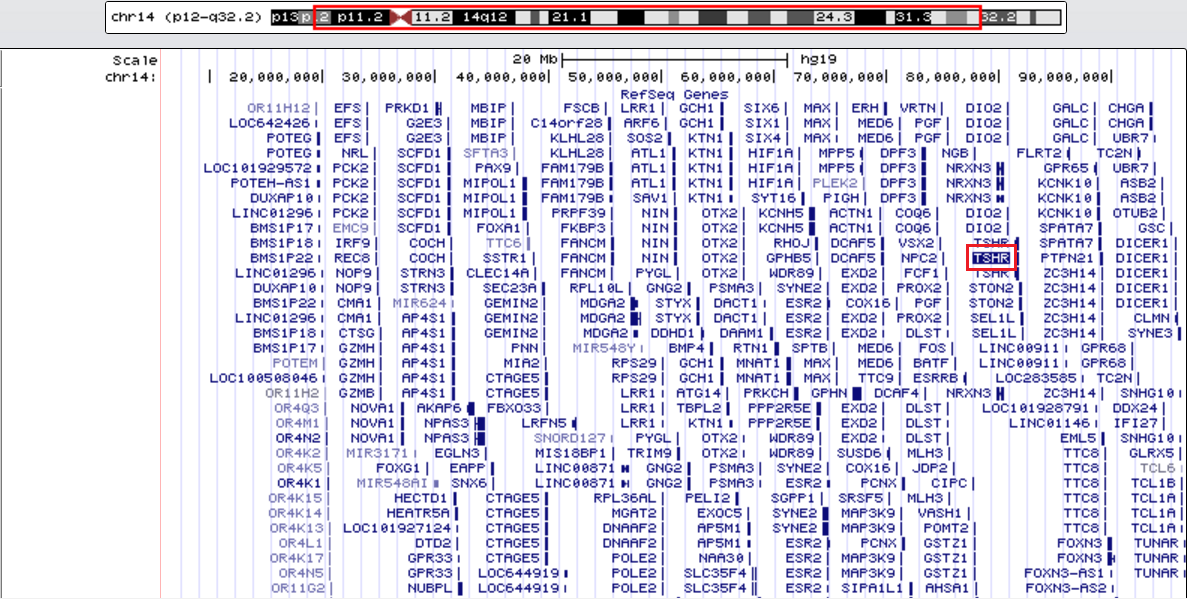
Если бегло просмотреть лист no-repeat, то можно увидеть, что контиги картировались в три области хромосомы (21025016-21028741; 81421789-81574687; 102228970-102394334), между которыми присутствуют два огромных разрыва в 60 и 20 млн п.н. соответственно ("стекинг"-области обозначены в таблице темно-красным цветом). Я решила посмотреть на хромосоме три эти области (Рис. 6а-с) и увидела, что каждая область целиком входит в один ген (ген РНКазы А, вышеупомянутый ген рецептора тироид-стимулирующего гормона и ген регуляторной субчастицы гамма фосфатазы 2, соответственно). Контиги, картировавшиеся много раз, как было выяснено выше, редко попадают в кодирующие области. Таким образом, наглядно видно, что экзом был отсеквенирован не целиком, а только экзоны из нескольких (возможно, целевых, то есть не случайных) генов.


А где же все гены?..
К примеру, на Рис. 6d показана область первого разрыва (21028741-81421789), в которой очень много генов, но, видимо, они не должны нас интересовать.
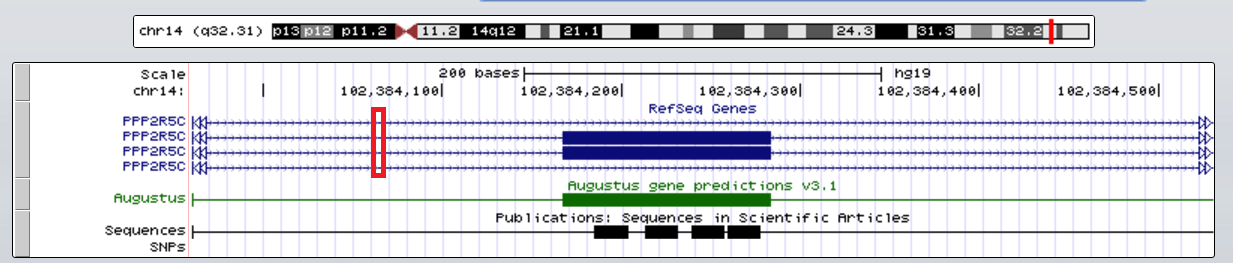
Также большой интерес представляет происхождение маленьких разрывов: например, между откартировавшимися на хромосому контигами 364 и 365 в таблице обнаружен разрыв в 2 нуклеотида.
Участок хромосомы, содержащий оба контига и разрыв между ними, попадает в интрон и последний экзон гена регуляторной субчастицы гамма фосфатазы 2А (Рис. 7а), что не дает нам никакой информации.
Еще не до конца отчаявшись, я решила запустить blastn (megablast) последовательности, состоящей из обоих контигов, по базе Human genomic + transcript.
На выравнивании находки из 14 хромосомы видны присутствует гэп длиной 2 нуклеотида, подтверждая наличие разрыва (Рис. 7b). Если почитать запись о данном участке хромосомы в GenBank, то можно подтвердить, что наш участок затрагивает интрон и экзон некоторых из транскриптных вариантов гена регуляторной субчастицы гамма фосфатазы 2А.
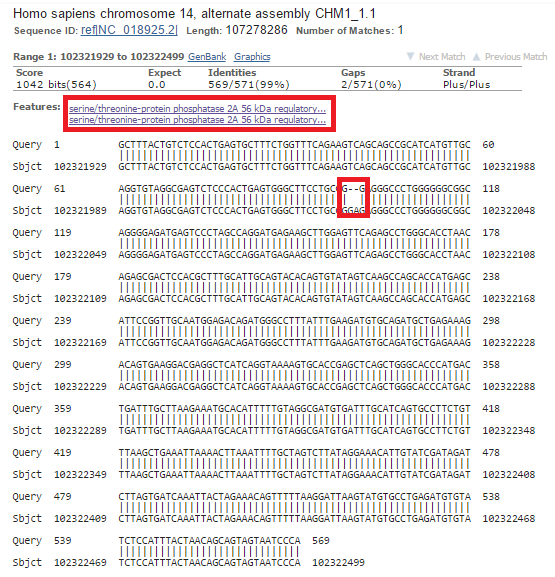
Несмотря на все старания, трудно сказать, откуда взялся этот разрыв, не сославшись на странности в работе программы.
Другая группа разрывов - от полусотни до нескольких тысяч пар нуклеотидов - могут с высокой вероятностью соответствовать интронам между экзонами. Но на деле все оказалось не так просто: я смотрела участки хромосомы, включающие пару контигов и разрыв между ними, ожидая увидеть экзоны и интрон между ними. Но мне, очевидно, не везло, так как я попадала в "чистые" интроны или межгенные промежутки, либо, в лучшем случае, захватывала экзон только с одной стороны. Так как я не смогла объяснить тот факт, что некоторые контиги вообще не картировались на хромосому, то на этом этапе я смотрела разрывы между соседними контигами. Лучший среди худших пример (разрыв между контигами 164 и 165) на Рис. 8.
Работа с разрывами, к счастью, до разрыва сердца меня не довела, но до разрыва мозга... Если с длинными разрывами гипотезу о принадлежности к интронам можно с оговорками признать правдоподобной, то, к сожалению, не удалось понять ничего определенного про происхождениемаленьких разрывов (здесь гипотезы простираются вплоть до неточностей работы программы).
Глава 4. "Скрещенья рук, скрещенья ног, судьбы скрещенья".
Возможно, перекрывания контигов принесут мне больше счастья.
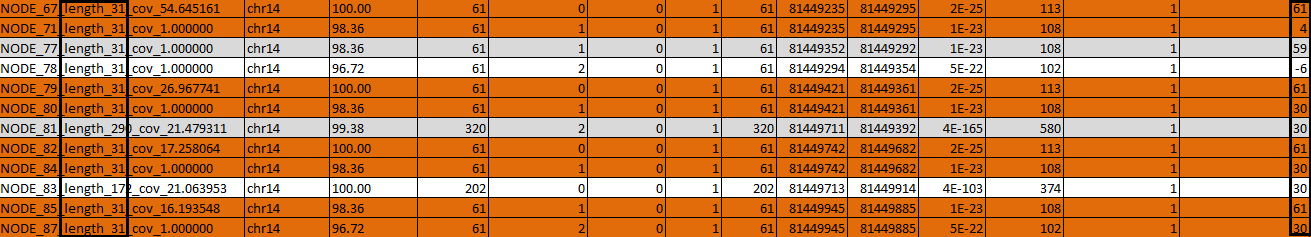
Например, рассмотрим перекрывание между 57 и 66 контигами - по данным таблицы, оно равно 10. На первый взгляд, удивительно, что между номерами контигов такой большой промежуток, но на Рис. 9а видно, что даже в файле contigs.faa отсутствуют контиги между указанными. Это очень странно, потому что получается, что общее число контигов меньше, чем 411, хотя в выдаче программы было указанно именно это число, что наводит на мысли о неполадках в работе программы.

Для выяснения отношений я вручную выровняла эти два контига: на Рис. 9b хорошо видно, что ровно 10 нуклеотидов образуют 100%-но консервативное перекрывание.

Внимание, вопрос: почему программа не объединила контиги с таким очевидным перекрыванием в один? По всей видимости, это слишком короткое перекрывание, и программе его недостаточно.
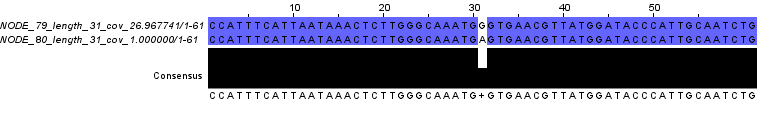
Еще один интересный факт, который хотелось бы отметить, это наличие огромного числа коротких контигов (61 нуклеотид), многие из которых перекрываются друг с другом по всей длине (Рис. 10а) (также часто встречаются перекрывания ровно на 30).
На Рис. 10b-c представлены выравнивания контигов 79-80 и 82-84, в каждом из которых присутствует всего по одному несоответствию. Но, видимо, программе достаточно этого несовпадения, чтобы не объединять участки в один контиг.
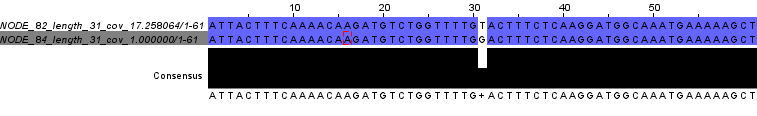
Хотя, на мой взгляд, с этими короткими контигами все нечисто.
Эпилог.
Жизнь как коробка шоколадных конфет, а сборка генома de novo как девайс из IKEA без инструкции по сборке: сложно, но невероятно увлекательно и совершенно необходимо.
Хочу выразить свою благодарность за крайне интересные задания, а также прошу не судить меня строго за выплески креативности, нацеленные на то, чтобы разнообразить довольно объемный отчет.