Реконструкция филогении по нуклеотидным последовательностям. Паралоги
Задание 1. Построение дерева по нуклеотидным последовательностям
В данном задании требовалось построить филогенетическое дерево 8 представителей протеобактерий (Таблица 1), используя нуклеотидную последовательность РНК малой субъединицы рибосомы (16S rRNA).
Последовательности 16S rRNA бактерий нужных видов (штамм указан в Таблице 1) были взяты из файлов с расширением .frn в записи, принадлежащей хромосоме каждой из бактерий, на сайте NCBI.
Таблица 1. Выбранные представители протеобактерий.
| Название | Мнемоника | Штамм |
| *Bradyrhizobium japonicum (Bradyrhizobium diazoefficiens) | *BRAJA (BRADU) | USDA_110_uid57599 |
| *Agrobacterium radiobacter (Agrobacterium tumifaciens) | *AGRRK | K84_uid58269 |
| Burkholderia cenocepacia | BURCA | MC0_3_uid58769 |
| Neisseria meningitidis | NEIMA | NZ_05_33_uid162077 |
| Salmonella typhimurium | SALTY | DT104_uid223287 |
| Yersinia pestis | YERPE | Nepal516_uid58609 |
| Yersinia pseudotuberculosis | YERPS | PB1__uid59153 |
| Pasteurella multocida | PASMU | Pm70_uid57627 |
* - в скобках указаны исходные виды; для выполнения данного задания для отмеченных позиций были взяты другие виды того же рода
Ссылка на fasta-файл с последовательностями всех требуемых 16S rRNA: 16S rRNA.
Затем полученные последовательности были открыты в JalView и выровнены программой Muscle.
Выравнивание в fasta-формате было импортировано в программу MEGA, где было реконструировано дерево методом Neighbor-Joining. Изображение дерева представлено на Рис. 1а (справа).
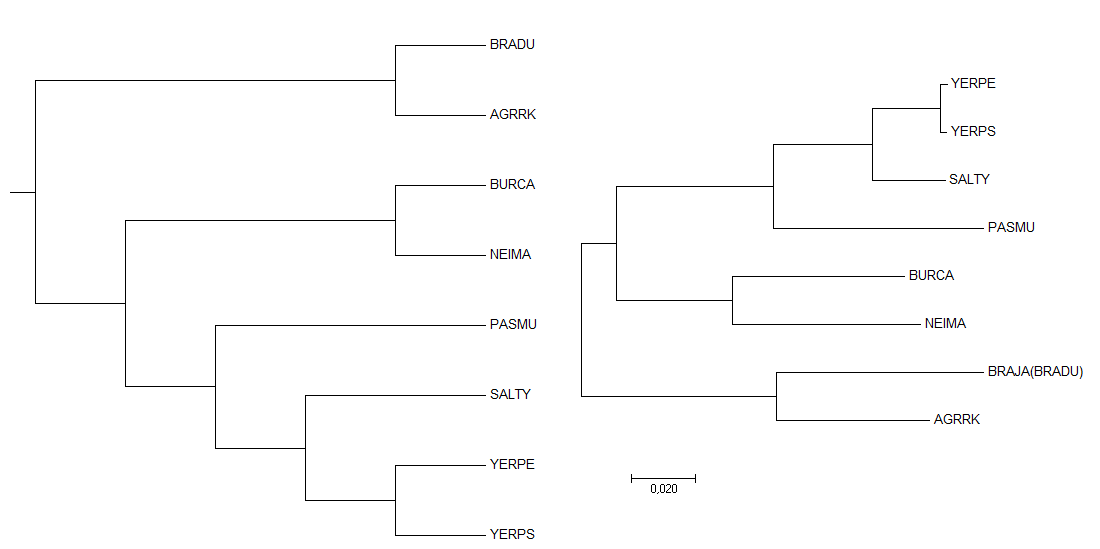
Как видно из Рис. 1а, дерево, построенное по последовательностям 16S rRNA, полностью совпадает с исходным, в отличие от деревьев, построенных по последовательности белка пептидил-тРНК гидролазы. В качестве напоминания на Рис. 1b представлено одно из деревьев, построенных по последовательности белка, реконструированное методом Neighbor-Joining и укорененное в среднюю точку. Оно отличается от правильного тем, что в нем листья BURCA и NEIMA не составляют отдельную кладу.
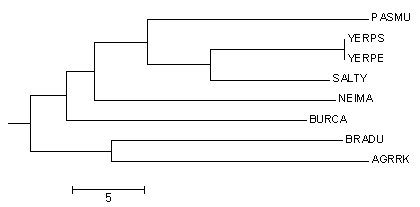
Таким образом, дерево, построенное по последовательностям РНК получилось правильным в отличие от дерева, построенного по последовательностям белка.
Задание 2. Построение и анализ дерева, содержащего паралоги
В данном задании было необходимо построить и проанализировать дерево гомологов белка CLPX_ECOLI из выбранных бактерий.
Чтобы найти гомологов, я взяла из файлы из директории P:\y14\term4\Proteomes, содержащие полные протеомы нужных мне бактерий, скачанные из базы UniProt, и командой "cat file1 >> file2" сложила их в один файл.
Затем я произвела поиск гомологов программой blastp с входной последовательностью белка CLPX_ECOLI по базе данных - файлу с нужными протеомами. Порог по E-value был взят 0.001.
Использованные команды:
- makeblastdb -in prot.fasta -dbtype prot -out db.fasta
- blastp -query clpx_ecoli.fasta -evalue 0.001 -db db.fasta -outfmt 6 -out res.fasta
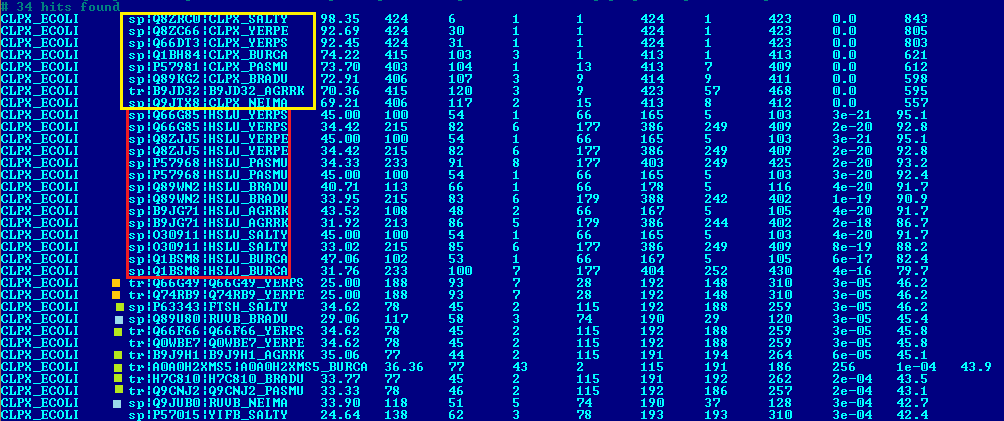
В выдаче (Рис. 2) всего 34 находки, из них 14 - по 2 находки, принадлежащие белку HSLU, на каждую из 7 бактерий (у бактерии NEIMA этого белка вообще нет). Остальные находки представлены в одном экземпляре для каждого белка.
Для удобства в Таблице 2 перечислены мнемоники белков, встретившихся в выдаче программы blastp.
Таблица 2. Мнемоники и графические выделения белков, встретившихся в выдаче blastp.
| Функция | Мнемоника | Графическое изображение на Рис. 2 |
| ATP-dependent Clp protease ATP-binding subunit ClpX | CLPX, B9JD32 | Желтая рамка |
| ATP-dependent protease ATPase subunit HslU | HSLU | Красная рамка |
| Putative magnesium chelatase family protein | Q66G49, Q74RB9 | Оранжевая точка слева |
| ATP-dependent zinc metalloprotease FtsH | FTSH, Q66F66, B9J9H1, A0A0H2XMS5, H7C810, Q9CNJ2 | Светло-зеленая точка слева |
| Holliday junction ATP-dependent DNA helicase RuvB | RUVB | Голубая точка слева |
| Cell division protein | Q0WBE7 | |
| Uncharacterized protein YifB | YIFB |
Затем последовательности были выровнены программой Muscle, по выравниванию было реконструировано дерево методом Neighbor-Joining в программе MEGA. Результат на Рис. 3.
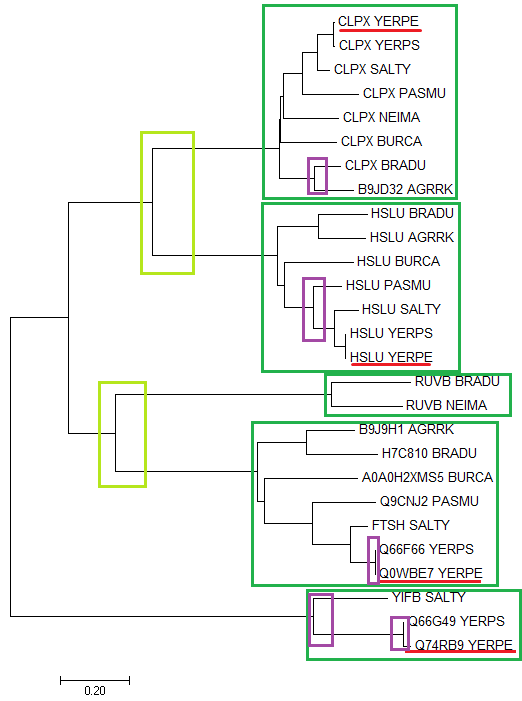
На полученном дереве указаны следующие эволюционные события:
- дупликация гена (салатовые рамки) - подтверждается тем, что два белка выполняют разные функции и просто являются разными, а присутствуют у всех/почти всех видов: белки CLPX и HSLU представляют собой разные субъединицы АТФ-зависимой протеазы и оба встречаются почти у всех видов (нет белка HSLU у вида NEIMA); о дупликации, приведшей к появлению белков RUVB (хеликаза) и FtsH (АТФ-зависимой цинк металлопротеазе - разные мнемоники), свидетельствует то, что у BRADU есть оба белка
- разделение путей эволюции белков в результате видообразования (фиолетовые рамки) - по сути выделяют ортологичные группы
Также на дереве указаны паралоги (красное подчеркивание) и ортологичные группы (темно-зеленые рамки).
- под паралогами понимаются два гомологичных белка из одного организма (выделены все гомологи из организма YERPE, попарно являющиеся друг другу паралогами)
- ортологами называем два гомологичных белка, если они: а) из разных организмов; б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования (рамками выделены группы, внутри которых белки попарно являются ортологами); интересно, что ортологичные группы по белкам ClpX (CLPX), HslU (HSLU) и FtsH (разные мнемоники) отображают внутри деревья, совпадающие с деревом на Рис. 1b (дерево, построенное по последовательности пептидил-тРНК гидролазы)