Поиск сигналов
Задание 3. Определение сайтов связывания данного транскрипционного фактора в данном участке хромосомы человека
В данном задании требовалось определить сайты связывания транскрипционного фактора в участке хромосомы человека. В моем случае работа выполнялась с файлом chipseq_chunk17.fastq, содержащим риды Illumina, полученные при проведении сhip-seq эксперимента.
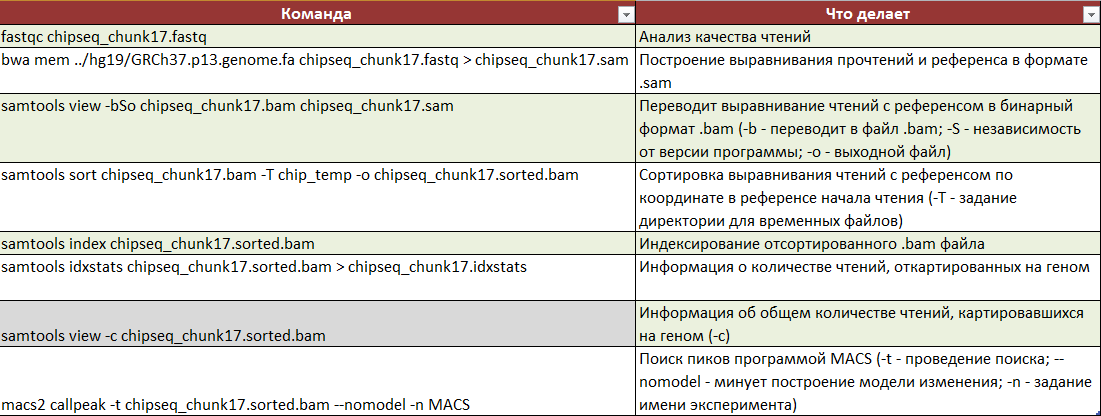
При выполнении заданий использовались команды, запускаемые через PuTTY. Список команд и описание того, что они делают, приведено в Таблице 1.
Подготовительный этап работы
Для начала было необходимо проверить качество ридов с помощью программы FastQC (команда fastqc).
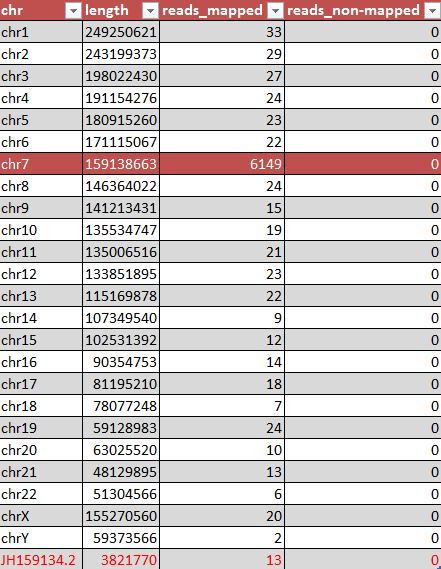
Результатом работы этой программы является архив chr14_fastqc.zip и файл в формате html, в котором нас, в частности, интересует картинка "Per base quality" (РИс. 1), на которой показано качество определения основания в каждой позиции рида. Видно, что качество ридов очень хорошее и вообще близко к максимальному - все значения только вверху зеленой области, что свидетельствует о хорошем качестве. Здесь же узнаем число ридов и их длину: 6605 и 36 нуклеотидов, соответственно.
Поэтому без скрипа в сердце было принято решение о том, что чистить риды программой Trimmomatic не нужно.
Затем потребовалось картировать на геном человека полученный участок, под чем подразумевается построение выравнивания прочтений и референсной последовательности (команда bwa mem). Результатом является файл в формате .sam, который мы не можем проанализировать напрямую.
Дальнейшие действия направлены на то, чтобы получить информацию о картировании нашего участка на человеческий геном:
- Сначала нужно перевести выравнивание чтений с референсом из формата .sam в бинарный формат .bam (команда samtools view). Результат: файл .bam.
- Затем было нужно отсортировать выравнивание чтений с референсом по координате начала чтения в референсе (команда samtools sort). Результат: файл .sorted.bam.
- Теперь проиндексируем отсортированный файл .bam (команда samtools index).
- Наконец получим информацию о количестве чтений и местах их картирования на геном (команда samtools idxstats). Результат: файл .idxstats.
- Также можно получить информацию об общем количестве откартировавшихся ридов (команда samtools view с опцией -с). Результат: 6605 (то есть все).
Итак, внимание, результаты картирования:
- Риды картировались на 49 участков, среди которых все хромосомы, кроме митохондриального генома (Лист 1 файла Excel - все ненулевые картирования), а природу остальных участков выяснить не удалось.
- На Рис. 1 представлены результаты картирования для всех хромосом и для одного из неопознанных участков, на который картировалось больше 3 ридов (выделен в таблице на Рис. 1 текстом красного цвета).
- Больше всего ридов (6149) картировалось на хромосому 7 - ее участок и был исходно дан (строка выделена в таблице на Рис. 1 бордовым цветом).
- На геном картировались все риды (полная выдача на Листе 2 файла Excel).
- Картирование ридов на другие хромосомы и участки, вероятно, свидетельствует о вхождении повторов в участки и/или о присутствии сайтов связывания некоторых факторов транскрипции в нескольких хромосомах.
Поиск пиков (PEAK CALLING) с помощью программы MACS
Сначала я запустила команду macs2 callpeak так:
macs2 callpeak -t chipseq_chunkX.sorted.bam
. Но программа выругалась, так как пиков было слишком мало, поэтому запуск был произведен так:macs2 callpeak -t chipseq_chunkX.sorted.bam --nomodel -n MACS
.* oпция -n (--name) позволяет задать имя эксперимента
Результатом работы программы являются три файла:
Комментарий найденного:
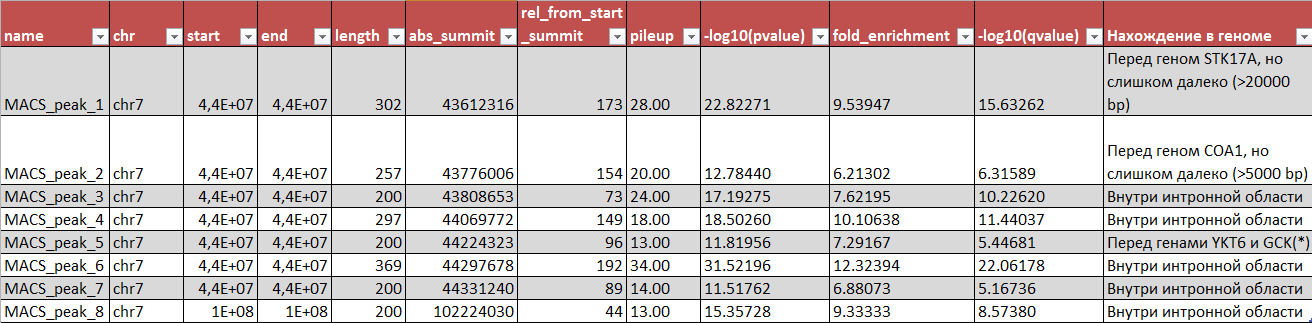
Обнаружено 8 пиков, причем 7 расположены приблизительно в одной локации (43-44 млн п.н.), а последний расположен далеко (102 млн п. н.). Ширина пиков от 200 (самое распространенное значение) до 369; в целом, у более широких пиков лучше (меньше) p-value и больше значения fold enrichment (кратность превышения случайного распределения Пуассона). Более подробная информация о ширине и координатах вершин пиков (указаны как абсолютные координаты, так и местоположение относительно начала пика) на Рис. 2. На том же рисунке также содержится информация о количественных характеристиках, связанных с достоверностью пиков (подробнее ниже), и краткая информация о местоположении пиков относительно функциональных элементов генома (подробнее см. далее).
Визуализация результатов
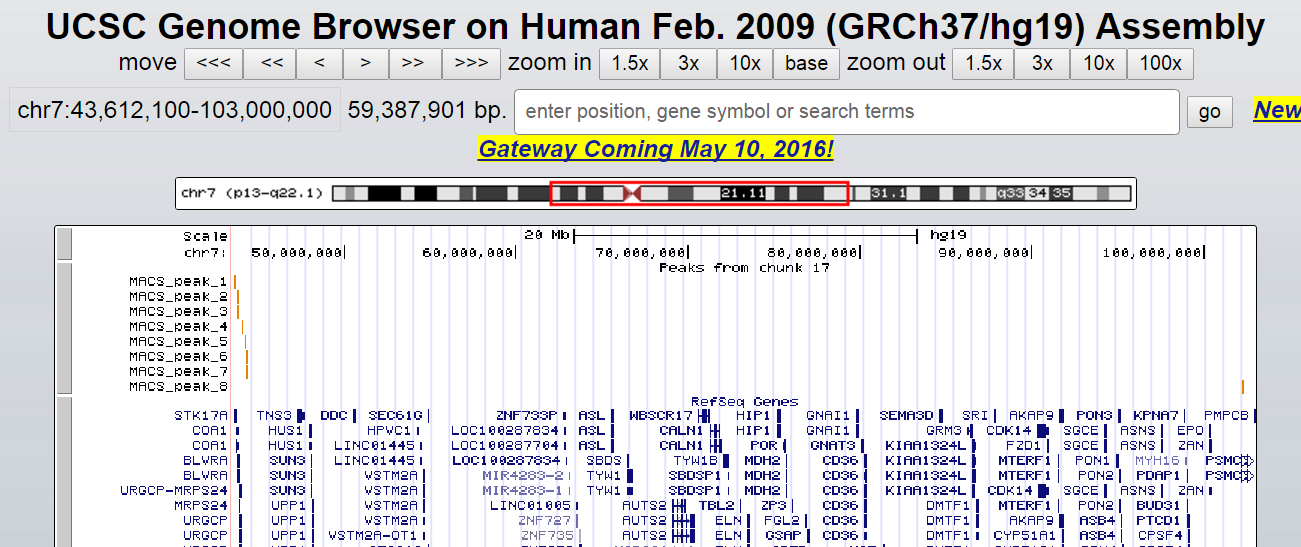
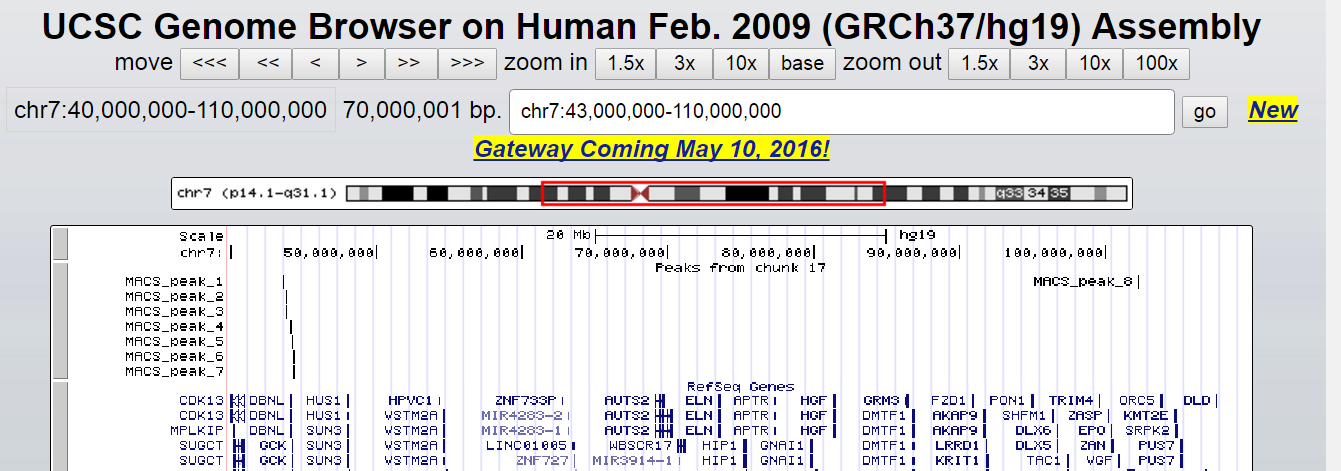
Результаты были визуализированы с помощью UCSC Genome Browser. Для этого в файл MACS_peaks.narrowPeak была дописана строчка "track type=narrowPeak visibility=3 db=hg19 name="my_peaks" description="Peaks from chunk 17" browser position chr7:43612100-102300000". На Рис. 3a представлен скриншот окна геномного браузера, где в мелком масштабе отображены все найденные пики (загружено из файла MACS_peaks.narrowPeak); на Рис. 3b отображены только вершины пиков (загружен файл MACS_summits.bed).
Достоверность пиков
Как известно из статистики, величина p-value характеризует достоверность эксперимента: если его значение ниже порогового, что результат можно признавать статистически значимым. q-value, которое по-другому также называется "adjusted p-value", является той же самой характеристикой, но для множественных экспериментов (высчитывается по распределению значений многих p-value, то есть это оценка достоверности с поправкой на множество экспериментов). Аналогично, чем меньше q-value, тем достовернее результат (более подробно можно прочитать здесь).
Эти выводы подтверждаются информацией, которую можно найти в геномном браузере. Так, можно добавить в окно поле с информацией о сайтах связывания факторов транскрипции из ранее проведенных ChIP-seq экспериментов. На Рис. 4a-b показаны в крупном масштабе пики 1 и 6, соответственно, с хорошими значениями p-value и q-value (см. Рис. 2 : лучше => меньше, но в таблице -log 10 от этих величин, поэтому там больше => достовернее), которые подтверждены результатами других экспериментов, то есть уже ранее кем-то были обнаружены. Обратная ситуация на Рис. 4с: там изображен пик 3, у которого показатели p-value и q-value гораздо хуже (см. Рис. 2) и который не подтвержден в других исследованиях.
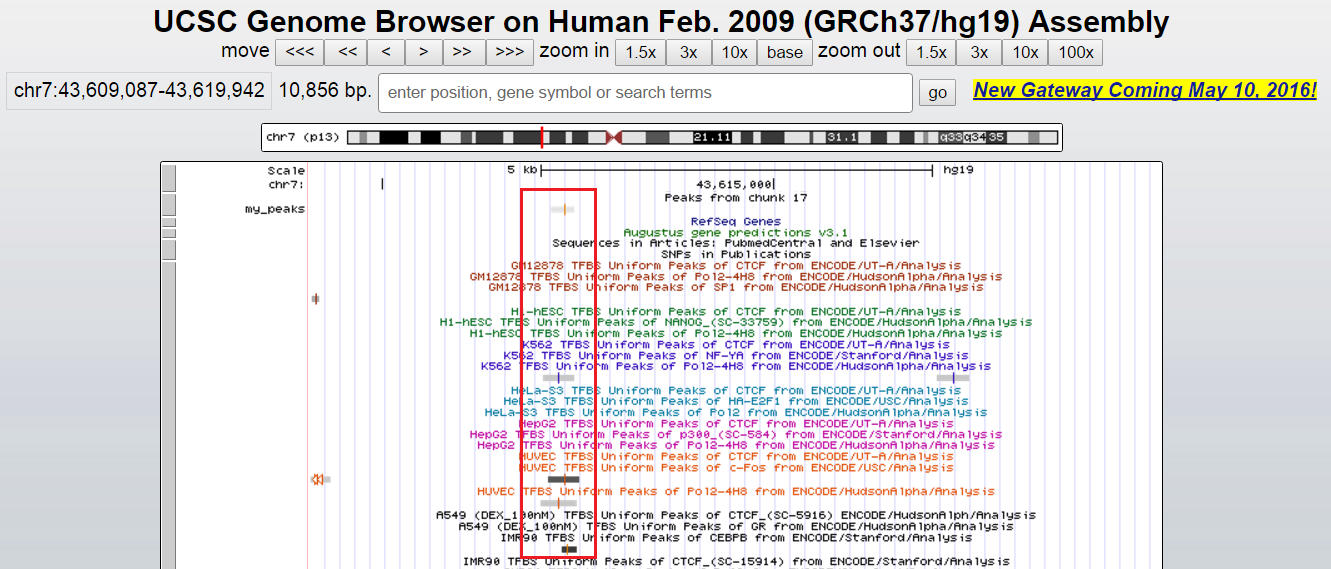


Расположение относительно функциональных элементов генома
Рассматривая геном человека, трудно определить по сайту связывания фактора транскрипции, какой ген этим фактором регулируется, так как сайт может находиться на разных расстояниях относительно гена (сайт может быть сильно удален по последовательности, но пространственно приближен в определенных условиях ввиду укладки хроматина; также часто транскрипция генов регулируется комплексом факторов транскрипции, следовательно, некоторые из них не обязательно должны быть приближены к гену). К сожалению, без помощи специфичной для конкретного гена информации вывод о такой сложной регуляции сделать невозможно, поэтому в геномном браузере можно было только посмотреть, не находится ли сайт транскрипции около какого-то гена.
Увы, пики 3, 4 и 6-8 расположены внутри интронной области генов, а другие гены расположены от них далеко, поэтому в этих случаях ничего сказать нельзя.
Пики 1 и 2 находятся "вблизи" некоторых генов, но это расстояние превышает 20000 и 5000, соответственно, что, на мой взгляд, слишком много. К тому же, в других экспериментах найдены другие сайты (выделены красными рамками на Рис. 5а-b), которые расположены гораздо ближе к этим генам и с гораздо большей вероятностью являются сайтами связывания факторов транскрипции этих генов.
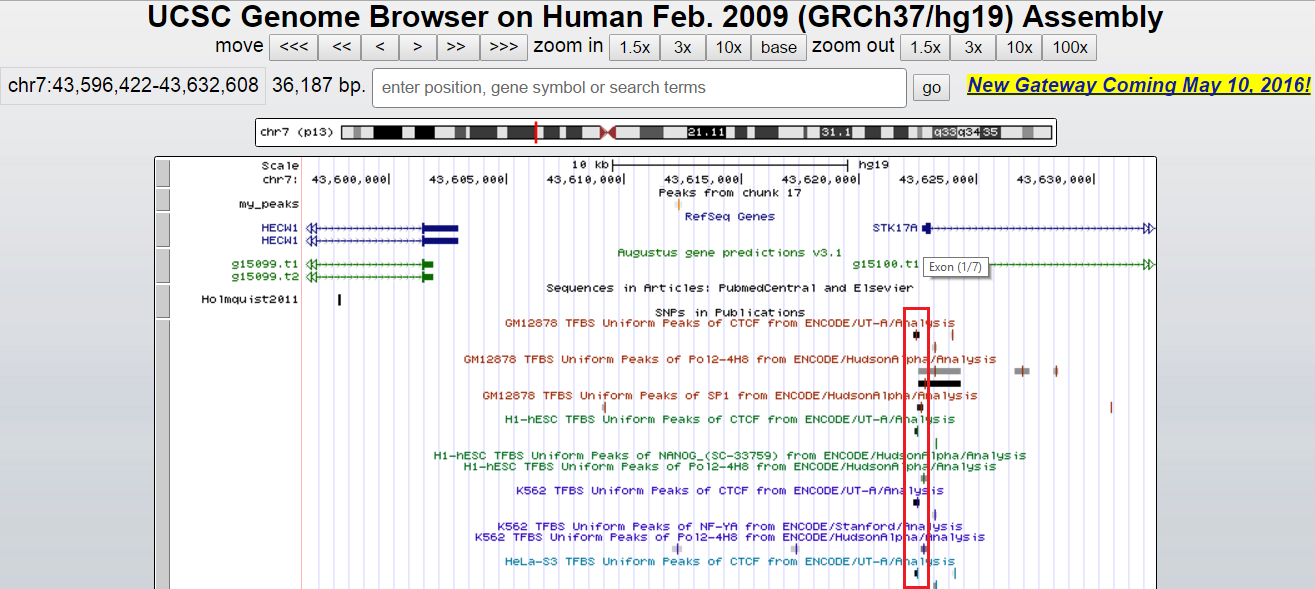
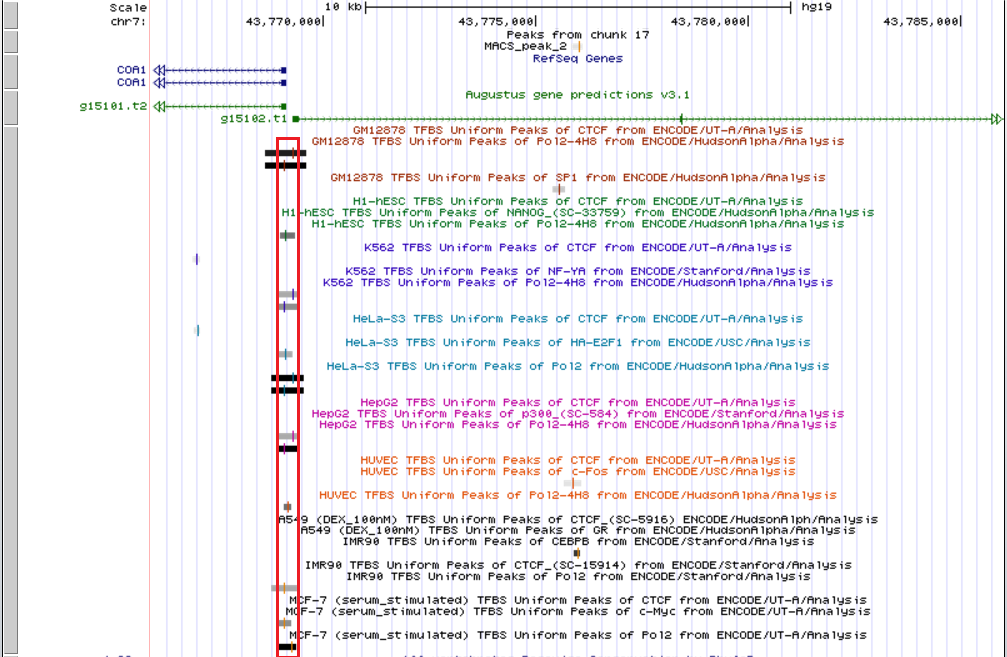
Гораздо более интересная ситуация с пиком 5 (Рис. 5с), так как он "приближен" к началам гена YKT6 и двух из трех изоформ (вариантов сплайсинга) гена GCK.
YKT6 - ген SNARE-узнающего ассоциированного с мембраной (изопренилированного) белка, участвующего в везикулярном транспорте между секреторными органеллами на этапе транспортировки из ЭПР через аппарат Гольджи.
GCK - ген глюкокиназы - фермента, фосфорилирующего глюкозу, что является первым этапом большинства путей метаболизма глюкозы, причем это вариант сплайсинга, экспрессирующийся в печени.
Однако наименьшее расстояние - до гена YKT6 - более 15000 п. н., поэтому я также не могу утверждать, что это сайт связывания транскрипционного фактора.

Таблица 1. Использованные команды.
Задание 2.
В данном задании требовалось проанализировать результаты ChIP-seq эксперимента по поиску ТАТА-box - сайтов связывания белка TBP. Это связывание необходимо и достаточно для инициации транскрипции у эукариот. ТАТА-бокс имеет консенсус T-A-T-A-A/T-A/G.
Для анализа был выбран эксперимент на клеточной линии H1-hESC c помощью антител кролика (Рис. 1).

TBP является субъединицей комплекса TFIID. TBP узнаетTATA-box и помогает направить инициаторный комплекс для РНК-полимераз II III.
Рассмотрим несколько примеров сайтов связывания TBP.
Гены, транскрипция которых инициируется ТАТА-бокс связывающим белком (TBP).
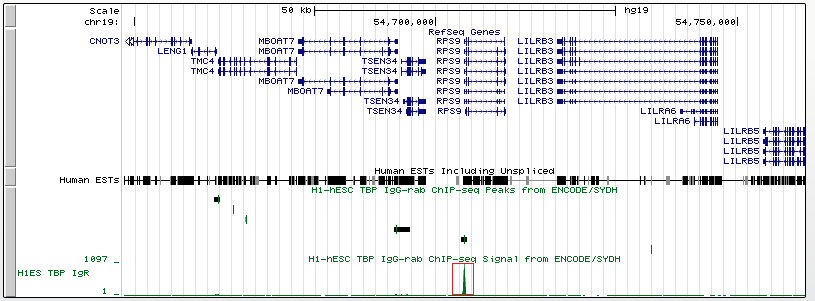
Ген RPS9
Ген RPS9 (вариант транскрипции 6) - ген рибосомального белка S9, локализованного в малой субъединице. Принадлежит семейству рибосомальных белков S4P. Также в комментариях к этому гену на сайте геномного браузера сказано, что для этого гена, как и для других генов рибосомальных белков, характерно большое количество псевдогенов, распространенных по всему геному.
Координата старта транскрипции (chr19:54704726, прямая цепь).
Длина гена: 6790 п.н.
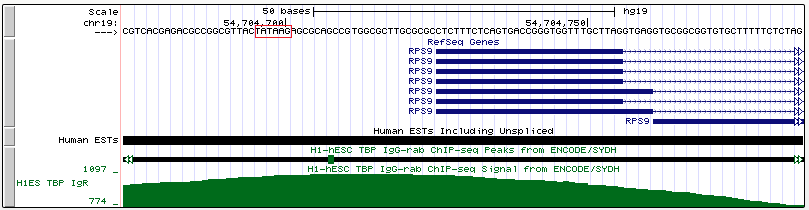
Видно, что ТАТА-бокс очень консервативный, и пик хороший. Вероятно, это связано с тем, что рибосомные белки очень важны для биосинтеза белка (известно, что эти белки самые экспрессируемые в клетке).
Ген FOSB
Ген FOSB (вариант транскрипции 1) - ген прото-онкогена, субъединицы транскрипционного факторв AP-1. Принадлежит Fos-семейству генов, к которому также относятся гены FOS, FOSL1, FOSL2. Эти гены кодируют лейциновые молнии, которые димеризуются вместе с белками семейства JUN, таким образом формируя комплекс транскрипционного фактора АР-1. Вследствие этого, данные белки рассматриваются как регуляторы пролиферации и дифференциации клеток.
Координата старта транскрипции (chr19:45971253, прямая цепь).
Длина гена: 7185 п.н.
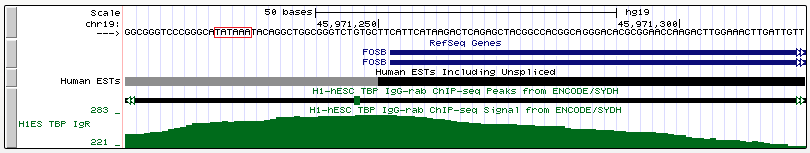

Данный случай очень интересный. С одной стороны, опять же ТАТА-бокс очень консервативный, что объясняется важностью транскрипционных факторов в жизни клеток. Однако здесь сам пик очень низкий (на Рис. 3b видно, что этот пик гораздо ниже соседнего).
Ген KLF2
Ген KLF2 - Kruppel-like factor - представителя семейства широкоэкспрессируемых транскрипционных факторов, содержащих цинковые пальцы. KLF2 регулирует траффик Т-клеток, способствуя экспрессии липид-связывающего рецептора S1PR1 и селектина CD62L.
Координата старта транскрипции (chr19:16435637, прямая цепь).
Длина гена: 3837 п.н.
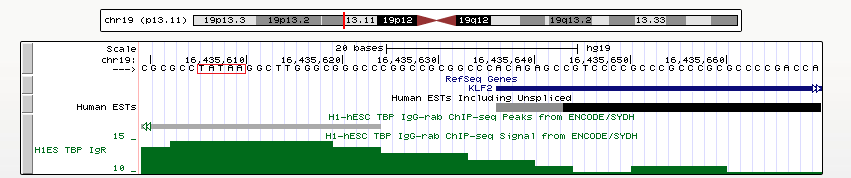
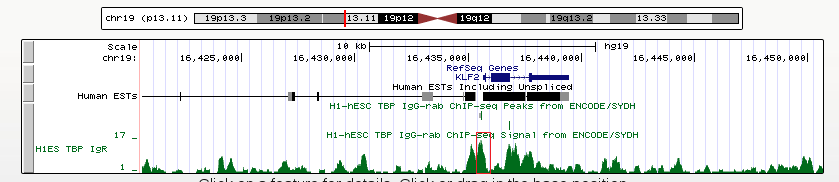
Снова хороший ТАТА-бокс, так как снова ген транскрипционного фактора. Но на этот раз пик лишь не намного превышает уровень шума.
Ген, не имеющий сигнала в промоторной области
Ген LAIR2
Ген LAIR2 (вариант транскрипции 2) - ген ассоциированного с лейкоцитами похожего на иммуноглобулин рецептора 2, представителя семейства иммуноглобулинов. Данный ген участвует в коллаген-индуцированной агрегации тромбоцитов и в формировании сосудов при закупоривании тромбоцитами.
Координата старта транскрипции (chr19:55014013, прямая цепь).
Длина гена: 7888 п.н.
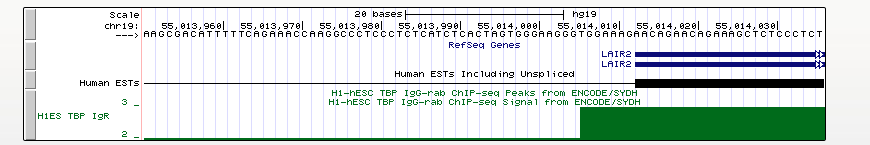
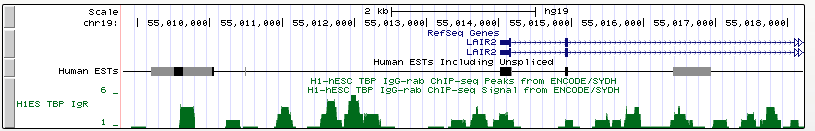
В этом случае пики в крестности старта транскрипции ниже уровня шума, и при близком рассмотрении ТАТА-бокс не находится.
Увлекательные цинковые пальцы на примере генов ZNF610 и ZNF880
Сначала посмотрим на данные пики при малом масштабе (Рис. 6а). Видно, что пики очень хорошие (одни из самых лучших во всем эксперименте, и это характерно почти для всех генов цинковых пальцев).
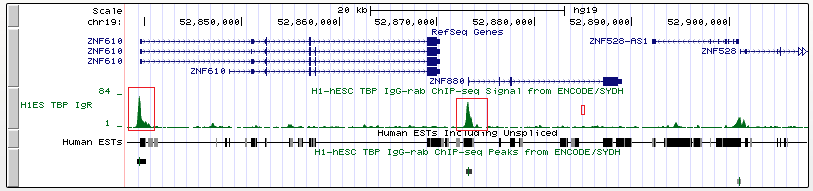
Теперь посмотрим на пики по отдельности и поближе.
Ген ZNF610
Координата старта транскрипции (chr19:52873170, прямая цепь).
Длина гена: 15876 п.н.
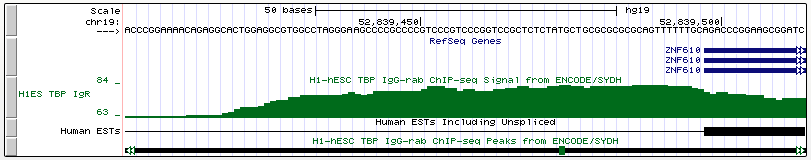
Несмотря на очень хороший пик, ТАТА-бокс не найден.
Ген ZNF880
Координата старта транскрипции (chr19:52839498, прямая цепь).
Длина гена: 30878 п.н.

Аналогично, ТАТА-бокса нет.
Следует отметить, что все эти примеры было найти достаточно трудно, и в целом, результаты работы подтверждают тот факт, что транскрипция только около 25% генов инициируется TBP, и только перед 10% генов имеется явно выраженный ТАТА-бокс.