|
Метаболические пути. KEGG
Задача — определить, существует ли
фрагмент метаболических путей от аспартата к аргинину(
информацию можно посмотреть в таблице
)
1. Поиск графических формул и идентификаторов заданных низкомолекулярных веществ в KEGG
быстрая ссылка: http://www.genome.jp
В этом задании пойск проводился в разделе Ligand
по ключевым словам aspartate (аспартат) и
arginine (аргинин)
| |
aspartate (аспартат) |
arginine (аргинин) |
| идентификатор
в базе данных KEGG |
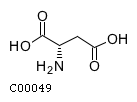
C00049
|
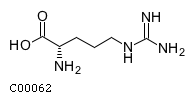
C00062 |
| английские
названия |
L-Aspartate
L-Aspartic acid
2-Aminosuccinic acid
|
L-Arginine
(S)-2-Amino-5-guanidinovaleric acid |
| русские
названия |
L-аспартат
L-аспарагиновая кислота
2-аминосуциновая кислота (термин в
настоящее время не употребляется в
русскоязычном варианте) |
L-аргинин
(S)-2-амино-5-гуанидиновалеровая кислота |
| изображение |
 |
 |
| формула |
C4H7NO4 |
C6H14N4O2 |
| молекулярная
масса |
133.0375 |
174.1117 |
| другое |
то же самое, что и
D00013 |
то же самое, что и
D02982 |
2. Поиск метаболического пути. Выделение цветом пути превращения заданных веществ
На главной страничке KEGG есть
ссылка на "Table of
contents", собственно, она и была
использована. Далее была использована
специальная программа
для раскрашивания объектов на
метаболических картах. Сначала в поле
запроса было введено:
c00049 red
c00062 red
это найденные идентификаторы с указанием
раскрашивания красным цветом). И клик по
кнопке подтверждения привел нас на
страничку с результатами поиска. В случае
данного исследования результатов 17. (среди
которых были замечены
карта биосинтеза лизина и бактериального
хемотаксиса, что наводит на мысль о том,
что задание об исследовании
метаболических путей может быть связано с
исследуемым белком (например одной из
функций белка, который я исследую (DAPB_ECOLI)
является синтез лизина, однако, это может
быть простое совпадение) Однако
я рассматривал цикл мочевины и метаболизм
аминогрупп, так как там найдены оба
вещества и процесс метаболизма, на мой
взгляд, наиболее нагляден. Aspartate -
L-Arginino-succinate - Arginine. Выбранные ферменты были
выделены желтым, для чего был создан
запрос:
c00049 red
c00062 red
6.3.4.5 yellow
4.3.2.1 yellow
Сразу заметим, что, помимо
выбранной ранее карты, были найдены и
другие (карта
метаболизма аргинина и пролина и карта
метаболизма аланина и аспартата),
которые тоже содержат указанные в запросе
ферменты, таким образом, можно, исходя от
противного, (зная коды ферментов) узнавать
об их функциях, или, скажем, находить
вещество, которое получается из исходного
под действием какого-то количества
заранее известных ферментов.
Это может быть очень полезно при
классификации, например, липидов, для
которых существует довольно большое
разнообразие разных классификаций, но нет
абсолютной и единой.
Эта картинка,
собственно, отражает ход метаболизма. (картинка
обработанная и урезанная, ее полностью
можно увидеть тут)
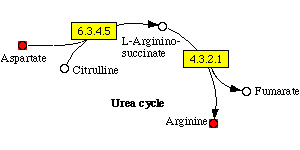
3. Исследование выбранного метаболического пути
В конкретном
метаболическом пути всего 2 этапа. (карта
была переведена в режим Reference pathway (Reaction))
Возможный путь превращения
аспартата в аргинин.
| Этап |
Ферментативная реакция |
Фермент |
| 1 |
ATP + L-Citrulline + L-Aspartate <=> AMP + Pyrophosphate +
N-(L-Arginino)succinate |
EC 6.3.4.5 |
| 2 |
N-(L-Arginino)succinate <=> Fumarate + L-Arginine |
EC 4.3.2.1 |
Интересно то, что
аспартат приходит цикл извне, тогда как
аргинин, судя по карте из него не выходит
никогда :)
Таблица, отражающая возможность выбранной цепочки реакций у конкретных организмов по данным
KEGG:
|
Организм
|
Возможно ли
превращение
аспартата в аргинин
|
Краткое обоснование
|
| Homo
sapiens |
ДА |
Известны гены всех ферментов, катализирующих все стадии. |
| Escherichia
coli |
ДА |
Известны гены всех ферментов, катализирующих все
стадии (проверялись только Escherichia
coli K12) |
| Pseudomonas
aeruginosa PAO1 |
ДА |
Известны гены всех ферментов, катализирующих все
стадии |
| Methanococcus
janaschii |
НЕТ |
Гены ни одного из ферментов, катализирующих стадии этой цепочки реакций
неизвестны. |
По всей видимости, наименее
изученным организмом является
Methanococcus
janaschii
©
Бирюков
|


